Variações genômicas de organelas sem precedentes primordiais revelam cenários evolutivos independentes de plantas parasitas e a diversificação de complexos mitocondriais vegetais
BMC Biology volume 20 , Número do artigo: 49 ( 2022 )
Resumo
Fundo
As glórias da manhã (Convolvulaceae) são distribuídas em todo o mundo e produzem culturas economicamente importantes, ervas medicinais e ornamentais. Os membros desta família são diversos em características morfológicas e modos tróficos, incluindo o parasita sem folhas Cuscuta (dodders). Os genomas de organelas eram geralmente usados para estudar a filogenia de plantas e variações genômicas. Notavelmente, plastomas em plantas parasitas sempre apresentam características não canônicas, como tamanho reduzido e taxas aceleradas. No entanto, poucos genomas de organelas desse grupo foram sequenciados, dificultando nossa compreensão de sua evolução, e do mitogenoma dodder em particular.
Resultados
Reunimos 22 novos mitogenomas e 12 novos plastomas em Convolvulaceae. Juntamente com os já conhecidos, analisamos totalmente os genomas de organelas de 23 espécies da família. Nossa amostragem inclui 16 espécies autotróficas folhosas e 7 robalos parasitas sem folhas, abrangendo 8 das 12 tribos. Ambos os genomas plastidiais e mitocondriais dessas plantas encontraram variações que raramente foram observadas em outras angiospermas. Todos os plastomas possuíam limites IR atípicos. Além das perdas de genes e IR em dodders, algumas espécies folhosas também apresentaram perdas de genes e íntrons, duplicações, variações estruturais e inserções de DNAs estranhos. A filogenia reconstruída por sequências codificadoras de proteínas plastidiais confirmou a relação anterior das tribos. No entanto, a monofilia de 'Merremieae' e do grupo irmão de Cuscuta permaneceu incerta. O mitogenoma foi significativamente inflado em Cuscuta japonica , que ultrapassou mais de 800 kb e integrou DNAs maciços de outras espécies. Em outros dodders, os mitogenomas foram mantidos em tamanho reduzido, revelando estratégias evolutivas divergentes. Mutações exclusivas de plantas foram detectadas no gene mitocondrial ccmFc , que se quebrou em três fragmentos por meio de fissão gênica e mudança de splicing. As mudanças incomuns provavelmente aconteceram inicialmente com o ancestral comum da família e foram causadas por uma inserção estrangeira de rosídeos seguida de quebras de fita dupla e reparos imprecisos de DNA. As regiões codificadoras de ccmFc se expandiram em ambos os lados após a fissão, o que pode ter alterado a estrutura da proteína.
Conclusions
Our family-scale analyses uncovered unusual scenarios for both organelle genomes in Convolvulaceae, especially in parasitic plants. The data provided valuable genetic resources for studying the evolution of Convolvulaceae and plant parasitism.
Fundo
Convolvulaceae Juss., também conhecida como glória-da-manhã e trepadeiras, é uma grande família pertencente à ordem Solanales das eudicotiledôneas. Contém aproximadamente 1900 espécies de 59 gêneros [ 1 ]. Os membros da família incluem culturas (por exemplo , batata-doce), vegetais (por exemplo, espinafre de água), plantas medicinais (por exemplo, sementes secas de dodders e videiras de Erycibe ), ornamentais (por exemplo, glória da manhã, cipreste e flor da lua) , e ervas daninhas resistentes (por exemplo, dodders). Convolvulaceae é cosmopolita e exibe uma rica diversidade de características morfológicas, incluindo ervas, arbustos, árvores, trepadeiras e plantas parasitas sem folhas Cuscuta (dodders).
Nas últimas décadas, as espécies de Convolvulaceae foram classificadas em 12 tribos e seis clados usando vários marcadores de DNA [ 2 , 3 , 4 , 5 ]. Com grandes avanços nas tecnologias de sequenciamento de genomas, genomas de plastídeos (plastomas) de muitos táxons de plantas foram concluídos. No entanto, os estudos de plastoma de Convolvulaceae se concentraram principalmente em Ipomoea [ 6 , 7 , 8 ] e dodders [ 9 , 10 , 11 , 12 ]. Muitos gêneros desta família foram negligenciados, deixando a relação filogenética não testada com conjuntos de dados maiores e as posições de 'Merremieae' e Cuscuteae não resolvidas [ 4 , 13 ]. Além disso, apenas dois genomas mitocondriais (mitogenomas) nesta família eram conhecidos, um é Evolvulus alsinoides [ 14 ] e o outro é Ipomoea nil , que foi obtido a partir de todo o projeto de sequenciamento do genoma [ 15 ].
Por outro lado, genomas de organelas em Convolvulaceae exibiram múltiplas características peculiares. Por exemplo, a deleção do intron de rpl2 em plastomas de Convolvulaceae foi uma sinapomorfia e um evento único em Asteridae [ 3 ]. O infA foi perdido do plastoma e os limites de repetição invertida (IR) foram alterados em Ipomoea e dodders [ 8 , 12 ]. O mitogenoma de I. nil tem apenas 266 kb de comprimento, que é muito menor do que a maioria dos mitogenomas conhecidos em angiospermas. Além disso, a anotação do mitogenoma (GenBank ID: AP017303) revela um ccmFc , com fragmentos localizados em três posições diferentes. Mais estudos são necessários para conciliar esses achados. Plastomas de dodders, como muitas outras plantas heterotróficas (como [ 16 , 17 ]), exibiram tamanho menor, genes reduzidos, estrutura variável e substituições de nucleotídeos elevadas [ 10 , 11 , 18 ]. Os genomas nucleares do dodder também sofreram perdas maciças de genes [ 19 , 20 ]. Em contraste, o padrão evolutivo de seus mitogenomas permanece inexplorado.
Para desvendar os segredos dos genomas de organelas de Convolvulaceae, montamos 22 mitogenomas e 16 plastomas. Nossas coleções abrangeram cinco dos seis clados e oito das 12 tribos, representando uma das amostras mais abrangentes desse grupo. Revelamos as variações incomuns do genoma das organelas e descrevemos suas trajetórias evolutivas. Os mitogenomas em dodders parasitas evoluíram em diferentes direções, adquirindo grandes quantidades de DNA estranho ou retendo um tamanho pequeno. Mutações incomuns foram detectadas no ccmFc em todos os membros da família, e dados ômicos foram empregados para confirmar a fissão gênica, mudança de splicing e expansão de codificação.
Results and discussion
Plastome variations in Convolvulaceae
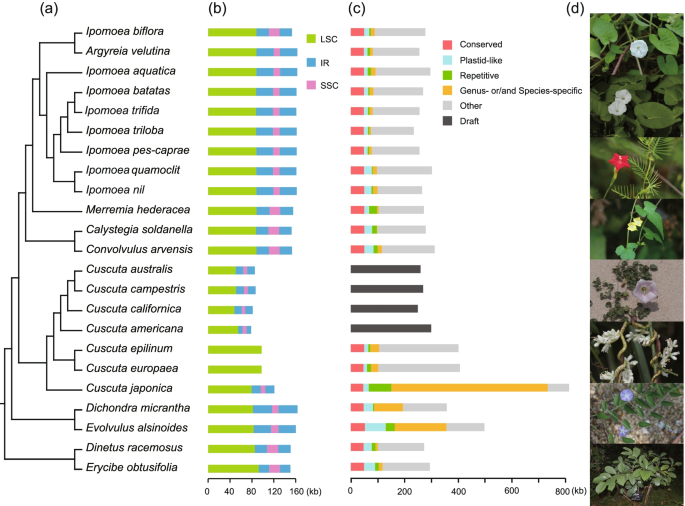
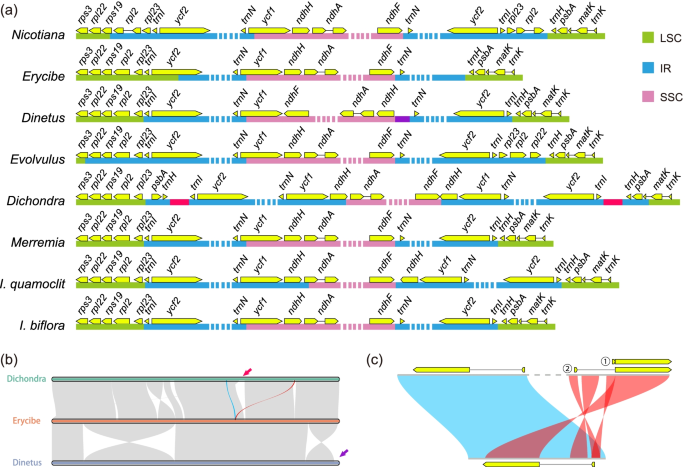
Our assemblies include at least 12 new plastomes, among which six genera were sequenced for the first time (Additional file 1). We also assembled four dodders for the first time, two from the subgenus Cuscuta (Cu. epilinum and Cu. europaea), and two from the subgenus Grammica (Cu. americana and Cu. californica). These new plastomes showed similar features to the known ones in Cuscuta, including the losses of genes, introns, and IR regions (Fig. 1b; Additional file 1) [12, 21]. In the leafy species, the plastid DNA sequences or the gene content did not differ much, and the main distinction came from the variation of the IR regions (Figs. 1b and 2a; Additional file 1). IR regions expanded or shrank in different directions and resulted in different architectures—all the species in this family were atypical in IR boundaries (Fig. 2a). Dinetus and Dichondra contained some inversions, and they were the only two leafy species that lost the gene rpl23 (Fig. 2b; Additional file 1). The Dichondra plastome also had some unusual duplications, which could create another copy of rpl16 (Fig. 2b, c, blue and red ribbon represented the original and second copy of rpl16, respectively). Rps16 and rpoC1 in Evolvulus, and rps16 and ycf3 in Dichondra have experienced intron losses. InfA was lost in Convolvulaceae, and BLASTP searches in Convolvulaceae nuclear peptides have confirmed the transfer to nucleus, like many other species [22].
Amostragem, conteúdo de plastoma e mitogenoma e diversidade fenotípica de Convolvulaceae. a Amostras deste estudo. A sua relação foi referida à árvore CDS plastidial na Fig. 5 . b Tamanho e estrutura do plastoma. Região de cópia única longa de LSC, região de repetição invertida de IR, região de cópia única curta de SSC. c Tamanho e proporções do mitogenoma. Os comprimentos totais das barras representaram o comprimento de cada mitogenoma, com cores diferentes indicando a proporção de cada tipo de sequência. As quatro espécies de Cuscuta subgênero Grammica tiveram apenas tamanhos de calados determinados sem as proporções quantificadas. d Morfologia de I. biflora , I. aquatica , I. quamoclit , Merremia hederacea , Calystegia soldanella , Cu. japonica , Evolvulus alsinoides e Erycibe obtusifolia (de cima para baixo)
Variações de plasmoma em Convolvulaceae. um limite IR muda. Calystegia e Convolvulus e outras Ipomoea spp. não foram mostrados, uma vez que são semelhantes a I. quamoclit em estrutura. Os segmentos roxo e rosa localizados em Dinetus e Dichondra indicam inserções estrangeiras. b Plastomes (último IR removido) de Dichondra e Dinetus sofreram inversões. As setas rosa e roxa mostraram a posição das inserções estranhas, que não possuíam homólogos com outros plastomas. A fita vermelha exibiu a posição de ( c ). c Repetições dispersas em Dichondra podem criar outra cópia de rpl16 . O primeiro exão foi duplicado duas vezes. Portanto, a segunda cópia do rpl16 pode ter duas combinações potenciais (marcadas com os números 1 e 2)
Dinetus and Dichondra had a ca. 1.5 and 2.2 kb foreign DNA inserted into the short single-copy (SSC) region and the IR region, respectively (Fig. 2a, b, purple and red bars and arrows). These two foreign sequences had no homologs in Convolvulaceae mitogenomes, nor were they similar to any sequences in NCBI nt database (no significant hits were yielded from BLASTN searches), so their origins were mysterious. Foreign DNAs in plastomes were mainly known to be transferred from mitogenome, such as in Apiaceae [23,24,25], Apocynaceae [26], Anacardiaceae [27], Orobanchaceae [28], and Poaceae [29,30,31,32], while transfers from other sources were rare [25]. The foreign insertions in Dinetus and Dichondra plastomes could represent two additional examples of non-mitochondrial origin.
Variações do mitogenoma em Convolvulaceae
Reunimos mitogenomas de 22 espécies da família Convolvulaceae, tornando este um dos poucos estudos que compreende um grande número de mitogenomas novos e completos em plantas (Fig. 1 c, Arquivo adicional 2 ). Mitogenomas completos foram obtidos para 18 destes, incluindo todas as espécies folhosas, Cuscuta epilinum , Cu. europaea e Cu. japonica , enquanto apenas os rascunhos foram obtidos para os quatro dodders do subgênero Grammica ( Cu. americana , Cu. australis , Cu. californica e Cu. campestris ) devido à grande quantidade de repetições. Mitogenomas em Convolvulaceae são principalmente cerca de 300 kb de comprimento, que é mais curto do que a maioria das angiospermas, incluindo seus parentes próximos Solanales. O conteúdo gênico dos mitogenomas de Convolvulaceae é semelhante ao de outras angiospermas (Arquivo adicional 2 ). Os genes “core” estão bem preservados, exceto ccmFc (veja abaixo). Comparado com Solanales, rpl2 e sdh3 estavam ausentes em todos os familiares investigados. O rps7 foi encontrado apenas em I. biflora , e a análise filogenética indicou que era mais próximo de Rosales (valor de bootstrap 99% com Morus notabilis e Cannabis sativa ; Arquivo adicional 3 , rps7 ). Portanto, o rps7 provavelmente foi perdido no ancestral comum de Convolvulaceae e I. biflora por transferência horizontal de gene/DNA (HGT). Os mitogenomas dos Grammica também foram os mais degenerados da família – eles perderam mais genes, semelhantes aos seus plastomas. Cu. epilinum , Cu. europaea e Cu. japonica não apresentou diferenças significativas no conteúdo gênico (Arquivos adicionais 1 e 2 ). A maioria dos genes mitocondriais em dodders teve taxas de substituição aumentadas, mas nenhum era de HGT (Arquivo adicional 3 ).
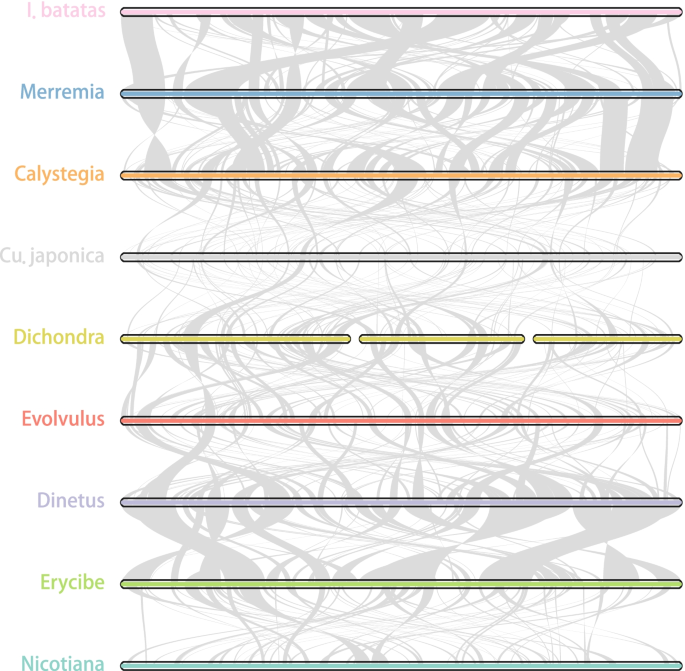
Em contraste com o tamanho geralmente pequeno da família, o mitogenoma em Cu. japonica ultrapassou 800 kb, atingindo o dobro dos outros (Fig. 1 c). Além disso, Dichondra , Evolvulus , Cu. epilinum e Cu. europaea também apresentou expansões. A sintenia mitogenômica de pares das tribos foi comparada, o que revelou mudanças rápidas na estrutura mitogenômica e conteúdo de DNA (Fig. 3 ). Para entender as causas da diferença, dissecamos as sequências mitogenômicas em cinco classes: conservadas (têm homólogos em todas as Convolvulaceae), plastid-like (potenciais inserções plastidiais), repetitivas, específicas de gênero ou/e espécie e outras (as repouso) (Fig. 1c ). Apenas uma pequena proporção de DNA existe em todos (principalmente regiões intragênicas; Fig. 1 c, a proporção “Conservada”). A diferença mais significativa foi de sequências específicas de gênero e/ou espécie (GSS, detalhes veja seção “Métodos”), especialmente em Cu. japonês .
Sintenia de mitogenomas em pares entre tribos. As barras indicavam os mitogenomas e as fitas mostravam as sequências homólogas entre as espécies adjacentes. Dichondra tem três cromossomos. Apenas uma espécie de cada tribo foi usada, e sua ordem se refere à árvore CDS plastidial na Fig. 5 . Tabaco ( Nicotiana tabacum , GenBank: NC_006581) também foi comparado
Para identificar a origem potencial, GSS de Dichondra , Evolvulus e Cu. japonica foram detonados contra nt e selecionados os melhores resultados (Arquivo adicional 4 ). Os melhores acertos foram agrupados em pedidos. Embora muitos deles tenham origens desconhecidas (ou seja, não obtiveram resultados das pesquisas do BLAST), a maioria dos GSS compartilhava alta similaridade com táxons distantemente relacionados (Fig. 4 a). Fabales, Solanales, Lamiales, Malpighiales e Gentianales ocuparam grandes proporções no GSS em todas as três espécies, o que pode indicar que sua expansão tem experimentado eventos semelhantes. Em contraste, Caryophyllales e Santalales em Evolvulus e Rosales, Sapindales, Apiales, etc., em Cu. japonica poderia implicar evolução independente.
Mitogenome variations in Convolvulaceae. a Orders of GSS best hits in Evolvulus, Dichondra, and Cu. japonica. The numbers showed the total length (bp) of each order. Only orders >5 kb are displayed. The white sectors showed the GSS had no homologs in NCBI nt database. b, c The two potential evolutionary paths of the small mitogenome size in subgenus Grammica—either degenerated after inflation or stayed small. Red, yellow, and blue branches indicated the large, middle, and small mitogenome sizes, respectively. d Significant best hits of Cu. japonica GSS (>500 bp, i.e., HGT-like) were used to search for homologs in other dodders, with the coverage shown in the heatmap. Only a few could be found with homologs (green cells). This supported the possibility that the large mitogenome size of Cu. japonica has evolved independently from other dodders
Divergent evolution of the mitogenomes in dodders
As plantas parasitas evoluíram pelo menos 12 vezes independentemente nas angiospermas [ 33 , 34 ], o que fornece um excelente modelo para estudar as interações vegetais e a evolução molecular. Seus genomas muitas vezes mostram características não comumente observadas em outras plantas, incluindo perdas de genes e HGTs [ 19 , 20 , 35 , 36 , 37 ]. Plastomas em plantas autotróficas verdes são muito conservados, enquanto aqueles em plantas parasitas sempre apresentam tamanho reduzido, perda de genes de fotossíntese, diferentes limites de IR e taxas de substituição ascendentes [ 38 ], como relatado para Cuscuta [ 10 , 11 , 18 ]. Para os mitogenomas, muitas plantas parasitárias obrigatórias exibiram extensas HGTs de hospedeiros, incluindo até mesmo as substituições dos genes nativos, como em Cynomorium (Cynomoriaceae [ 39 , 40 ];), Lophophytum mirabile (Balanophoraceae [ 41 , 42 ];, Viscum album ( Santalaceae [ 43 ];) e Aeginetia indica (Orobanchaceae [ 44 ];). Portanto, pode não ser surpreendente que Cu. japonica recebido grandes quantidades de DNA de outras espécies. Uma questão interessante é quão grande era o ancestral comum dos dodders em tamanho de mitogenoma - se o mitogenoma de Grammica dodders degraded or Cu. japonica expanded (Fig. 4b, c). We hypothesized that if the mitogenomes shrank from big to small, there should contain some remnants, even though the remnants were very short. We used the significant HGT-like sequences of Cu. japonica (Additional file 4, length >500 bp) as queries to search for homologs in the other six dodders and then calculated the coverage rate of each GSS. Only a few GSS yielded hits from the other dodders (Fig. 4d). It suggested that though dodders had some HGT-like sequences in common, most expansions in Cu. japonica occurred independently after the speciation. The mitogenome size evolved in a divergent manner in dodders. A similar situation was also observed for Viscum scurruloideum, which possesses the smallest mitogenome in angiosperms (only 66 kb) while its relative V. album outnumbers it 8.6-fold in size [45, 46]. Why some dodders and V. scurruloideum evolved in an opposite direction, and how they maintained a small mitogenome are intriguing questions to be answered in the future.
Relações filogenéticas
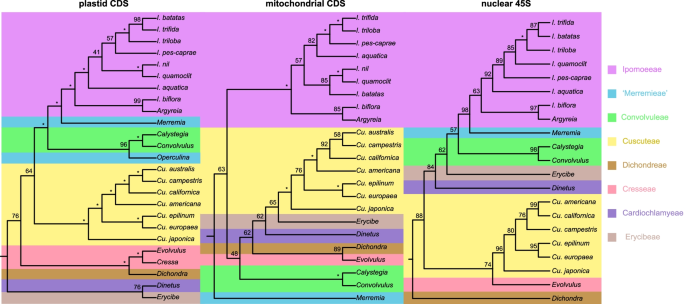
Sequências de sequências de codificação de proteína plastidial (CDS), CDS mitocondrial e 45S nuclear (rRNAs 18S, 5.8S e 25S e as regiões espaçadoras) foram empregadas para construir as árvores de máxima probabilidade (ML) de Convolvulaceae (Fig. 5 ; Adicional arquivo 5 ). Plastomos de Operculina macrocarpa (GenBank: KF242502) e Cressa cretica (NC_035516) foram incluídos. A matriz plastidial continha 77.711 colunas com 16.689 sítios informativos de parcimônia (PIS). A árvore plastidial tem uma topologia semelhante a estudos anteriores [ 3 , 4 , 5 ]. No entanto, as duas espécies de “Merremieae”, Merremia e Operculina , foram aninhadas com Convolvuleae. Merremieae foi sugerida como polifilética [ 5 , 13 ] e então reclassificada com base em vários marcadores de DNA e dados morfológicos [ 1 ]. Nosso resultado revelou que a monofilia de Merremieae provavelmente ainda era incerta. O apoio do clado Cuscuteae era baixo, o grupo irmão de dodders permaneceu sem solução.
A matriz mitocondrial tinha 30.753 colunas mas apenas com 1033 PIS. A árvore mitocondrial obteve uma topologia muito diferente e foi mal suportada. A capacidade das sequências mitocondriais na filogenia em nível de família ou ordem foi desafiada [ 47 ]. A árvore nuclear sugeriu uma relação mais próxima entre dodders e Erycibeae. No entanto, o suporte da árvore nuclear também foi fraco, o que também pode ser causado pela falta de PIS (5906 colunas com 548 PIS).
Todos os membros da família abrigam o ccmFc quebrado
Um fenômeno muito incomum foi observado no ccmFc . ccmFc é uma subunidade do sistema de maturação do citocromo c (CCM) e está envolvida na fase final do processo de maturação [ 48 , 49 ]. O c é um componente essencial da cadeia de transporte de elétrons mitocondrial e fornece elétrons entre os complexos III e IV. Tipicamente, o gene contém dois éxons e um cis íntron de Diferentemente, em todos os membros de Convolvulaceae, ela foi dividida em três fragmentos. A primeira quebra ocorreu perto da extremidade 3' do primeiro exon, causando um ca. 43 aa perda (entre as posições 164 a 206 no tomate); a segunda quebra ocorreu dentro do íntron sem perdas de base (Fig. 6 a). Nossos dados incluíram várias leituras de sequenciamento longo e não detectamos grandes repetições em torno ccmFc , argumentando contra a montagem incorreta. Nossa rica amostragem sugere que as quebras provavelmente ocorreram com o ancestral comum dessa família.
ccmFc encontrou duas quebras independentes e se tornou dois genes. a Pontos de interrupção em ccmFc . Uma inserção longa no segundo ponto de interrupção significava que FR2 e FR3 ainda são adjacentes em espécies folhosas. Em Cu. epilinum e Cu. europaea , a inserção é muito mais curta; a conexão quebrou em outro Cuscuta . b , c Cobertura do transcriptoma dos três fragmentos em batata-doce ( I. batatas ). Linhas azuis e vermelhas sob a cobertura indicavam cobertura menor que 50 e maior que 400, respectivamente. A cobertura foi dimensionada pela função Log em GENEIOUS. d Cobertura do transcriptoma das partes do exão após a remoção das regiões intergênicas. e Posições dos primers e análise do gel de agarose de RT-PCR confirmando a fissão do gene. Escalas de comprimento de genes indicadas no tomate ccmFc . Pistas 1 a 7: Cu. austral , Cu. japonica , batata doce, Dichondra , tomate, Arabidopsis e sem modelo
A distribuição genômica dos três ccmFc variou entre as espécies (Fig. 6 a). Em membros folhosos, o primeiro fragmento (FR1) formou um novo bloco de genes juntamente com nad5 exon 3, rps13 e ccmC (este bloco foi rearranjado em dodders). O segundo (FR2) e o terceiro (FR3) fragmentos ainda estavam conectados em espécies frondosas, com ca. inserção de 3,5 kb (2,8 kb de origem plastidial) no meio; em Cu. epilinum e Cu. europaea , a inserção foi reduzida para ca. 1,3 kb; enquanto em outros dodders, a conexão foi quebrada e FR2 e FR3 se separaram.
CcmFc encontrou fissão e um deslocamento de emenda
Considerando a rápida evolução das regiões não codificantes mitocondriais (Fig. 3 ), a presença de todos os três ccmFc em Convolvulaceae provavelmente implica que eles ainda são funcionais. Procuramos sequências de DNA e aminoácidos de ccmFc em genomas conhecidos de Convolvulaceae para detectar potenciais transferências nucleares, mas nenhuma correspondência de alta confiança foi obtida. Em seguida, mapeamos o transcriptoma de depleção de rRNA da batata-doce para o mitogenoma para verificar evidências de expressão ativa. Todos os três fragmentos produziram altos níveis de expressão (Fig. 6 b, c). FR1 formou uma nova unidade de transcrição com o nad5 exon 3 a jusante, rps13 e ccmC (Fig. 6 b). Nenhum local de splicing foi encontrado em torno da extremidade 3' de FR1, enquanto FR2 e FR3 tiveram leituras mapeadas para ambas as extremidades (Fig. 6 c, d). Estes resultados suportam FR1 tornando-se um gene independente (daqui em diante, ccmFc1 ), enquanto FR2 e FR3 são dois éxons de outro gene (daqui em diante, ccmFc2 ). Para validar isso, sintetizamos cDNA de batata-doce, Dichondra , Cu. austral , Cu. japonica , tomate e Arabidopsis a partir de mRNA usando transcriptase reversa (RT) e análises de PCR realizadas. O par de primers “F1 + R1” produziu uma banda em tomate e Arabidopsis mas não em espécies de Convolvulaceae (Fig. 6 e). Em contraste, o par de primers “F2 + R2” que deveria amplificar uma parte dentro da região contendo FR2 e FR3 produziu uma banda para todos os moldes de cDNA, servindo como controle positivo para a qualidade dos moldes de cDNA. Esses resultados suportam a ocorrência do evento de fissão deduzido dos dados do transcriptoma.
Os dois exons de ccmFc2 ainda estão concatenados na maioria dos membros da família, mas estão separados em alguns dodders (Fig. 6 b). A existência de cis - e trans -splicing em mitogenomas de angiospermas levanta a questão óbvia de como ccmFc2 é spliced. A seguinte evidência indicou que o splicing pode ser trans em todas as espécies. Por um lado, o pré-mRNA contendo íntrons sem splicing pode ser clonado, uma vez que primers aleatórios foram usados para transcrição reversa. Outra banda mais fraca em brilho foi observada no gel para tomate e Arabidopsis , mas não para espécies de Convolvulaceae (Fig. 6 e). Por outro lado, um cis íntron cis íntron de nad5 na Fig. 6 c). No entanto, o mapeamento transcriptômico da batata-doce apresentou uma cobertura relativamente baixa próxima a FR2 (Fig. 6 c, linha azul). Resultados semelhantes também foram obtidos a partir do trabalho muito recente dos dois dodders Cu. austral e Cu. campestris [ 50 ].
Sequências de codificação expandidas após a fissão
Junto com a fissão, houve um aumento de indels e bases divergentes nas regiões codificadoras de ccmFc , o que acelerou as taxas de substituição (ver arquivo adicional 3 , ccmFc1 e ccmFc2 ). Além disso, após a quebra, FR1 foi separado do códon de parada. Assim, exigia um novo códon de parada a jusante; da mesma forma, FR2 exigiu um novo códon de iniciação a montante. No entanto, determinamos que os códons de parada e iniciação de ATG mais próximos estavam distantes, resultando em um ca. 185 e 118 aa expansão de ccmFc1 e ccmFc2 , respectivamente. Novos sítios de edição de RNA estavam ausentes nas regiões gênicas expandidas. Nós clonamos o cDNA para confirmar a expansão do gene (Fig. 7 a). As expansões foram ainda confirmadas pelos dados de espectrometria de massa da batata-doce, com múltiplos acertos detectados durante a identificação do peptídeo (Fig. 7 b). Em dodders, ccmFc1 ganhou mutações independentes sem sentido, encurtando seu comprimento de volta ao ponto de interrupção (Fig. 7 c).
Coding regions of ccmFc1 and ccmFc2 expanded after breaking. a Position and direction of primers and agarose gel of cDNA confirming gene expansion. Gene lengths referred to sweet potato. Lanes 1 to 7 represent Cu. australis, Cu. japonica, sweet potato, Dichondra, tomato, Arabidopsis, and no template. b Peptides (shown by short green segments) were identified by mass spectrometry data. c Independent nonsense mutations truncated the expansion of ccmFc1 amino acid sequences in Cuscuta. Blue lines indicated the position of F1 and R3 primers. Red lines under the sequence showed coding regions. The vertical black dotted line indicated the breakpoint. d Transmembrane helices predicted by TMHMM: top, tomato ccmFc; middle, sweet potato ccmFc1; bottom, sweet potato ccmFc2
CcmFc is localized to the mitochondrial inner membrane and contains multiple predicted transmembrane helices in both exons (Fig. 7d [48];. However, in sweet potato, our predictions for ccmFc1 and ccmFc2 (performed by TMHMM Server 2.0 [51]) revealed four transmembrane helices in ccmFc1 but none in ccmFc2 (Fig. 7d). The breaks and expansions might have altered the protein structure.
Cis e trans-splicing podem não diferir muito
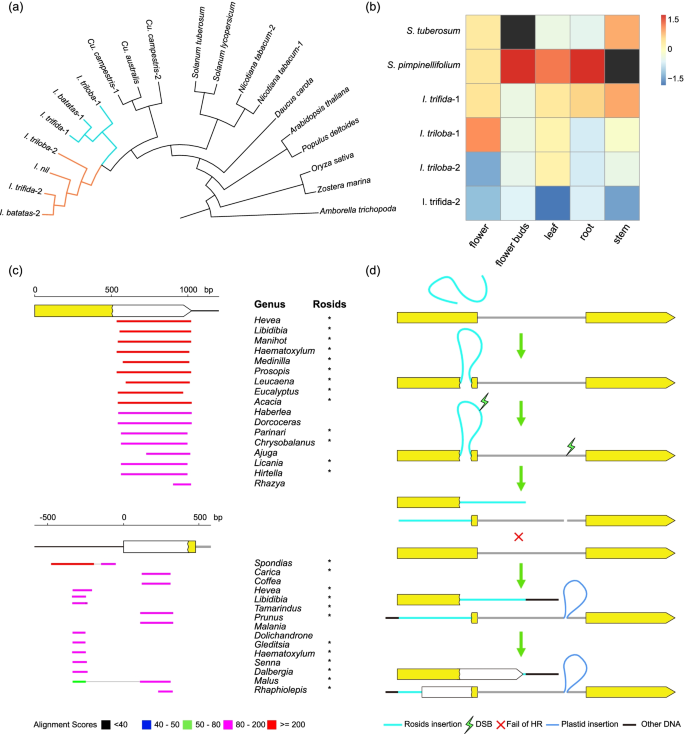
O mecanismo da mudança de cis - para trans -splicing permanece obscuro. O que é isso Fator 9 (WTF9), uma proteína de reconhecimento de RNA de organela vegetal codificada no núcleo, pode se ligar diretamente aos 48 nucleotídeos do ccmFc para regular o cis -splicing [ 49 , 52 ]. A proteína de choque térmico de chaperona molecular 60 (HSP60) interage com WTF9 durante este processo [ 52 ]. Para detectar possíveis alterações causadas pelo trans splicing, usamos Arabidopsis WTF9 e HSP60 como referências para busca de homólogos em genomas de Convolvulaceae e outras angiospermas. WTF9 é retido em todas as espécies de Convolvulaceae e até duplicado em Ipomoea (Fig. 8 a). Verificamos o nível de expressão de WTF9 em I. trifida , I. triloba , e duas espécies de Solanaceae, batata e S. pimpinellifolium . O padrão de expressão de Ipomoea WTF9-1 assemelhava-se ao de Solanum (Fig. 8 b). Situação semelhante foi observada para HSP60 (Arquivo adicional 6 ). Após o splicing shift, os dois fragmentos intrônicos foram quase inteiramente preservados em todas as espécies, incluindo a região de ligação de 48 nucleotídeos (exceto Cu. america , que tem uma inserção de 28 pb na região de ligação). Com base nisso, especulamos que o mecanismo entre cis - e trans -splicing pode ser semelhante, mas mais pesquisas são necessárias para testar essa hipótese.
Filogenia e expressão de WTF9 e uma potencial história evolutiva de ccmFc em Convolvulaceae. a Filogenia de WTF9 em angiospermas. Ramos coloridos em Ipomoea mostraram duplicação. b WTF9 de expressão de Solanum e Ipomoea . Caixas pretas indicavam dados ausentes. c Pesquisas BLASTN do downstream de FR1 e upstream de FR2 contra o banco de dados NCBI nt . Os principais acertos foram principalmente de rosídeos (*) depois de acertos mascarados de Convolvulaceae e outras plantas parasitas. d Uma trajetória evolutiva potencial de ccmFc em Convolvulaceae
Variações em Convolvulaceae CcmFc é inédita entre as angiospermas
Sob pressão de seleção e reparo por recombinação homóloga (HR), os genes mitocondriais de plantas evoluem mais lentamente do que outros compartimentos do genoma [ 53 , 54 ]. A estrutura mitogenômica nas angiospermas é excepcionalmente dinâmica, enquanto as sequências e a estrutura dos genes, bem como os métodos de splicing, são bem conservados [ 55 , 56 ]. Trans -splicing evoluiu independentemente várias vezes em plantas terrestres, como em licófitas e gimnospermas [ 57 , 58 , 59 , 60 ]. Em angiospermas, o surgimento de trans -splicing foi menos frequente. Além de nad1 , nad2 e nad5 , apenas cox2 em Allium foi relatado para sofrer trans -splicing [ 61 , 62 , 63 , 64 ]. Nossa descoberta de ccmFc em Convolvulaceae representa um novo tipo de trans -splicing em angiospermas. A fissão gênica também ocorreu várias vezes em mitogenomas de plantas, como ccmFc em Marchantia e ccmFn em Brassicaceae, Allium e Trifolium , causada por mutação de mudança de quadro baseada em deleção ou rearranjos [ 65 , 66 , 67 , 68 ]. Até onde sabemos, a fissão seguida de expansões encontrada neste estudo é a primeira vez relatada em mitogenomas. Entre eles, o ccmFc em Convolvulaceae pode ser o mais complicado, integrando fissão gênica, mudança de splicing e expansões. Também pesquisamos a jusante de FR1 e a montante de FR2 contra o banco de dados NCBI nt , descobrindo que ambos eram mais semelhantes às sequências mitocondriais de rosídeos após ataques mascarados de Convolvulaceae (Fig. 8 c; Arquivo adicional 7 ). Nós hipotetizamos que uma longa sequência mitocondrial de rosídeos inserido no ccmFc do ancestral comum Convolvulaceae ocasionalmente (Fig. 8 d). Então quebras de fita dupla (DSBs [ 69 ];) ocorreram dentro da inserção e do íntron. A inserção longa pode dificultar o reparo da mutação por meio do HR. O reparo não homólogo impreciso finalmente criou a fissão do gene e trouxe a inserção do plastídio para dentro do íntron. Este último pode ter interrompido a estrutura secundária padrão do íntron do grupo II, resultando em uma mudança de cis -splicing para trans . HGTs são difundidos entre os mitogenomas, mas principalmente não funcionais [ 70 , 71 ]. Em alguns casos, o HGT pode substituir os genes nativos total ou parcialmente [ 41 , 72 ]. Os quiméricos ccmFc1 e ccmFc2 em Convolvulaceae fundem novas sequências de codificação HGT e nativas, representando uma nova maneira de como HGT impacta funcionalmente o organismo.
Uma questão imediata é como essas mudanças modificariam a estrutura do complexo CCM ou mesmo a eficiência da fosforilação oxidativa. As elevadas taxas de substituição e as potenciais alterações nas hélices transmembranares podem ter afetado a estrutura da proteína. Estudos recentes mostraram que a modificação da maturação do citocromo c pode otimizar a transferência de elétrons extracelular para um maior fluxo de elétrons em Escherichia coli [ 73 ]. Nossos resultados fornecem um exemplo de diversidade respiratória das plantas, o que pode ter implicações para a engenharia da respiração das plantas no futuro.
Conclusões
Convolvulaceae . é famosa por seus amplos usos, diversas morfologias e diferentes modos tróficos Neste estudo, nossas análises em escala familiar revelaram que o mesmo espanto também existe em seus materiais genéticos. Plastídeos (incluindo cloroplastos) e mitocôndrias desempenham papéis cruciais no suprimento de energia celular e no metabolismo. Normalmente, a estrutura e os genes do plastoma e as sequências de codificação do mitogenoma são bem conservados. No entanto, apresenta algumas alterações em Convolvulaceae. Mesmo em membros de folhas verdes, os limites IR são todos atípicos, e também existem características incomuns, como perdas de íntrons, transferência nuclear ( infA ), duplicações e inserções estrangeiras. O mitogenoma do Cu. japonica foi significativamente inflado por sequências semelhantes a HGT, enquanto outros dodders preservados em um tamanho pequeno como seus parentes folhosos - o mitogenoma evolui de forma divergente em dodders. Além disso, os genes mitocondriais dodder têm mais perdas e taxas de substituição mais altas, mas nenhum evento HGT. A variação mais notável é a mitocondrial ccmFc . É altamente conservado em outras plantas, mas provavelmente encontrou fissão gênica, mudança de splicing e expansão de codificação no ancestral comum de Convolvulaceae, que é único em angiospermas. Os genes nucleares envolvidos no processo de splicing parecem ainda funcionais, levantando a questão do que difere entre o cis - e trans -splicing. Essas alterações incomuns de ccmFc provavelmente foram causadas pela inserção de HGT seguida por quebras de fita dupla e reparos imprecisos. Nossos dados fornecem recursos genéticos valiosos para estudar a evolução e filogenia de Convolvulaceae e parasitismo de plantas. Os resultados apresentados neste estudo também são úteis para entender a diversificação dos complexos mitocondriais e a inovação gênica.
Métodos
Amostragem e sequenciamento
Acessos de 12 espécies de Convolvulaceae foram coletados em diferentes locais da China (Arquivo adicional 8 ). As bibliotecas de sequenciamento foram preparadas usando um NEB Next® Ultra DNA Library Prep Kit e, em seguida, sequenciadas na plataforma Illumina Hiseq 4000 para gerar leituras de PE150. Dichondra micrantha também foi sequenciado usando a plataforma Oxford Nanopore promethION. Os dados brutos de sequenciamento para as outras 10 espécies foram obtidos do NCBI SRA ( https://www.ncbi.nlm.nih.gov/sra ). Os detalhes das leituras são fornecidos no arquivo adicional 9 .
Montagem e anotação do genoma
As leituras curtas foram filtradas usando TRIMMOMATIC v0.36 [ 74 ]. Plastomes foram montados usando GetOrganelle [ 75 ] e anotados usando I. nil (GenBank: NC_031159) como referência. A montagem de mitogenomas foi como descrito em nosso trabalho anterior [ 47 ]. Resumidamente, montagens de novo de leituras curtas e longas foram realizadas usando SPAdes v3.13.1 [ 76 ] e FLYE v2.8.3 [ 77 ], respectivamente. Em seguida, contigs mitocondriais foram identificados a partir de contigs totais por BLASTN contra o I. nil mitogenoma Esses contigs foram conectados e/ou estendidos manualmente no GENEIOUS R10 (Biomatters, Inc.) mapeando as leituras de volta e verificando ambas as extremidades. Repetições longas e inserções de plastídios foram os dois maiores desafios da montagem. As regiões repetidas foram resolvidas usando cobertura de sequenciamento de leitura curta; as inserções plastidiais foram resolvidas com base nas localizações de suas contrapartes plastidiais. O mitogenoma poderia obter um ou vários círculos após as repetições e inserções de plastídios terem sido devidamente determinadas. Cu. japonica produziu um cromossomo linear, uma vez que as repetições em ambas as extremidades não podiam ser conectadas. Os quatro dodders do subgênero Grammica ( Cu. americana , Cu. australis , Cu. californica e Cu. campestris ) só obteve mitogenomas de esboço devido às grandes quantidades de repetições. Genes de codificação de proteínas mitocondriais putativos e rRNA foram anotados com base na semelhança com genes mitocondriais conhecidos. tRNAs foram previstos usando tRNAscan-SE v2.0 [ 78 ]. A inserção do plastídio e o comprimento da repetição foram determinados por BLASTN v2.10.0+ [ 79 ] contra plastomas de Convolvulaceae (identidade > 90%) e ele próprio (identidade > 95%), respectivamente. As sequências comuns foram determinadas inicialmente por BLASTN (tamanho da palavra 16, e -valor 1e-5) e depois contadas usando scripts personalizados.
Análises de sintenia do genoma e sequências específicas de gênero
Plastome (IR removed) and mitogenome syntenies were plotted using Python version MCscan of JCVI 192 utility libraries v1.1.17 [80]. The DNA sequences were first divided into 50 bp fragments, and these fragments were then forced to be used as “genes” to search for homologous regions. Genus-specific sequences (GSS) meant sequences that shared no homology with other genera. For species in Ipomoea and Cuscuta, GSS also included the species-specific sequences. GSS were identified by searching each mitogenome against other Convolvulaceae mitogenomes, with e-value of 1e-5 and word size of 16. The GSS of Cu. japonica, Dichondra, and Evolvulus were searched against NCBI nt database to find the most potential donors, with only hits longer than 100 bp considered. Subsequently, the best matches were 184 grouped into orders (Additional file 3). Orders with total length >5 kb were plotted as pie charts in R (https://www.r-project.org/). To test whether other dodders have experienced expansion, high-confidence HGT-like sequences of Cu. japonica (best hits >500 bp) foram pesquisados contra outros dodders, com a porcentagem da cobertura calculada. Por fim, os mapas de calor foram gerados usando PHEATMAP ( https://github.com/raivokolde/pheatmap ) em R.
Análises filogenéticas
Foram detectados potenciais eventos HGT dos genes mitocondriais. CDS de Convolvulaceae e outras 42 plantas de sementes foram alinhadas usando MAFFT v7 [ 81 ] com o modo “auto”. Em seguida, as árvores de ML foram construídas usando IQTREE v1.6.12 [ 82 ] com parâmetro -bb 1000 -m GTR+G4+F -me 0,0001 runs 5 . As árvores foram plotadas usando FIGTREE v.1.4.2 ( http://tree.bio.ed.ac.uk/software/figtree/ ). Os acessos do GenBank foram listados atrás das espécies nas árvores (Arquivo adicional 3 ).
Os CDS plastidiais foram obtidos de nossos conjuntos e Ipomoea batatas (GenBank: NC_026703), I. nil (NC_031159), I. trifida (NC_034670), I. triloba (NC_037913), Cressa cretica (NC_035516), Cuscuta australis (NC_045885), Cu. campestris (NC_052920), Convolvulus arvensis (MW054627), Operculina macrocarpa (KF242502), Nicotiana tabacum (Z00044), Solanum melongena (MN218080) e Solanum lycopersicum (NC_007898). CDS mitocondrial foram obtidos de nossas montagens e I. nil (AP017303), N. tabacum (NC_006581), S. lycopersicum (NC_035963) e S. melongena (NC_050334). Os locais de edição de RNA foram previstos usando o site PREP (Mower, 2009) e removidos manualmente. Os 45S nucleares foram obtidos de novo contigs Nicotiana benthamiana (KP824745). e mitocondrial alinhados ( rps7 foi excluído de um HGT) CDS foram concatenados em uma matriz, respectivamente. Em seguida, os três conjuntos de dados (CDS plastidial, CDS mitocondrial e 45S nuclear) foram construídos as árvores ML como mencionado acima. As solanáceas sempre foram utilizadas como grupos externos.
Identificação de peptídeos a partir de dados de proteômica publicados
Dados de espectrometria de massa de proteína para batata-doce ( I. batatas ) foram baixados de ProteomeXchange PXD012999 [ 83 ]. Uma pesquisa de cometa Trans-Proteomic Pipeline (TPP) v5.2.0 [ 84 ] foi usada para identificação de peptídeos com parâmetros padrão.
Gene family analyses
Sequências de aminoácidos de Arabidopsis thaliana WTF9 (AT3G24320), HSP60-2 (AT2G33210), HSP60-3A (AT3G13860) e HSP60-3B (AT3G23990) foram usadas como referências para pesquisar homólogos em Convolvulaceae, incluindo batata-doce [ 85 ], I. trifida e I. triloba [ 86 ], I. nil [ 15 ], Cuscuta australis [ 19 ] e Cu. campestris [ 20 ], e outras angiospermas, incluindo Nicotiana tabacum [ 87 ], Amborella trichopoda v1.0 [ 88 ], Daucus carota v2.0 [ 89 ], Populus trichocarpa v3.0 [ 90 ], Oryza sativa v7 [ 91 ], tomate Solanum lycopersicum iTAG2.4 [ 92 ], batata Solanum tuberosum v4.03 [ 93 ] e Zostera marina v2.2 [ 94 ] (os últimos sete foram baixados do Phytozome v13: https://phytozome.jgi.doe.gov / ). As sequências de aminoácidos foram então alinhadas através de MAFFT, e as árvores foram então reconstruídas usando IQTREE (parâmetro: -bb 1000 -m LG+F+G4 execuções 10 -me 0,0001 ).
Os dados de nível de expressão de Ipomoea trifida e I. triloba foram obtidos de Sweetpotato Genomics Resource [ 86 ]; os de Solanum pimpinellifolium foram do Tomato Functional Genomics Database [ 95 ] e os de batata foram do SpudDB [ 96 ]. Os mapas de calor foram plotados usando PHEATMAP em R ( https://r-charts.com/correlation/pheatmap/ ) normalizando cada gene com Z-score.
Exame de expressão
Transcriptomas enriquecidos com oligo-dT introduziriam viés, uma vez que os transcritos de organelas geralmente não contêm uma cauda poliA [ 97 ]. Com base nisso, dados do transcriptoma de depleção de rRNA de batata-doce (SRA: SRR10538086 [ 98 ]) foram empregados e mapeados para o mitogenoma para verificar a expressão.
Folhas frescas de batata-doce, Dichondra , tomate, Arabidopsis e videiras frescas de Cuscuta australis e Cu. japonica foram usados para extração de RNA usando um kit de isolamento de RNA Omega e transcritos de forma reversa em cDNA usando primers aleatórios. Primers usados para RT-PCR foram projetados usando GENEIOUS (Arquivo adicional 10 ).
Disponibilidade de dados e materiais
As leituras brutas foram depositadas no NCBI sob BioProject PRJNA737311 e acesso SRR14812959 - SRR14812971 ( https://www.ncbi.nlm.nih.gov/bioproject/PRJNA737311 ) [ 99 ]. Os mitogenomas e plastomas montados foram depositados no NCBI sob os acessos MZ240723 - MZ240750 e BK059236 - BK059244 (TPA) e CNGBdb sob o projeto CNP0001927 e os acessos N_000011218 - N_000011230 e N_000011273 - N_000011309 ( https /CNP0001927 ). Os scripts usados podem ser encontrados no Github ( https://github.com/fengyanlei33/Convolvulaceae-mitogenome-project ) [ 100 ].
Abreviaturas
- RI:
-
Repetição invertida
- LSC:
-
Cópia única longa
- SSC:
-
Cópia única curta
- HGT:
-
Horizontal gene transfer
- GSS:
-
Sequências específicas de gênero e/ou espécie
- CDS:
-
Sequências de codificação
- ML:
-
Probabilidade máxima
- PIS:
-
Sites informativos de parcimônia
- MCC:
-
do citocromo c Maturação
- FR1:
-
O primeiro fragmento
- FR2:
-
O segundo fragmento
- FR3:
-
O terceiro fragmento
- RT:
-
Transcriptase reversa
- WTF9:
-
O que é esse fator 9
- HSP60:
-
Proteína de choque térmico 60
- RH:
-
Recombinação homóloga
- DSBs:
-
Quebras de fita dupla
Referências
Simoes AR, Staples G. Dissolução da tribo Convolvulaceae Merremieae e uma nova classificação dos gêneros constituintes. Bot J Linn Soc. 2017;183(4):561–86. https://doi.org/10.1093/botlinnean/box007 .
Stevens PF. Site de Filogenia de Angiospermas. Versão 14, 2017. 2001 em diante. http://www.mobot.org/MOBOT/research/APweb/ .
Stefanovic S, Krueger L, Olmstead RG. Monofilia das Convolvulaceae e circunscrição de suas principais linhagens com base em sequências de DNA de múltiplos loci de cloroplastos. Sou J Bot. 2002;89(9):1510–22. https://doi.org/10.3732/ajb.89.9.1510 .
Stefanovic S, Olmstead RG. Testando a posição filogenética de uma planta parasita (Cuscuta, Convolvulaceae, asteridae): inferência Bayesiana e bootstrap paramétrico em dados extraídos de três genomas. Syst Biol. 2004;53(3):384–99. https://doi.org/10.1080/10635150490445896 .
Stefanovic S, Austin D, Olmstead R. Classificação de Convolvulaceae: uma abordagem filogenética. Bot do sistema. 2009;28:791–806. https://doi.org/10.1043/02-45.1 .
Eserman LA, Tiley GP, Jarret RL, Leebens-Mack JH, Miller RE. Filogenética e diversificação de glórias-da-manhã (tribo Ipomoeeae, Convolvulaceae) com base em sequências plastômicas inteiras. Sou J Bot. 2014;101(1):92–103. https://doi.org/10.3732/ajb.1300207 .
Park I, Yang S, Kim WJ, Noh P, Lee HO, Moon BC. Os genomas completos de cloroplastos de seis espécies de ipomoea e desenvolvimento de marcadores indel para a discriminação de sêmen autêntico de pharbitidis (sementes de I. nil ou I. purpurea). Frente. Planta Sci. 2018;9:965. https://doi.org/10.3389/fpls.2018.00965 .
Sun J, Dong X, Cao Q, Xu T, Zhu M, Sun J, et al. Uma comparação sistemática de oito novas sequências de plastoma de Ipomoea L. PeerJ. 2019;7:e6563. https://doi.org/10.7717/peerj.6563 .
Funk HT, Berg S, Krupinska K, Maier UG, Krause K. Complete DNA sequences of the plastid genomes of two parasitic flowering plant species, Cuscuta reflexa and Cuscuta gronovii. BMC Plant Biol. 2007;7:45. https://doi.org/10.1186/1471-2229-7-45.
McNeal JR, Kuehl JV, Boore JL, de Pamphilis CW. Complete plastid genome sequences suggest strong selection for retention of photosynthetic genes in the parasitic plant genus Cuscuta. BMC Plant Biol. 2007;7. https://doi.org/10.1186/1471-2229-7-57.
Braukmann T, Kuzmina M, Stefanovic S. Plastid genome evolution across the genus Cuscuta (Convolvulaceae): two clades within subgenus Grammica exhibit extensive gene loss. J Exp Bot. 2013;64(4):977–89. https://doi.org/10.1093/jxb/ers391.
Banerjee A, Stefanović S. Reconstructing plastome evolution across the phylogenetic backbone of the parasitic plant genus Cuscuta (Convolvulaceae). Bot J Linn Soc. 2020;194(4):423–38. https://doi.org/10.1093/botlinnean/boaa056.
Simoes AR, Culham A, Carine M. Resolving the unresolved tribe: a molecular phylogenetic framework for the Merremieae (Convolvulaceae). Bot J Linn Soc. 2015;179(3):374–87. https://doi.org/10.1111/boj.12339.
Shidhi PR, Biju VC, Anu S, Vipin CL, Deelip KR, Achuthsankar SN. Genome characterization, comparison and phylogenetic analysis of complete mitochondrial genome of evolvulus alsinoides reveals highly rearranged gene order in solanales. Life (Basel). 2021;11(8). https://doi.org/10.3390/life11080769.
Hoshino A, Jayakumar V, Nitasaka E, Toyoda A, Noguchi H, Itoh T, et al. Genome sequence and analysis of the Japanese morning glory Ipomoea nil. Nat Commun. 2016;7:13295. https://doi.org/10.1038/ncomms13295.
Wicke S, Muller KF, de Pamphilis CW, Quandt D, Wickett NJ, Zhang Y, et al. Mechanisms of functional and physical genome reduction in photosynthetic and nonphotosynthetic parasitic plants of the broomrape family. Plant Cell. 2013;25(10):3711–25. https://doi.org/10.1105/tpc.113.113373.
Feng YL, Wicke S, Li JW, Han Y, Lin CS, Li DZ, et al. Reduções específicas de linhagem de genomas plastidiais em uma tribo de orquídeas com espécies parcialmente e totalmente micoheterotróficas. Genoma Biol Evol. 2016;8(7):2164–75. https://doi.org/10.1093/gbe/evw144 .
Revill MJ, Stanley S, Hibberd JM. Estrutura do genoma plastidial e perda da capacidade fotossintética no parasita do gênero Cuscuta. J Exp Bot. 2005;56(419):2477–86. https://doi.org/10.1093/jxb/eri240 .
Sun G, Xu Y, Liu H, Sun T, Zhang J, Hettenhausen C, et al. Perdas de genes em larga escala estão subjacentes à evolução do genoma da planta parasita Cuscuta australis. Nat Comun. 2018;9(1):2683. https://doi.org/10.1038/s41467-018-04721-8 .
Vogel A, Schwacke R, Denton AK, Usadel B, Hollmann J, Fischer K, et al. Pegadas de parasitismo no genoma da planta parasita Cuscuta campestris. Nat Comun. 2018;9(1):2515. https://doi.org/10.1038/s41467-018-04344-z .
McNeal JR, Kuehl JV, Boore JL, Leebens-Mack J, dePamphilis CW. Perda paralela de íntrons plastidiais e sua maturase no gênero Cuscuta. Plos Um. 2009;4(6):e5982. https://doi.org/10.1371/journal.pone.0005982 .
Millen RS, Olmstead RG, Adams KL, Palmer JD, Lao NT, Heggie L, et al. Muitas perdas paralelas de infA do DNA do cloroplasto durante a evolução das angiospermas com múltiplas transferências independentes para o núcleo. Célula Vegetal. 2001;13(3):645–58. https://doi.org/10.1105/tpc.13.3.645 .
Iorizzo M, Senalik D, Szklarczyk M, Grzebelus D, Spooner D, Simon P. A montagem de novo do genoma mitocondrial da cenoura usando o sequenciamento de próxima geração de DNA genômico inteiro fornece a primeira evidência de transferência de DNA para um genoma plastidial de angiosperma. BMC Plant Biol. 2012;12(1):61. https://doi.org/10.1186/1471-2229-12-61 .
Downie SR, Jansen RK. Uma análise comparativa de genomas de plastídeos inteiros dos apiales: expansão e contração da repetição invertida, transferência de DNA mitocondrial para plastídeo e identificação de regiões não codificantes altamente divergentes. Bot do sistema. 2015;40(1):336–51. https://doi.org/10.1600/036364415x686620 .
Spooner DM, Ruess H, Iorizzo M, Senalik D, Simon P. Filogenia plastidial inteira do gênero cenoura (Daucus, Apiaceae): concordância com dados nucleares e inserções de DNA mitocondrial e nuclear no plastídio. Sou J Bot. 2017;104(2):296–312. https://doi.org/10.3732/ajb.1600415 .
Straub SCK, Cronn RC, Edwards C, Fishbein M, Liston A. Transferência horizontal de DNA do genoma mitocondrial para o plastídeo e sua subsequente evolução em serralhas (Apocynaceae). Genoma Biol Evol. 2013;5(10):1872–85. https://doi.org/10.1093/gbe/evt140 .
Rabah SO, Lee C, Hajrah NH, Makki RM, Alharby HF, Alhebshi AM, et al. Plastome sequencing of ten nonmodel crop species uncovers a large insertion of mitochondrial DNA in cashew. Plant. Genome. 2017;10(3). https://doi.org/10.3835/plantgenome2017.03.0020.
Gandini CL, Sanchez-Puerta MV. Foreign plastid sequences in plant mitochondria are frequently acquired via mitochondrion-to-mitochondrion horizontal transfer. Sci Rep. 2017;7:43402. https://doi.org/10.1038/srep43402.
Ma PF, Zhang YX, Guo ZH, Li DZ. Evidence for horizontal transfer of mitochondrial DNA to the plastid genome in a bamboo genus. Sci Rep. 2015;5:11608. https://doi.org/10.1038/srep11608.
Saarela JM, Wysocki WP, Barrett CF, Soreng RJ, Davis JI, Clark LG, et al. Plastid phylogenomics of the cool-season grass subfamily: clarification of relationships among early-diverging tribes. AoB Plants. 2015;7. https://doi.org/10.1093/aobpla/plv046.
Wysocki WP, Clark LG, Attigala L, Ruiz-Sanchez E, Duvall MR. Evolution of the bamboos (Bambusoideae; Poaceae): a full plastome phylogenomic analysis. BMC Evol Biol. 2015;15:50. https://doi.org/10.1186/s12862-015-0321-5.
Burke SV, Ungerer MC, Duvall MR. Investigation of mitochondrial-derived plastome sequences in the Paspalum lineage (Panicoideae; Poaceae). BMC Plant Biol. 2018;18(1):152. https://doi.org/10.1186/s12870-018-1379-1.
Westwood JH, Yoder JI, Timko MP, dePamphilis CW. The evolution of parasitism in plants. Trends Plant Sci. 2010;15(4):227–35. https://doi.org/10.1016/j.tplants.2010.01.004.
Nickrent DL. Parasitic angiosperms: how often and how many? Taxon. 2020;69(1):5–27. https://doi.org/10.1002/tax.12195.
Yoshida S, Kim S, Wafula EK, Tanskanen J, Kim YM, Honaas L, et al. Genome sequence of striga asiatica provides insight into the evolution of plant parasitism. Curr Biol. 2019;29(18):3041–3052 e3044. https://doi.org/10.1016/j.cub.2019.07.086.
Cai L, Arnold BJ, Xi Z, Khost DE, Patel N, Hartmann CB, et al. Deeply altered genome architecture in the endoparasitic flowering plant Sapria himalayana Griff. (Rafflesiaceae). Curr Biol. 2021;31(5):1002–1011 e1009. https://doi.org/10.1016/j.cub.2020.12.045.
Lyko P, Wicke S. Genomic reconfiguration in parasitic plants involves considerable gene losses alongside global genome size inflation and gene births. Plant Physiol. 2021. https://doi.org/10.1093/plphys/kiab192.
Wicke S, Naumann J. Molecular evolution of plastid genomes in parasitic flowering plants. In: Chaw S-M, Jansen RK, editors. Advances in botanical research: Academic Press; 2018. p. 315–47. https://doi.org/10.1016/bs.abr.2017.11.014.
Bellot S, Cusimano N, Luo S, Sun G, Zarre S, Groger A, et al. Assembled plastid and mitochondrial genomes, as well as nuclear genes, place the parasite family Cynomoriaceae in the Saxifragales. Genome Biol Evol. 2016;8(7):2214–30. https://doi.org/10.1093/gbe/evw147.
Cusimano N, Renner SS. Sequential horizontal gene transfers from different hosts in a widespread Eurasian parasitic plant, Cynomorium coccineum. Am J Bot. 2019;106(5):679–89. https://doi.org/10.1002/ajb2.1286.
Sanchez-Puerta MV, Garcia LE, Wohlfeiler J, Ceriotti LF. Unparalleled replacement of native mitochondrial genes by foreign homologs in a holoparasitic plant. New Phytol. 2017;214(1):376–87. https://doi.org/10.1111/nph.14361.
Sanchez-Puerta MV, Edera A, Gandini CL, Williams AV, Howell KA, Nevill PG, et al. Genome-scale transfer of mitochondrial DNA from legume hosts to the holoparasite Lophophytum mirabile (Balanophoraceae). Mol Phylogenet Evol. 2018;132:243–50. https://doi.org/10.1016/j.ympev.2018.12.006.
Petersen G, Cuenca A, Moller IM, Seberg O. Massive gene loss in mistletoe (Viscum, Viscaceae) mitochondria. Sci Rep. 2015;5:17588. https://doi.org/10.1038/srep17588.
Choi K-S, Park S. Complete plastid and mitochondrial genomes of aeginetia indica reveal intracellular gene transfer (IGT), horizontal gene transfer (HGT), and cytoplasmic male sterility (CMS). Int J Mol Sci. 2021;22(11). https://doi.org/10.3390/ijms22116143.
Skippington E, Barkman TJ, Rice DW, Palmer JD. Comparative mitogenomics indicates respiratory competence in parasitic Viscum despite loss of complex I and extreme sequence divergence, and reveals horizontal gene transfer and remarkable variation in genome size. BMC Plant Biol. 2017;17(1):49. https://doi.org/10.1186/s12870-017-0992-8.
Skippington E, Barkman TJ, Rice DW, Palmer JD. Miniaturized mitogenome of the parasitic plant Viscum scurruloideum is extremely divergent and dynamic and has lost all nad genes. Proc Natl Acad Sci U S A. 2015;112(27):E3515–24. https://doi.org/10.1073/pnas.1504491112.
Feng Y, Xiang X, Akhter D, Pan R, Fu Z, Jin X. Mitochondrial phylogenomics of fagales provides insights into plant mitogenome mosaic evolution. Front Plant Sci. 2021;12. https://doi.org/10.3389/fpls.2021.762195.
Rayapuram N, Hagenmuller J, Grienenberger JM, Bonnard G, Giege P. The three mitochondrial encoded CcmF proteins form a complex that interacts with CCMH and c-type apocytochromes in Arabidopsis. J Biol Chem. 2008;283(37):25200–8. https://doi.org/10.1074/jbc.M802621200.
des Francs-Small CC, Kroeger T, Zmudjak M, Ostersetzer-Biran O, Rahimi N, Small I, et al. A PORR domain protein required for rpl2 and ccmFC intron splicing and for the biogenesis of c-type cytochromes in Arabidopsis mitochondria. Plant J. 2012;69(6):996–1005. https://doi.org/10.1111/j.1365-313X.2011.04849.x.
Anderson BM, Krause K, Petersen G. Mitochondrial genomes of two parasitic Cuscuta species lack clear evidence of horizontal gene transfer and retain unusually fragmented ccmFC genes. BMC Genomics. 2021;22(1):816. https://doi.org/10.1186/s12864-021-08105-z.
Moller S, Croning MD, Apweiler R. Evaluation of methods for the prediction of membrane spanning regions. Bioinformatics. 2001;17(7):646–53. https://doi.org/10.1093/bioinformatics/17.7.646.
Hsu YW, Juan CT, Wang CM, Jauh GY. Mitochondrial heat shock protein 60s interact with what's this factor 9 to regulate RNA splicing of ccmFC and rpl2. Plant Cell Physiol. 2019;60(1):116–25. https://doi.org/10.1093/pcp/pcy199.
Wolfe KH, Li WH, Sharp PM. Rates of nucleotide substitution vary greatly among plant mitochondrial, chloroplast, and nuclear DNAs. Proc Natl Acad Sci U S A. 1987;84(24):9054–8. https://doi.org/10.1073/pnas.84.24.9054.
Palmer JD, Herbon LA. Plant mitochondrial DNA evolves rapidly in structure, but slowly in sequence. J Mol Evol. 1988;28(1-2):87–97. https://doi.org/10.1007/BF02143500.
Cole LW, Guo W, Mower JP, Palmer JD. High and variable rates of repeat-mediated mitochondrial genome rearrangement in a genus of plants. Mol Biol Evol. 2018;35(11):2773–85. https://doi.org/10.1093/molbev/msy176.
Mower JP. Variation in protein gene and intron content among land plant mitogenomes. Mitochondrion. 2020;53:203–13. https://doi.org/10.1016/j.mito.2020.06.002.
Grewe F, Viehoever P, Weisshaar B, Knoop V. A trans-splicing group I intron and tRNA-hyperediting in the mitochondrial genome of the lycophyte Isoetes engelmannii. Nucleic Acids Res. 2009;37(15):5093–104. https://doi.org/10.1093/nar/gkp532.
Hecht J, Grewe F, Knoop V. Extreme RNA editing in coding islands and abundant microsatellites in repeat sequences of selaginella moellendorffii mitochondria: the root of frequent plant mtDNA recombination in early tracheophytes. Genome Biol Evol. 2011;3:344–58. https://doi.org/10.1093/gbe/evr027.
Guo W, Grewe F, Fan W, Young GJ, Knoop V, Palmer JD, et al. Ginkgo and Welwitschia mitogenomes reveal extreme contrasts in gymnosperm mitochondrial evolution. Mol Biol Evol. 2016;33(6):1448–60. https://doi.org/10.1093/molbev/msw024.
Guo W, Zhu A, Fan W, Adams RP, Mower JP. Extensive shifts from cis- to trans-splicing of gymnosperm mitochondrial introns. Mol Biol Evol. 2020;37(6):1615–20. https://doi.org/10.1093/molbev/msaa029.
Malek O, Knoop V. Trans-splicing group II introns in plant mitochondria: the complete set of cis-arranged homologs in ferns, fern allies, and a hornwort. RNA (New York, NY). 1998;4(12):1599–609.
Qiu YL, Palmer JD. Many independent origins of trans splicing of a plant mitochondrial group II intron. J Mol Evol. 2004;59(1):80–9. https://doi.org/10.1007/s00239-004-2606-y.
Kim S, Yoon MK. Comparison of mitochondrial and chloroplast genome segments from three onion (Allium cepa L.) cytoplasm types and identification of a trans-splicing intron of cox2. Curr Genet. 2010;56(2):177–88. https://doi.org/10.1007/s00294-010-0290-6.
Kim S, Bang H, Patil BS. Origin of three characteristic onion (Allium cepa L.) mitochondrial genome rearrangements in Allium species. Sci Hortic. 2013;157:24–31. https://doi.org/10.1016/j.scienta.2013.04.009.
Oda K, Yamato K, Ohta E, Nakamura Y, Takemura M, Nozato N, et al. Gene organization deduced from the complete sequence of liverwort Marchantia polymorpha mitochondrial DNA. A primitive form of plant mitochondrial genome. J Mol Biol. 1992;223(1):1–7.
Handa H, Bonnard G, Grienenberger JM. The rapeseed mitochondrial gene encoding a homologue of the bacterial protein Ccl1 is divided into two independently transcribed reading frames. MGG Mol Gen Genet. 1996;252(3):292–302. https://doi.org/10.1007/bf02173775.
Kim B, Kim K, Yang TJ, Kim S. Completion of the mitochondrial genome sequence of onion (Allium cepa L.) containing the CMS-S male-sterile cytoplasm and identification of an independent event of the ccmF N gene split. Curr Genet. 2016;62(4):873–85. https://doi.org/10.1007/s00294-016-0595-1.
Choi IS, Ruhlman TA, Jansen RK. Comparative mitogenome analysis of the genus trifolium reveals independent gene fission of ccmFn and intracellular gene transfers in Fabaceae. Int J Mol Sci. 2020;21(6). https://doi.org/10.3390/ijms21061959.
Christensen AC. Genes and junk in plant mitochondria-repair mechanisms and selection. Genome Biol Evol. 2014;6(6):1448–53. https://doi.org/10.1093/gbe/evu115.
Bergthorsson U, Adams KL, Thomason B, Palmer JD. Widespread horizontal transfer of mitochondrial genes in flowering plants. Nature. 2003;424:197. https://doi.org/10.1038/nature01743.
Rice DW, Alverson AJ, Richardson AO, Young GJ, Sanchez-Puerta MV, Munzinger J, et al. Horizontal transfer of entire genomes via mitochondrial fusion in the angiosperm amborella. Science. 2013;342(6165):1468.
Park S, Ruhlman TA, Weng ML, Hajrah NH, Sabir JSM, Jansen RK. Contrasting patterns of nucleotide substitution rates provide insight into dynamic evolution of plastid and mitochondrial genomes of geranium. Genome Biol Evol. 2017;9(6):1766–80. https://doi.org/10.1093/gbe/evx124.
Su L, Fukushima T, Prior A, Baruch M, Zajdel TJ, Ajo-Franklin CM. Modifying cytochrome c maturation can increase the bioelectronic performance of engineered Escherichia coli. ACS Synth Biol. 2020;9(1):115–24. https://doi.org/10.1021/acssynbio.9b00379.
Bolger AM, Lohse M, Usadel B. Trimmomatic: a flexible trimmer for Illumina sequence data. Bioinformatics. 2014;30(15):2114–20. https://doi.org/10.1093/bioinformatics/btu170.
Jin JJ, Yu WB, Yang JB, Song Y, dePamphilis CW, Yi TS, et al. GetOrganelle: a fast and versatile toolkit for accurate de novo assembly of organelle genomes. Genome Biol. 2020;21(1):241. https://doi.org/10.1186/s13059-020-02154-5.
Bankevich A, Nurk S, Antipov D, Gurevich AA, Dvorkin M, Kulikov AS, et al. SPAdes: a new genome assembly algorithm and its applications to single-cell sequencing. J Comput Biol. 2012;19(5):455–77. https://doi.org/10.1089/cmb.2012.0021.
Kolmogorov M, Yuan J, Lin Y, Pevzner PA. Assembly of long, error-prone reads using repeat graphs. Nat Biotechnol. 2019;37(5):540–6. https://doi.org/10.1038/s41587-019-0072-8.
Chan PP, Lowe TM. tRNAscan-SE: searching for tRNA genes in genomic sequences. Methods Mol Biol. 2019;1962:1–14. https://doi.org/10.1007/978-1-4939-9173-0_1.
Camacho C, Coulouris G, Avagyan V, Ma N, Papadopoulos J, Bealer K, et al. BLAST plus : architecture and applications. BMC Bioinformatics. 2009;10. https://doi.org/10.1186/1471-2105-10-421.
Tang H, Bowers JE, Wang X, Ming R, Alam M, Paterson AH. Synteny and collinearity in plant genomes. Science. 2008;320(5875):486–8. https://doi.org/10.1126/science.1153917.
Katoh K, Standley DM. MAFFT multiple sequence alignment software version 7: improvements in performance and usability. Mol Biol Evol. 2013;30(4):772–80. https://doi.org/10.1093/molbev/mst010.
Nguyen LT, Schmidt HA, von Haeseler A, Minh BQ. IQ-TREE: a fast and effective stochastic algorithm for estimating maximum-likelihood phylogenies. Mol Biol Evol. 2015;32(1):268–74. https://doi.org/10.1093/molbev/msu300.
Al-Mohanna T, Ahsan N, Bokros NT, Dimlioglu G, Reddy KR, Shankle M, et al. Proteomics and proteogenomics analysis of sweetpotato ( Ipomoea batatas) Leaf and Root. J Proteome Res. 2019;18(7):2719–34. https://doi.org/10.1021/acs.jproteome.8b00943.
Deutsch EW, Mendoza L, Shteynberg D, Slagel J, Sun Z, Moritz RL. Trans-Proteomic Pipeline, a standardized data processing pipeline for large-scale reproducible proteomics informatics. Proteomics Clin Appl. 2015;9(7-8):745–54. https://doi.org/10.1002/prca.201400164.
Yang J, Moeinzadeh MH, Kuhl H, Helmuth J, Xiao P, Haas S, et al. Haplotype-resolved sweet potato genome traces back its hexaploidization history. Nat Plants. 2017;3(9):696–703. https://doi.org/10.1038/s41477-017-0002-z.
Wu S, Lau KH, Cao Q, Hamilton JP, Sun H, Zhou C, et al. Genome sequences of two diploid wild relatives of cultivated sweetpotato reveal targets for genetic improvement. Nat Commun. 2018;9(1):4580. https://doi.org/10.1038/s41467-018-06983-8.
Sierro N, Battey JN, Ouadi S, Bakaher N, Bovet L, Willig A, et al. The tobacco genome sequence and its comparison with those of tomato and potato. Nat Commun. 2014;5:3833. https://doi.org/10.1038/ncomms4833.
Amborella Genome P. The Amborella genome and the evolution of flowering plants. Science. 2013;342(6165):1241089. https://doi.org/10.1126/science.1241089.
Iorizzo M, Ellison S, Senalik D, Zeng P, Satapoomin P, Huang J, et al. A high-quality carrot genome assembly provides new insights into carotenoid accumulation and asterid genome evolution. Nat Genet. 2016;48(6):657–66. https://doi.org/10.1038/ng.3565.
Tuskan GA, Difazio S, Jansson S, Bohlmann J, Grigoriev I, Hellsten U, et al. The genome of black cottonwood, Populus trichocarpa (Torr. & Gray). Science. 2006;313(5793):1596–604. https://doi.org/10.1126/science.1128691.
Ouyang S, Zhu W, Hamilton J, Lin H, Campbell M, Childs K, et al. The TIGR Rice Genome Annotation Resource: improvements and new features. Nucleic Acids Res. 2007;35(Database issue):D883–7. https://doi.org/10.1093/nar/gkl976.
Tomato GC. The tomato genome sequence provides insights into fleshy fruit evolution. Nature. 2012;485(7400):635–41. https://doi.org/10.1038/nature11119.
Sharma SK, Bolser D, de Boer J, Sonderkaer M, Amoros W, Carboni MF, et al. Construction of reference chromosome-scale pseudomolecules for potato: integrating the potato genome with genetic and physical maps. G3 (Bethesda). 2013;3(11):2031–47. https://doi.org/10.1534/g3.113.007153.
Olsen JL, Rouze P, Verhelst B, Lin YC, Bayer T, Collen J, et al. The genome of the seagrass Zostera marina reveals angiosperm adaptation to the sea. Nature. 2016;530(7590):331–5. https://doi.org/10.1038/nature16548.
Fei Z, Joung JG, Tang X, Zheng Y, Huang M, Lee JM, et al. Tomato Functional Genomics Database: a comprehensive resource and analysis package for tomato functional genomics. Nucleic Acids Res. 2011;39(Database issue):D1156–63. https://doi.org/10.1093/nar/gkq991.
Hirsch CD, Hamilton JP, Childs KL, Cepela J, Crisovan E, Vaillancourt B, et al. Spud DB: a resource for mining sequences, genotypes, and phenotypes to accelerate potato breeding. The Plant. Genome. 2014;7(1). https://doi.org/10.3835/plantgenome2013.12.0042.
Lim B, Cheng S. Organelle transcriptomes in plants. Transcriptomics: Open Access. 2013;02(01). https://doi.org/10.4172/2329-8936.1000e106.
Liu Y, Su W, Wang L, Lei J, Chai S, Zhang W, et al. Integrated transcriptome, small RNA and degradome sequencing approaches proffer insights into chlorogenic acid biosynthesis in leafy sweet potato. Plos One. 2021;16(1):e0245266. https://doi.org/10.1371/journal.pone.0245266.
Organelle genome sequencing of morning glories raw sequence reads. NCBI BioProject PRJNA737311. (2021). https://www.ncbi.nlm.nih.gov/bioproject/PRJNA737311
Feng Y. Convolvulaceae-mitogenome-project. (2021). GitHub https://github.com/fengyanlei33/Convolvulaceae-mitogenome-project
Acknowledgements
We gratefully acknowledge Liansheng Xu and Pengwei Li (South China Botanical Garden, The Chinese Academy of Sciences) and Chang An (Guangxi University) for helping collect materials and providing photographs.
Funding
This work was supported by grants from the National Natural Science Foundation of China (32100168), the Chinese State Key Laboratory of Subtropical Silviculture (KF202001), the National Natural Science Foundation of China (31970225), the Leading Innovative and Entrepreneur Team Introduction Program of Zhejiang (2019R01002), the Natural Science Foundation of Fujian Province of China (2021J05061), and the Scientific Research Foundation for the High-level Talents, Fujian University of Traditional Chinese Medicine (X2020012-talents).
Author information
Affiliations
Contributions
Y.F. designed the project. Y.L. and P.L. collected the materials and did the sequencing. Y.L., P.L., Y.Z., D.A., R.P., Z.F., M.H., X.L., and Y.F. performed the research and wrote the manuscript. The authors read and approved the final manuscript.
Corresponding author
Ethics declarations
Ethics approval and consent to participate
Not applicable.
Consent for publication
Not applicable.
Competing interests
The authors declare that they have no competing interests.
Additional information
Publisher’s Note
Springer Nature remains neutral with regard to jurisdictional claims in published maps and institutional affiliations.
Supplementary Information
Additional file 1.
Plastome content. The black, grey and blank cells represent intact, pseudo and missing, respectively. Coding genes with disrupted reading frames, premature stop codons, or non-triplet frameshifts were annotated as pseudogenes.
Additional file 2.
Mitogenome content. The black, grey and blank cells represent intact, pseudo and missing, respectively. Coding genes with disrupted reading frames, premature stop codons, or non-triplet frameshifts were annotated as pseudogenes.
Additional file 3.
Most mitochondrial genes in dodders.
Additional file 4.
BLASTN results of Cu. japonica genus- and species-specific sequences.
Additional file 5.
Plastid CDS, Mitochondrial CDS, and Nuclear 45S.
Additional file 6
Similar situation observed for HSP60.
Additional file 7.
BLASTN searches of the downstream of FR1 and upstream of FR2. Erycibe was used as the query. Parameter: word_size 16, e-value 1e-5.
Additional file 8.
Collections of materials.
Additional file 9.
Data used for organelle genome assemblies.
Additional file 10.
Primers used to confirm gene fission and expansion.
Rights and permissions
Open Access This article is licensed under a Creative Commons Attribution 4.0 International License, which permits use, sharing, adaptation, distribution and reproduction in any medium or format, as long as you give appropriate credit to the original author(s) and the source, provide a link to the Creative Commons licence, and indicate if changes were made. The images or other third party material in this article are included in the article's Creative Commons licence, unless indicated otherwise in a credit line to the material. If material is not included in the article's Creative Commons licence and your intended use is not permitted by statutory regulation or exceeds the permitted use, you will need to obtain permission directly from the copyright holder. To view a copy of this licence, visit http://creativecommons.org/licenses/by/4.0/. The Creative Commons Public Domain Dedication waiver (http://creativecommons.org/publicdomain/zero/1.0/) applies to the data made available in this article, unless otherwise stated in a credit line to the data.
About this article
Cite this article
Lin, Y., Li, P., Zhang, Y. et al. Unprecedented organelle genomic variations in morning glories reveal independent evolutionary scenarios of parasitic plants and the diversification of plant mitochondrial complexes. BMC Biol 20, 49 (2022). https://doi.org/10.1186/s12915-022-01250-1
Received
Accepted
Published
DOIhttps://doi.org/10.1186/s12915-022-01250-1
Keywords
- Convolvulaceae
- Cuscuta
- Plastid genome
- Mitochondrial genome
- ccmFc
- Horizontal gene transfer



Nenhum comentário:
Postar um comentário
Observação: somente um membro deste blog pode postar um comentário.