O papel da seleção na evolução dos mitogenomas das tartarugas marinhas
Abstrato
As tartarugas marinhas são os únicos representantes de quelônios existentes que habitam o ambiente marinho. Uma chave para o sucesso da colonização deste habitat é a adaptação a diferentes demandas energéticas. Tal exigência energética está intrinsecamente relacionada à capacidade mitocondrial de gerar energia através do processo de fosforilação oxidativa (OXPHOS). Aqui, estimamos as relações filogenéticas de Testudines a partir de 90 genomas mitocondriais completos de quelônios e testamos a evolução adaptativa de 13 genes codificadores de proteínas mitocondriais de tartarugas marinhas para determinar como a seleção natural moldou os genes mitocondriais do clado Chelonioidea. Mitogenomas completos mostraram forte suporte e resolução, diferindo na posição do clado Chelonioidea em comparação com a filogenia de tartaruga baseada em dados genômicos nucleares. Os modelos de códons recuperaram um aumento relativamentedN/dS (ω) em três genes OXPHOS para linhagens de tartarugas marinhas. Além disso, encontramos evidências de seleção positiva em pelo menos três posições de códon, codificadas pelos genes da NADH desidrogenase ( ND4 e ND5 ). As taxas evolutivas aceleradas encontradas para tartarugas marinhas em COX2 , ND1 e CYTB e as pegadas moleculares de seleção positiva encontradas nos genes ND4 e ND5 podem estar relacionadas à adaptação molecular mitocondrial ao estresse, provavelmente resultado de um estilo de vida mais ativo em tartarugas marinhas. Nosso estudo fornece informações sobre a evolução adaptativa do genoma mtDNA em tartarugas marinhas e suas implicações para o mecanismo molecular da fosforilação oxidativa.
Introdução
Como diferentes condições ambientais moldam a evolução do DNA mitocondrial tornou-se uma questão comum no campo da biologia evolutiva, revelando mais sobre padrões adaptativos em organismos que enfrentam mudanças ecológicas do que o esperado. A mitocôndria é um componente celular diretamente envolvido no uso de oxigênio, metabolismo e produção de energia e, portanto, desempenha um papel importante na respiração aeróbica através da fosforilação oxidativa (OXPHOS) 1 . O mitogenoma é composto por 13 genes que codificam proteínas OXPHOS, 2 rRNAs (12S rRNA e 16S rRNA) e 22 tRNAs 2 , 3 , e acredita-se que evolua sob seleção purificadora contínua para regiões codificantes 4 , 5. No entanto, como os genes mitocondriais respondem a mudanças nos requisitos de energia, ambientes extremos podem favorecer a seleção positiva, conduzindo adaptações em diferentes genes do mitogenoma para algumas linhagens 5 , 6 , 7 , 8 .
A seleção nos genes do mtDNA tem sido associada à temperatura ambiental, metabolismo exigente elevado, altitude e disponibilidade de oxigênio em várias espécies 7 , 9 , 10 , 11 , 12 . Entre as condições ambientais extremas, a pesquisa sobre a evolução molecular do mtDNA relacionada à alta altitude tem recebido atenção especial 6 , 13 , 14 , 15 , 16 , principalmente porque a avaliação de pressões seletivas de temperatura ambiental e disponibilidade de oxigênio nas mudanças moleculares do mtDNA pode fornecer informações importantes sobre evolução adaptativa do mitogenoma 14. Os ambientes aquáticos apresentam condições desafiadoras semelhantes (hipóxia e baixas temperaturas) e, consequentemente, também exigem adaptações do mitogenoma em vertebrados, que foram relatadas para orcas 17 , botos 18 e pinguins 11 . Apesar da maioria das pesquisas sobre o papel da seleção do mtDNA na evolução dos organismos endotérmicos, recentemente tem havido um interesse crescente em espécies ectotérmicas que são altamente dependentes das condições ambientais. Por exemplo, Escalona et al. 19 estudaram a evolução mitocondrial em tartarugas softshell revelando sítios positivamente selecionados em genes do complexo I para o clado Trionychidae e Carettochelys insculpta, sugerindo evolução convergente dos genes OXPHOS em resposta a um estilo de vida aquático duradouro em ambas as linhagens.
Os quelônios são um grupo com mais de 300 espécies distribuídas em diversos nichos ecológicos ao redor do mundo, incluindo rios, lagos, florestas, desertos e oceanos 20 . Entre os quelônios, várias linhagens se adaptaram independentemente ao ambiente marinho e apenas os representantes do clado Chelonioidea (tartarugas marinhas existentes) sobreviveram até o presente (ver Evers e Benson 21 ). As tartarugas marinhas compreendem sete espécies existentes agrupadas em duas famílias irmãs, Dermochelyidae (uma espécie) e Cheloniidae (seis espécies) 22 . Essas espécies apresentam adaptações aos desafios ambientais marinhos, como nadadeiras, alta excreção de sal por glândulas lacrimais modificadas 23 , conchas hidrodinâmicas e adaptações cardiorrespiratórias para fornecer O 2aos tecidos durante os mergulhos 24 . Além disso, as tartarugas marinhas possuem um estilo de vida ativo, sendo organismos mergulhadores, altamente migratórios e com altas taxas de fecundidade 25 , 26 . As tartarugas marinhas estão entre os répteis existentes que se movem mais rapidamente e, uma vez que a demanda de energia para locomoção é considerada o principal determinante da taxa metabólica dos organismos marinhos 27 , espera-se que o estilo de vida ativo das tartarugas marinhas afete suas demandas energéticas, resultando em taxas metabólicas maiores em comparação com outros répteis 25 , 26 , 28. Portanto, é razoável supor que as adaptações de genes mitocondriais podem ter tido um papel importante no sucesso adaptativo de linhagens de tartarugas marinhas no ambiente marinho, deixando pegadas moleculares em seus mitogenomas.
Apesar dos trabalhos anteriores sobre a caracterização de mitogenomas de tartarugas marinhas, utilizados principalmente em abordagens filogenéticas e filogeográficas, apenas análises exploratórias examinaram as potenciais adaptações ao ambiente marinho dessas espécies em nível molecular 29 . Aqui pretendemos investigar os padrões evolutivos dos mitogenomas das tartarugas marinhas e abordar o possível papel da evolução do mtDNA na adaptação às altas demandas energéticas metabólicas dessas espécies. Fornecemos uma análise filogenética abrangente da evolução do mitogenoma em Testudines e investigamos as taxas evolutivas e assinaturas moleculares da seleção natural para todos os 13 genes codificadores de proteínas do mtDNA para tartarugas marinhas, em uma comparação filogenética com linhagens de tartarugas não marinhas e outras tartarugas não aviárias répteis.
Resultados
genomas mtDNA de tartarugas marinhas
A montagem dos 90 mitogenomas produziu comprimentos de mitogenoma completos entre 16.386 e 21.933 pb. Todas as 39 regiões comuns aos mitogenomas de vertebrados foram identificadas, destacando a translocação ND5 na família Platysternidae (Fig. S1 complementar online). Estimativas de diversidade genética de genes codificadores de proteínas (PCGs) recuperaram ATP8 como o gene mais diverso entre todas as espécies (π = 0,156) seguido por genes de NADH desidrogenase com ND6 (π = 0,133) apresentando maior diversidade. Os genes da citocromo c oxidase ( COX1 , COX2 e COX3 ) foram os menos diversos entre os genes do mtDNA (Tabela 1 ).
Reconstrução filogenética
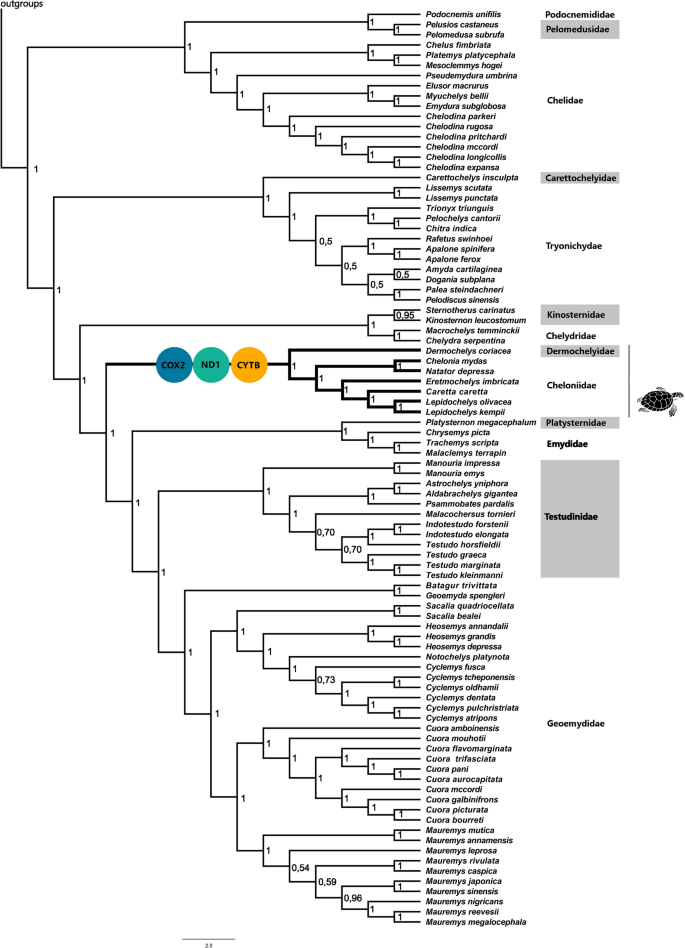
As inferências ML e bayesiana recuperaram topologias altamente semelhantes e bem resolvidas para um alinhamento de mitogenomas completos para as 110 espécies (conjunto de dados I) e um alinhamento dos 13 PCGs concatenados para as 110 espécies (conjunto de dados II) com alto bootstrap (BP > 50% ) e probabilidades posteriores (PP > 90%) suportam a maioria dos ramos (Suplementar Fig. S2 online). Poucas diferenças nas topologias de diferentes conjuntos de dados (I × II) obtidos por diferentes métodos (Bayesian × ML) foram encontradas, com foco nas relações entre algumas espécies das famílias Trionychidae, Testudinidae e Geoemydidae (especialmente o grupo Mauremis ) (Fig. Suplementar S2conectados). Como as abordagens ML e Bayesiana para ambos os conjuntos de dados renderam topologias altamente semelhantes, as análises subsequentes foram realizadas usando a árvore Bayesiana do conjunto de dados II (Fig. 1 ).
Árvore bayesiana (conjunto de dados II) para mitogenomas e topologia de testudines usada para analisar as pressões seletivas em Chelonioidea. Os círculos representam genes mitocondriais com maior dN/dS (ω) para Chelonioidea contra outras linhagens de quelônios identificadas pelo modelo de ramificação no codeML. O número de nós representa probabilidades Bayesianas posteriores.
Análises de seleção
modelo de filial
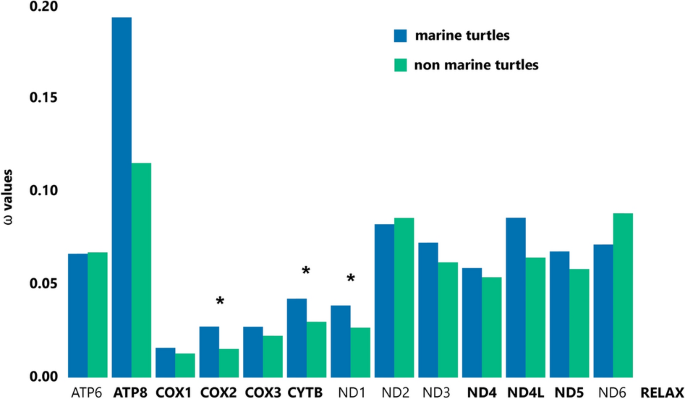
Para testar o papel da seleção no ramo Chelonioidea na árvore filogenética Testudines dos 13 PCGs, aplicamos testes de modelo de ramo codeML. O modelo de razão livre ajustou-se significativamente melhor aos nossos dados do que a hipótese nula (modelo de razão única) para todos os 13 PCGs, sugerindo diferentes taxas evolutivas entre as linhagens incluídas em nosso conjunto de dados. Os valores de dN , dS e ω para cada ramificação estão disponíveis em Dados Suplementares S1 online. O modelo de duas proporções, usado para calcular as pressões seletivas atuando na linhagem de tartarugas marinhas, ajustou-se melhor para os genes COX2 , ND1 e CYTB quando a linhagem de tartarugas marinhas foi rotulada como ramo de primeiro plano (Fig. 2 ). Além disso, o ATP8O gene apresentou os maiores valores de ω, tanto para os ramos foreground quanto background, quando comparado a outros genes. No entanto, o LRT não foi capaz de indicar o modelo de duas razões como o modelo com melhor ajuste para explicar as diferenças nos valores de ω para esse gene ( p = 0,09) (Tabela Suplementar S3 online). Além disso, RELAX detectou seleção intensificada significativa (K > 1) nos genes ATP8, COX1 , COX2 , COX3 , CYTB , ND4 , ND4L e ND5 para a linhagem de tartarugas marinhas (Fig. 2 e Tabela Suplementar S4 online) e nenhum gene foi identificado sob relaxamento.
A força da seleção natural e a proporção de substituições não sinônimas para sinônimas (ω) calculadas com o modelo de duas proporções em codeML para os 13 genes mitocondriais codificadores de proteínas (conjunto de dados III) de marinhos (clado Chelonioidea) e não marinhos (todos os outros clados quelônios). Os genes nos quais o modelo duplo se ajusta melhor aos dados ( p < 0,05) são marcados com um asterisco e foram estimados com o modelo de ramificação de proporção dupla no programa PAML. Os nomes dos genes em negrito representam genes com seleção intensificada significativa em tartarugas marinhas detectadas pelo RELAX ( p < 0,05).
Modelos de filiais
Para calcular as pressões seletivas atuando apenas na linhagem de tartarugas marinhas da filogenia quelônica, realizamos modelos de ramificação usando codeML, FITMODEL e aBSREL. Os testes de branch-site realizados com codeML e FITMODEL detectam sinais significativos de seleção positiva para algumas proporções dos genes testados na linhagem de tartarugas marinhas (Tabela Suplementar S5 online), correspondendo a apenas três sites sob seleção nos genes ND4 e ND5 (Tabela 2). Aplicamos quatro modelos de substituição de códons (M0, M3, M3 + S1 e M3 + S2) no FITMODEL (Guindon et al. 2004). O LRT entre modelos aninhados (M0 x M3; M3 x M3 + S1; M3 + S1 x M3 + S2) sugeriu que o modelo M3 + S2 se ajusta melhor para todos os 13 PCGs, considerando mudanças entre padrões de seleção em locais individuais na filogenia de Testudines ( Tabela Suplementar S6 online). Além disso, este teste indica a ação da seleção positiva no códon 36 do gene ND4 no ramo da tartaruga marinha (Tabela 2 e Figuras complementares. S3 e S4conectados). aBSREL relata dois ramos sob pressão de seleção diversificante episódica no clado Chelonioidea: seleção positiva no ramo ancestral das linhagens Chelonioidea para 4,2% dos sítios no gene ND5 , e seleção positiva na linhagem Eretmochelys imbricata para 24% dos sítios no gene ND6 (Tabela 2 ).
Modelos de sites
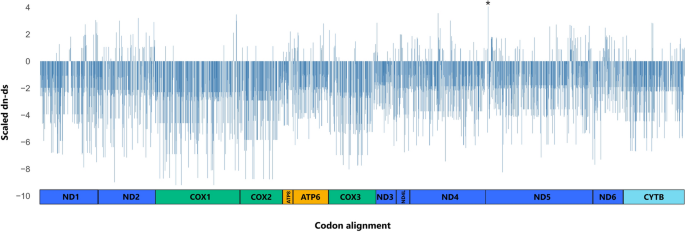
Para avaliar melhor os locais sob seleção dentro de Chelonioidea, realizamos análises de seleção positiva usando modelos de locais em FITMODEL, SLAC, MEME e FUBAR (Tabela 3 ). FITMODEL recuperou três locais selecionados positivamente nos genes ATP6 e ND4 (Tabela 3 ). SLAC retornou muitos códons com altos valores de ω (> 1), mas sem assinaturas significativas de seleção positiva. Nos resultados do SLAC, a terceira posição do gene ND5 se destaca com alto valor de ω (ω = 4,06 ep = 0,085) (Fig. 3 ). MEME revelou seleção positiva diversificada e episódica para ND4 e ND4Lem três posições de códon em linhagens dentro do ramo Chelonioidea (Tabela 3 e Tabela Suplementar S7 online). Como esperado, o teste FUBAR recuperou um padrão geral de seleção purificadora em vários códons para todos os PCGs. ND5 foi o gene com maior número de códons sob seleção purificadora (42,86%) enquanto ND6 com o menor número (10,92%). Apenas duas posições foram atribuídas com seleção positiva diversificada com testes FUBAR: o códon 3 de ND5 (PP = 0,92) e o códon 169 de ND4 (PP = 0,95) (Tabela 3 e Tabela Suplementar S8 online).
Assinaturas de seleção para cada códon para 13 genes mtDNA usando o método SLAC dentro da filogenia Chelonioidea (conjunto de dados IV). Seleção positiva (valores positivos) e seleção negativa (valores negativos) são mostradas. ND6 é transcrito a partir da cadeia de luz. * é a terceira posição do códon de ND5 encontrada sob seleção positiva por FUBAR e MEME.
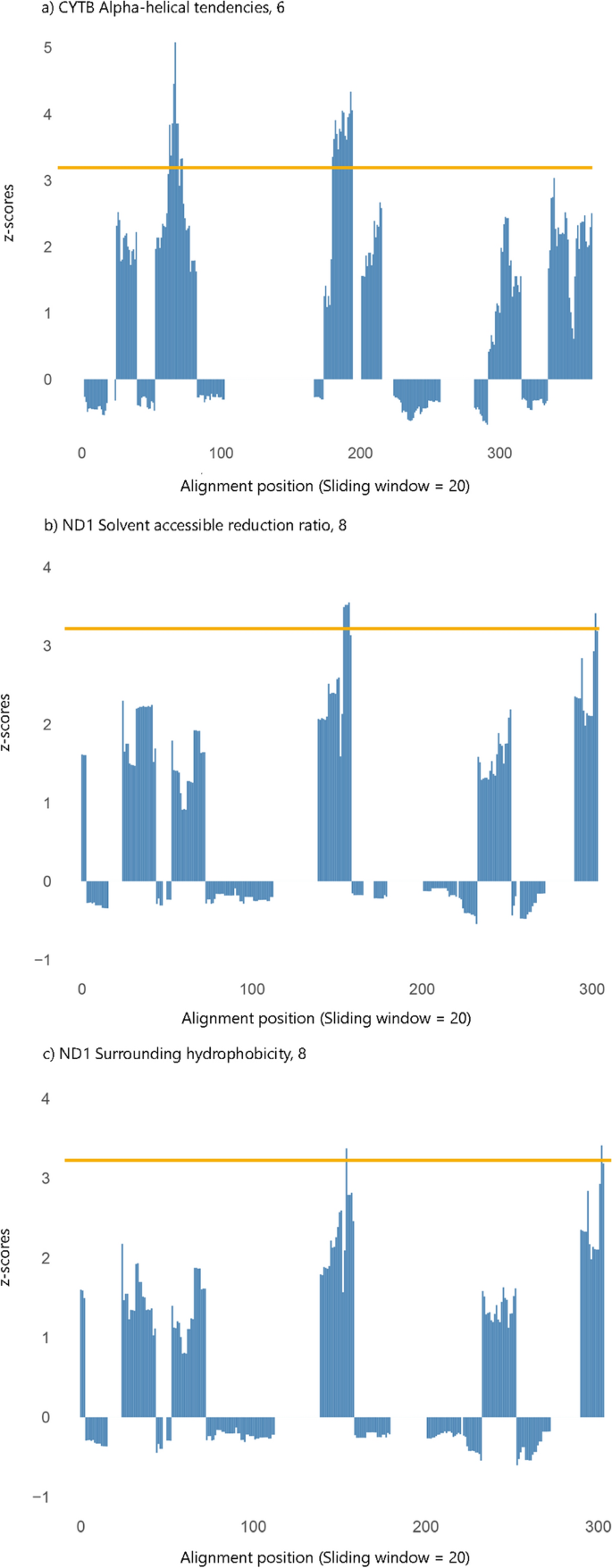
Mudanças nas propriedades físico-químicas de aminoácidos causadas por substituições ao longo da filogenia estimadas usando TreeSAAP sugeriram uma prevalência de seleção purificadora para os três genes com uma diferença significativa na taxa de ω entre o clado Chelonioidea e outros ramos de quelônios identificados no teste de razão de dois modelos com codeML . No entanto, encontramos evidências de seleção positiva em quatro propriedades físico-químicas, com escores z globais > 3,09 ( p < 0,001) para o gene ND1 [taxa de redução acessível ao solvente; Hidrofobicidade circundante; Poder estar no meio da alfa-hélice e constante de equilíbrio (ionização de COOH)] e em uma propriedade físico-química para o gene CYTB (tendências alfa-hélice) (Fig. 4). A análise funcional revela que a posição do 36º códon selecionada positivamente está localizada dentro do domínio catalítico para a atividade da oxidoredutase do gene da subunidade ND4 (Fig. S3 complementar online).
Detecção de alterações físico-químicas significativas de aminoácidos usando TreeSAAP na filogenia Chelonioidea (conjunto de dados IV). Esta análise foi realizada nos genes que apresentam maiores valores de ω identificados pela análise codeML no clado Chelonioidea. Regiões acima do z-score de 3,09 (linha amarela) foram significativamente diferentes do que o assumido sob neutralidade. A respectiva propriedade e categoria são mostradas acima dos gráficos.
Discussão
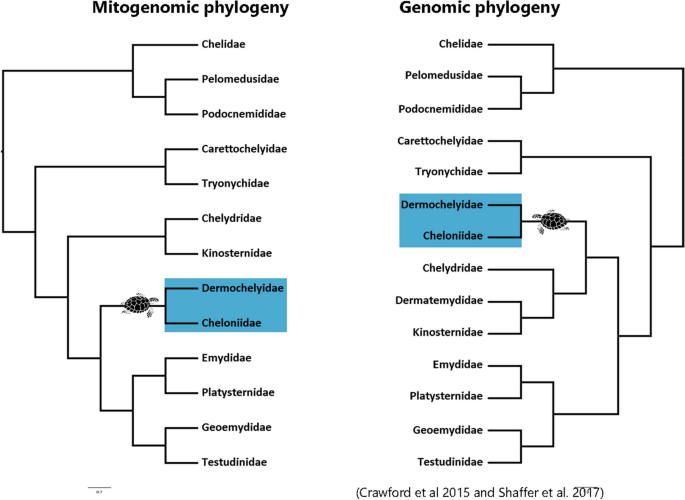
Neste estudo apresentamos a análise mais abrangente da evolução do mitogenoma em Testudines, incluindo dados para 90 espécies, com foco na evolução molecular dos genes OXPHOS em tartarugas marinhas. Como os dados que compilamos são derivados de fontes disponíveis publicamente, nenhum representante da família Dermatemydidae foi incluído nas análises filogenéticas. Assim, a geração do mitogenoma de Dermatemys mawii é importante para alcançar uma filogenia mitocondrial completa. Além da falta desse mitogenoma, as relações filogenéticas que recuperamos eram congruentes com uma filogenia recente de quelônios baseada em 57 genomas mitocondriais completos de tartarugas 19mas com incongruências quanto às posições da linhagem Chelydridae + Dermatemyidae + Kinosternidae e da linhagem das tartarugas marinhas (Cheloniidae + Dermochelyidae) quando comparadas com filogenias recentes usando genes nucleares 30 , 31 (Fig. 5 ). Várias razões podem explicar as divergências nas relações de linhagem nas filogenias de mtDNA e nuDNA: (1) classificação de linhagem incompleta, mais comum para nuDNA do que para mtDNA devido ao menor tamanho efetivo da população do genoma do mtDNA 4 , 32 , 33 (2) disparidades com viés de sexo na dispersão e na suscetibilidade de introgressão diferencial de alelos mitocondriais versus nucleares, resultando em captura mitocondrial 33 , 34 ,35 e (3) seleção, causando convergência de traços ou diversificando sítios adaptativos 7 , 36 , 37 . A discordância entre árvores recuperadas de mitogenomas e genes nucleares já foi relatada para outros grupos de animais. Por exemplo, Li et al. 38 recuperaram diferentes topologias usando marcadores mtDNA e nuDNA na filogenia felina e se concentraram na segunda hipótese para explicar essa diferença, em que a mistura histórica e a captura mitocondrial podem ter ocorrido entre linhagens de ancestrais felinos que tiveram suas divergências há cerca de 11 milhões de anos (MYA) 38 , 39. Aqui encontramos assinatura generalizada de seleção purificadora em mitogenomas quelônios, consistente com a afirmação anterior de que a seleção purificadora atua restringindo a evolução do mitogenoma para conservar a funcionalidade das proteínas OXPHOS 40mas também encontramos evidências de seleção positiva nos genes OXPHOS, reveladas pelo teste baseado em códons ao comparar linhagens de tartarugas marinhas com outras linhagens de quelônios. Além disso, alguns genes OXPHOS parecem estar sob seleção intensificada com base nos resultados RELAX (K > 1) e nos resultados codeML (ω maior em relação a outros genes devido a restrições evolutivas menos conservadoras), sugerindo que em tartarugas marinhas, esses genes experimentaram uma aceleração sobre sua evolução e essa seleção pode ser um fator que poderia explicar a discordância entre as árvores genéticas mitocondriais e nucleares. No entanto, não podemos descartar possível introgressão antiga e classificação incompleta de linhagem (ILS) como fontes plausíveis para essa divergência. Embora os efeitos de hibridização e ILS sejam mais fortes no caso de grupos com tempos de divergência consideravelmente recentes33 , processos microevolutivos também podem impactar divergências profundas 41 como a divergência da linhagem ancestral de tartarugas marinhas de seu grupo irmão não marinho 41 , que data de cerca de 66 MYA (± 30 MYA, 95% HPD) 30 , 31 , 42 . Além disso, a captura mitocondrial antiga já foi sugerida como uma explicação para topologias conflitantes das árvores nucleares e mitocondriais bem suportadas para outros grupos de tartarugas 43 , 44 , 45 e o uso de sequenciamento de espécimes históricos pode ser eficaz para destacar esse problema para o mar tartarugas.
A análise PAML mostrou maior ω em três genes OXPHOS ( COX2 , CYTB e ND1 ) especificamente para o clado das tartarugas marinhas em relação a outras espécies de quelônios, embora não haja evidência estatisticamente significativa para a seleção positiva atuando em uma proporção maior desses genes. Além disso, nossa análise mostrou uma taxa de evolução mais rápida para todas as tartarugas para o gene ATP8, quando comparada aos outros genes, evidenciada pela análise da diversidade genética e pelos altos valores estimados de ω. Os resultados do PAML, juntamente com os sinais de intensificação da seleção indicados pelos testes RELAX, sugerem que as linhagens marinhas acumularam um número maior de alterações de aminoácidos não sinônimos do que as outras linhagens de testudines nesses genes.
A utilização de métodos e pressupostos distintos permitiu-nos explorar melhor os padrões de evolução molecular nas tartarugas marinhas. Juntos, todos os testes de seleção foram capazes de identificar 5 sítios evoluindo sob seleção positiva em quatro genes ( ATP6 , ND4 , ND4L e ND5 ) (Tabelas 2 , 3 ). Além disso, a análise aBSREL encontrou evidências de seleção diversificada episódica em 4,2% dos locais no ramo ancestral do clado Chelonioidea. No entanto, apenas os sites fortemente suportados, identificados por mais de um teste de seleção, foram considerados sob seleção positiva real. Esses sites são 169 e 181 do gene ND4 e 3 do ND5gene e foram detectados em linhagens dentro do clado Chelonioidea. Uma exceção que merece ser mencionada é o 36º sítio do gene ND4 , que foi identificado como evoluindo sob seleção positiva pelo FITMODEL e o único sítio positivamente selecionado com substituição de aminoácidos compartilhada por todas as espécies de tartarugas marinhas. Além disso, este sítio está localizado dentro de uma região funcional baseada na organização do domínio da subunidade ND4 da NADH desidrogenase (ubiquinona), sugerindo um impacto fisiológico neste gene em tartarugas marinhas.
Os três genes que mostraram evidências de evolução acelerada codificam subunidades de três complexos separados da cadeia de transporte de elétrons ( COX2 —complexo IV, CYTB —complexo III e ND1 —complexo I). Esse resultado revela que esses genes têm um excesso de substituições não sinônimas do que o esperado para a linhagem de tartarugas marinhas, diferenciando a taxa de evolução desses genes para a linhagem de tartarugas marinhas do restante das tartarugas. Muitos estudos investigaram o papel da seleção em genes mitocondriais em organismos que habitam grandes altitudes 6 , 10 , 14 , 15 , 16, mas poucos se concentraram em organismos com um estilo de vida mais ativo. Por exemplo, estudos comparando espécies voadoras e não voadoras em grupos de morcegos 46 e gafanhotos 13 encontraram sete genes positivamente selecionados ( ATP8 , COX3 , ND2 , ND4 , ND4L , ND5 e ND6 ) relacionados ao voo em gafanhotos, enquanto ND2 , ND3 , ND4L , ND4 , ND5 , ND6 e COX2 apresentaram valores mais altos de ω em morcegos 46. Nossos resultados, combinados com esses estudos, sugerem que genes complexos I e IV podem ser importantes candidatos a serem impactados pela ação da seleção positiva relacionada a um estilo de vida mais ativo. Por outro lado, como em outros estudos, os genes COX1 e COX3 apresentaram os menores valores de ω e os maiores sinais de seleção purificadora 7 , 47 .
A análise TreeSaap desses genes indicou que regiões com substituições não sinônimas correspondem a mudanças radicais de aminoácidos, embora não mostrando sinais significativos de seleção positiva. Essas mudanças radicais ocorrem quando o resíduo alterado não compartilha propriedades físico-químicas semelhantes com o resíduo ancestral indicando impacto funcional significativo na proteína 48 . Vários estudos correlacionaram a variação nas propriedades dos aminoácidos nas regiões de codificação mitogenômica em várias espécies com (a) requisitos metabólicos mais especializados, como elefantes e seu grande tamanho corporal 49 , dugongos, preguiças e pangolins e sua dieta de baixa energia 50 , (b) uma maior tolerância na faixa térmica em cetáceos e pinguins 11 , 17, (c) voando em morcegos 46 , e (d) vivendo em grandes altitudes em aves galiniformes 16 e faisões alpinos 15 . Comparado a esses estudos, o mitogenoma das tartarugas marinhas mostrou menos evidências da ação penetrante da seleção natural. Este padrão era esperado porque, apesar da profunda divergência entre as espécies marinhas (pelo menos 30 MYA) 29 , estudos anteriores suportam uma razão evolutiva mais lenta e um relógio molecular mais lento para o mitogenoma quelônio 51 , 52 . Além disso, as tartarugas marinhas, como répteis ectotérmicos, são consideradas de baixa taxa metabólica, usando suas reservas energéticas mais lentamente do que as espécies endotérmicas 53. Além disso, essas fracas habilidades termogênicas geralmente confinam as tartarugas marinhas, exceto D. coriacea , a águas tropicais rasas 53 , reduzindo a pressão seletiva da mudança de temperatura, uma das principais hipóteses utilizadas para explicar seus padrões de evolução do genoma mitocondrial 11 . Essas características podem explicar as poucas pegadas moleculares adaptativas neste sistema quelônio em comparação com as inúmeras mudanças moleculares já descritas para organismos endotérmicos.
Os genes OXPHOS compreendem numerosas subunidades que são codificadas tanto pelo mitogenoma quanto pelo genoma nuclear 54 , 55 . Assim, pesquisas considerando seus componentes nucleares podem contribuir para o entendimento completo da evolução dos genes OXPHOS em tartarugas marinhas 56 , 57 , 58 , 59 . Neste cenário, investigar todo o sistema OXPHOS em tartarugas é importante principalmente devido à alta incidência de hibridização entre as espécies de tartarugas marinhas 60 , 61 , 62 , 63 , 64 , 65 , 66 , 67. A compatibilidade entre os componentes nucleares e mitocondriais do sistema OXPHOS é de extrema importância para a aptidão metabólica e energética ideal 54 , 55 , e o impacto desse fenômeno pode ser investigado em tartarugas híbridas. Além disso, abordagens integrando estudos de genética populacional com experimentos bioquímicos e fisiológicos podem representar um próximo passo 57 para entender a evolução do sistema OXPHOS nesses organismos.
Conclusão
Em resumo, aqui investigamos padrões evolutivos e pegadas de seleção em genes OXPHOS de tartarugas marinhas sob a hipótese de que o estilo de vida mais ativo das tartarugas marinhas poderia estar exercendo maior pressão seletiva sobre esses genes. Encontramos evidências de seleção positiva no nível de codificação para vários locais nos genes ND4 e ND5 para diferentes espécies de tartarugas marinhas, destacando um local dentro de um domínio funcional do ND4gene com sinal de seleção compartilhado por todas as espécies do clado Chelonioidea. Embora o estilo de vida ativo das tartarugas marinhas não pareça exercer forte pressão seletiva sobre os genes mitocondriais do sistema OXPHOS, os poucos genes com maiores valores de ω em comparação com outros quelônios e a maior fixação de mutações não sinônimas nesses genes encontrados para o A linhagem de tartarugas marinhas pode ser responsável pelas incongruências entre as topologias inferidas por marcadores mitocondriais ou nucleares. Nossos resultados enfatizam a importância do uso de diferentes análises ao avaliar a seleção no nível do mitogenoma. Além disso, nosso estudo fornece os primeiros insights sobre a evolução adaptativa do genoma mtDNA em tartarugas marinhas, o que pode ter facilitado a radiação bem-sucedida e a diversificação de espécies de tartarugas no ambiente marinho.
material e métodos
conjunto de dados
Recuperamos todas as sequências do genoma mtDNA de 90 tartarugas do banco de dados NCBI, compreendendo 13 das 14 famílias Testudines (Tabela Suplementar S1 online). Apenas a família Dermatemyididae não foi incluída em nossas análises devido à ausência do mitogenoma de Dermatemys mawii em bancos de dados públicos, a única espécie viva desta família. Também recuperamos 20 mitogenomas de répteis que abrangem as ordens Squamata, Aves e Crocodylia para serem usados como grupos externos (Tabela Suplementar S1 online). Geramos vários alinhamentos de sequência usando MAFFT versão 7 68 , 69 . As sequências de nucleotídeos foram primeiro alinhadas, traduzidas em aminoácidos, alinhadas novamente e depois convertidas em um alinhamento de códons, usando a ferramenta PAL2NAL 70e inspecionados manualmente. Editamos manualmente essas sequências para preservar o quadro de leitura esperado (quadro 0) antes do alinhamento, pois algumas espécies com inserções e/ou deleções (indels) de deslocamento de quadro de 1 ou 2 pb no gene ND3 foram observadas por 71 . Devido a uma extensa translocação do agrupamento gênico trnH/trnS1/trnL1/nad5 em Platysternon megacephalum 72 e os sítios comuns de sobreposição entre os genes ATP8 e ATP6 encontrados no mitogenoma 19 de tartarugas , extraímos genes individuais e regiões não codificantes para todas as espécies, com base nas anotações do genoma no GenBank e nos alinhamentos de sequência para manter o tamanho correto dos genes e inferir corretamente os modelos de substituição. Todas as sequências paraND6 foram complementados reversamente devido à sua codificação pela fita reversa do mitogenoma. Essas regiões foram alinhadas individualmente e depois concatenadas, resultando em 39 partições. Quatro conjuntos de dados diferentes foram usados em diferentes etapas analíticas: (I) mitogenoma completo para 110 espécies, (II) apenas genes codificadores de proteínas (PCGs) alinhados separadamente e depois concatenados para 110 espécies, (III) PCGs alinhados individualmente para 110 espécies e (IV) PCGs alinhados individualmente apenas para as sete espécies marinhas.
Para explorar os padrões de diversidade nos genes codificadores de proteínas do mtDNA para tartarugas marinhas, estimamos o número de sítios polimórficos (S) e a diversidade de nucleotídeos (π) usando o DNAsp v. 6.12.03 73 para todos os 13 PCGs mitocondriais.
Reconstrução filogenética
Usamos o software IQ-TREE v. 1.6.8 74 para reconstruir as árvores de máxima verossimilhança (ML) para todos os conjuntos de dados, com 1.000 réplicas de bootstrap ultrarrápidas para avaliar o suporte nodal 75 . Apenas nós com valores de suporte ≥ 80 foram considerados robustos. Também estimamos árvores bayesianas para os conjuntos de dados I e II usando MRBAYES v. 3.2.6 76 , aplicando os modelos particionados estimados com PARTITION FINDER v. 2.1.1 77 (Tabela Suplementar S2 online), de acordo com o Bayesian Information Criterion (BIC). A cadeia de Markov Monte Carlo (MCMC) foi executada por 5.000.000 gerações com quatro cadeias, e as árvores foram amostradas a cada 100 gerações. A convergência de parâmetros foi avaliada usando TRACER v. 1.7.1 78, após excluir 10% iniciais para cada execução. Árvores filogenéticas foram construídas usando os conjuntos de dados I e II para comparar a eficiência da presença de regiões não codificantes no conjunto de dados I na resolução da topologia nas abordagens Bayesiana e ML para ambos os conjuntos de dados.
Análises de seleção
Para testar um possível efeito da topologia na inferência dos sítios sob seleção, também realizamos as análises de seleção usando a topologia inferida com dados genômicos. Como não houve diferença, são apresentados apenas os resultados com a topologia inferida com os dados mitocondriais. Realizamos análises de seleção usando um alinhamento dos PCGs alinhados para as 110 espécies (conjunto de dados III) e um alinhamento dos PCGs alinhados apenas para os alinhamentos de 7 espécies do clado Chelonioidea (conjunto de dados IV) após a conversão em alinhamentos de códons no programa PAL2NAL 70 . Para explorar os padrões de seleção natural e identificar os locais visados pela seleção positiva em cada gene de codificação mitocondrial para a linhagem de tartarugas marinhas, exploramos a variação na proporção ⍵ ( dN / dS , ondedN é a taxa de substituições não sinônimas e dS são as substituições sinônimas), em uma estrutura Bayesiana usando FUBAR do pacote HyPhy v. 2.1 79 , uma estrutura ML usando o programa codeML do PAML v. 4.9 h pacote 80 , FITMODEL v. 0.5. 3 software 81 , MEME e RELAX do pacote HyPhy v. 2.1 82 e em uma abordagem conjunta de ML e métodos de contagem em SLAC, também do pacote HyPhy 83 . Ver Spielman et al. 84 para uma comparação detalhada dos métodos de HyPhy.
Análise do modelo de filial
Para testar se o ω nas tartarugas marinhas era diferente do restante da árvore, usamos modelos de ramificação codeML no conjunto de dados III, o que permite que o ω varie entre as ramificações na filogenia 85 . codeML agrupa vários modelos diferentes, que variam em termos de suas suposições sobre como ω varia entre ramificações da filogenia (modelos de ramificação), através da sequência (modelos de local) e entre ambos (modelo de local de ramificação) 86. Para modelos de ramificação, primeiro estimamos um valor único de ω para todas as ramificações ao longo da árvore com o modelo de uma razão. Então, usando o modelo de razão livre, assumimos um ω independente para cada ramo. Por fim, estimamos um ω para a linhagem de tartarugas marinhas e outro para o restante da filogenia aplicando um modelo de duas proporções. Rotulamos a superfamília Chelonioidea (linhagem de tartarugas marinhas) como o ramo de primeiro plano em cada árvore filogenética gerada para cada PCG em análises separadas, para o modelo de duas proporções e todos os modelos de local de ramificação descritos abaixo. O clado alvo para essas análises é representado em negrito na Fig. 1(clado Chelonioidea). Todos os ramos restantes, que incluem tartarugas terrestres e de água doce, não foram marcados, sendo considerados pelo algoritmo como ramos de fundo. O mesmo esquema de rótulo de ramificação foi usado em RELAX (conjunto de dados III) para inferir se a força de seleção foi intensificada (K > 1) ou relaxada (K < 1) na superfamília Chelonioidea.
Análise de modelos de filiais
Modelos de locais de ramificação foram usados para determinar se alguma proporção de locais está sujeita à seleção positiva ao longo da linhagem Chelonioidea. Portanto, para análise de local de ramificação em codeML e FITMODEL, também dividimos a árvore inferida do conjunto de dados III em ramo de primeiro plano (clado Chelonioidea), onde os locais podem estar evoluindo sob seleção positiva e ramos de fundo (todas as linhagens restantes ou tartarugas não marinhas) , onde a seleção positiva está ausente 80 , 86 , 87 . No codeML, usamos o modelo A versus o modelo nulo. FITMODEL é adequado para análise exploratória e testamos se nossos dados se ajustavam aos modelos de substituição de códons aninhados M0 e M3 80 , 86 , 87. Enquanto o modelo M0 assume que todos os locais em um alinhamento de sequência estão sujeitos ao mesmo processo de seleção, o modelo M3 assume a variação na restrição seletiva entre os locais e é modelado como três classes de razão de taxas com ω 1 , ω 2 e ω 3 . O FITMODEL permite comutações site-specific entre diferentes valores da razão de taxas não sinônimas/sinônimas 81 e testamos os modelos de comutação M3 + S1 e M3 + S2. Sob o teste de comutação no FITMODEL, um processo de Markov reversível no tempo com três parâmetros adicionais é modelado: a taxa geral de intercâmbio entre as classes de razão de taxa (δ), um coeficiente para deslocamentos entre ω 1 e ω 3 (α) e um coeficiente para mudanças entre ω 2e ω 3 (β). O modelo S1 impõe taxas de troca iguais entre classes de razão de taxas ω 1 , ω 2 e ω 3 (α = β = 1), enquanto o modelo S2 permite que α e β variem livremente levando em consideração taxas desiguais de trocas entre classes de seleção 81 . Por fim, aplicamos um terceiro teste de local de filial usando aBSREL (“probabilidade de efeitos aleatórios adaptativos de local de filial”) do HyPhy 88usando o conjunto de dados III. Diferentemente de codeML e FITMODEL, as taxas são calculadas para cada ramo em aBSREL, permitindo a seleção positiva em ramos de fundo. Para evitar sobreparametrização, o aBSREL infere, usando a correção do Critério de Informação de Akaike (AICc) de amostra pequena, o número ideal de categorias de tarifas por ramal, em vez de assumir que cada ramal deva ser equipado com três classes de tarifas. Além disso, os valores de p obtidos a partir de testes individuais para comparações múltiplas foram corrigidos por aBSREL usando o procedimento de Bonferroni-Holm para controlar as taxas de falso-positivo familiar 84 , 88 .
Site-modelos
Também aplicamos análise de modelo de sítio dentro da filogenia de Chelonioidea usando o conjunto de dados IV. Usamos Single-Likelihood Ancestor Counting (SLAC) 89 para obter uma visão geral das 13 assinaturas de seleção de PCGs para tartarugas marinhas. O SLAC infere as taxas de dN e dS por local usando uma combinação de ML e abordagens de contagem. Usamos um modelo de evolução de efeitos mistos (MEME) 90detectar sítios evoluindo sob seleção positiva dentro do ramo Chelonioidea. Semelhante ao FITMODEL, o MEME aplica uma estrutura filogenética de efeitos aleatórios de local de ramificação, permitindo que a distribuição de ω varie de local para local, bem como de ramo para ramo, o que permite ao MEME identificar instâncias de seleção positiva episódica e generalizada. Também usamos o software FUBAR para estimar o número de substituições não sinônimas e sinônimas em cada códon em 13 PCGs para filogenia de tartarugas marinhas, fornecendo a probabilidade posterior de cada códon pertencer a um conjunto de classes de ω (ω = 1, ω < 1 ou ω > 1) 91 . A significância foi avaliada pela probabilidade posterior (PP) > 0,95.
Genes detectados com maiores valores de ω em testes codeML foram então analisados em TreeSAAP v. 3.2 92 dentro da filogenia Chelonioidea (dataset IV). O TreeSAAP conta com o modelo MM01 implementado em baseML do pacote PAML 85 e usa uma filogenia para reconstruir os estados ancestrais mais prováveis para as sequências genéticas sob investigação. Este software atribui valores de peso às alterações de códons não sinônimos, para os quais os efeitos físico-químicos gerais são avaliados usando um modelo com 31 propriedades físico-químicas de aminoácidos. A magnitude da mudança é classificada de 1 (mais conservadora) a 8 (mais radical). Um desvio significativo da evolução neutra é testado por meio de um escore z e interpretado como resultado da seleção positiva. Um escore z altamente significativo calculado no TreeSAAP (z > 3,09,p < 0,01) indica mais substituições não sinônimas do que assumidas no modelo neutro 92 . Para garantir a chamada conservadora de sítios de códons selecionados positivamente, foram usadas apenas alterações de aminoácidos com pontuação entre 6 e 8 e com pontuação z positiva < 0,001 92 . Finalmente, empregamos a análise funcional de PCGs usando o recurso da Web InterPro 76.0 93 , para prever domínios de proteínas e investigar se os locais identificados sob seleção positiva estão presentes dentro ou perto de regiões funcionais.
Referências
Saraste, M. Fosforilação oxidativa no fin de siècle. Science 283 , 1488-1493 (1999).
CASPubMedPUBLICIDADESGoogle ScholarAnderson, S. et al. Sequência e organização do genoma mitocondrial humano. Nature 290 , 457-465 (1981).
CASPubMedPUBLICIDADESGoogle ScholarMelo-Ferreira, J. et al. A natureza indescritível da evolução adaptativa do DNA mitocondrial de uma linhagem ártica propensa a introgressão frequente. Genoma Biol. Evolução 6 , 886-896 (2014).
CASPubMedPubMed CentralGoogle ScholarBallard, JWO & Whitlock, MC A história natural incompleta das mitocôndrias. Mol. Eco. 13 , 729-744 (2004).
PubMedGoogle ScholarPavlova, A. et al. A seleção purificadora e a deriva genética moldaram a evolução do genoma mitocondrial do Pleistoceno em um peixe de água doce australiano ameaçado de extinção. Hereditariedade 118 , 466–476 (2017).
CASPubMedPubMed CentralGoogle ScholarYu, L., Wang, X., Ting, N. & Zhang, Y. Análise mitogenômica de macacos chineses de nariz arrebitado: evidência de seleção positiva em genes NADH desidrogenase em adaptação a grandes altitudes. Mitochondrion 11 , 497-503 (2011).
CASPubMedGoogle ScholarMorales, HE, Pavlova, A., Joseph, L. & Sunnucks, P. Seleção positiva e purificadora em genomas mitocondriais de uma ave com discordância mitonuclear. Mol. Eco. 24 , 2820-2837 (2015).
CASPubMedGoogle ScholarJin, Y. et al. Análise evolutiva de proteínas codificadas mitocondrialmente de lagartos com cabeça de sapo, Phrynocephalus , ao longo de um gradiente altitudinal. BMC Genomics 19 , 185 (2018).
PubMedPubMed CentralCASGoogle ScholarBalloux, F., Handley, L.-JL, Jombart, T., Liu, H. & Manica, A. O clima moldou a distribuição mundial da variação da sequência do DNA mitocondrial humano. Proc. R. Soc. B Biol. ciência 276 , 3447–3455 (2009).
CASGoogle ScholarXu, S. et al. Adaptação de alta altitude e análise filogenética do cavalo tibetano com base no genoma mitocondrial. J. Genet. Genomics 34 , 720-729 (2007).
CASPubMedGoogle ScholarRamos, B. et al. Genômica da paisagem: a seleção natural impulsiona a evolução do mitogenoma em pinguins. BMC Genomics 19 , 53 (2018).
PubMedPubMed CentralCASGoogle ScholarMori, S. & Matsunami, M. Assinatura de seleção positiva no DNA mitocondrial em Cetartiodactyla. Gene Genet. Sist. 93 , 65–73 (2018).
CASPubMedGoogle ScholarLi, X.-D. e outros A seleção positiva conduziu a adaptação dos genes mitocondriais às demandas de vôo e ambientes de alta altitude em gafanhotos. Frente. Genet. 9 , 605 (2018).
CASPubMedPubMed CentralGoogle ScholarLuo, Y., Yang, X. & Gao, Y. Resposta do DNA mitocondrial à alta altitude: uma nova perspectiva sobre a adaptação à alta altitude. DNA mitocondrial 24 , 313–319 (2013).
CASPubMedGoogle ScholarGu, P. et al. Evidência de evolução adaptativa de faisões alpinos ao ambiente de alta altitude da perspectiva mitogenômica. DNA mitocondrial A DNA Mapp. Seq. Anal. 27 , 455-462 (2016).
CASPubMedGoogle ScholarZhou, T., Shen, X., Irwin, DM, Shen, Y. & Zhang, Y. Análises mitogenômicas propõem seleção positiva em genes mitocondriais para adaptação de alta altitude em aves galiformes. Mitochondrion 18 , 70-75 (2014).
CASPubMedGoogle ScholarFoote, AD et ai. Seleção positiva no mitogenoma da baleia assassina. Biol. Deixe 7 , 116–118 (2011).
CASPubMedGoogle ScholarCaballero, S., Duchêne, S., Garavito, MF, Slikas, B. & Baker, CS Evidência inicial de seleção adaptativa na subunidade dois de NADH de golfinhos de água doce por análises de genomas mitocondriais. PLoS ONE 10 , e0123543 (2015).
PubMedPubMed CentralCASGoogle ScholarEscalona, T., Weadick, CJ & Antunes, A. Padrões adaptativos da evolução do mitogenoma estão associados à perda de escamas nas tartarugas. Mol. Biol. Evolução 34 , 2522–2536 (2017).
CASPubMedPubMed CentralGoogle ScholarVitt, LJ & Caldwell, JP Herpetologia: Uma biologia introdutória de anfíbios e répteis (Academic Press, Nova York, 2013).
Evers, SW & Benson, RBJ Uma nova hipótese filogenética de tartarugas com implicações para o tempo e número de transições evolutivas para estilos de vida marinhos no grupo. Paleontologia 62 , 93–134 (2019).
Google ScholarLutz, PL, Musick, JA & Wyneken, J. A Biologia das Tartarugas Marinhas Vol. 2 (CRC Press, Boca Raton, 2002).
Google ScholarSchmidt-Nielsen, K. & Fange, R. glândulas de sal em répteis marinhos. Nature 182 , 783-785 (1958).
PUBLICIDADESGoogle ScholarLutz, PL & Lutcavage, ME Mergulho fisiologia. Em A Biologia das Tartarugas Marinhas Vol. 1 (eds Lutz, PL & Musick, JA) 291–310 (CRC Press, Boca Raton, 2017).
Wallace, BP & Jones, TT O que faz as tartarugas marinhas irem: uma revisão das taxas metabólicas e suas consequências. J. Exp. Mar. Biol. Eco. 356 , 8–24 (2008).
Google ScholarWyneken, J., Lohmann, KJ & Musick, JA A Biologia das Tartarugas Marinhas Vol. 3 (CRC Press, Boca Raton, 2013).
Google ScholarSeibel, BA & Drazen, JC A taxa de metabolismo em animais marinhos: Restrições ambientais, demandas ecológicas e oportunidades energéticas. Philos. Trans. R. Soc. Lond. BBiol. ciência 362 , 2061-2078 (2007).
CASGoogle ScholarDavenport, J., Munks Sarah, A., Oxford, PJ & Fogg, GE Uma comparação da natação de tartarugas marinhas e de água doce. Proc. R. Soc. Lond. Ser. BBiol. ciência 220 , 447-475 (1984).
Google ScholarDuchene, S. et ai. Filogenética e evolução do mitogenoma das tartarugas marinhas. Mol. Filogeneta. Evolução 65 , 241–250 (2012).
PubMedGoogle ScholarCrawford, NG et ai. Uma análise filogenômica de tartarugas. Mol. Filogeneta. Evolução 83 , 250–257 (2015).
PubMedGoogle ScholarShaffer, HB, McCartney-Melstad, E., Near, TJ, Mount, GG & Spinks, PQ Análises filogenômicas de 539 loci altamente informativos datam uma árvore de tempo totalmente resolvida para os principais clados de tartarugas vivas (Testudines). Mol. Filogeneta. Evolução 115 , 7–15 (2017).
PubMedGoogle ScholarPamilo, P. & Nei, M. Relações entre árvores genéticas e árvores de espécies. Mol. Biol. Evolução 5 , 568-583 (1988).
PubMedGoogle ScholarFunk, DJ & Omland, KE Parafilia e polifilia em nível de espécie: frequência, causas e consequências, com percepções do DNA mitocondrial animal. Annu. Rev. Ecol. Evolução Sist. 34 , 397-423 (2003).
Google ScholarSang, T. & Zhong, Y. Testando hipóteses de hibridação baseadas em árvores genéticas incongruentes. Sist. Biol. 49 , 422–434 (2000).
CASPubMedGoogle ScholarRheindt, FE & Edwards, SV Introgressão genética: Um componente integral, mas negligenciado, da especiação em aves. 128 de agosto, 620–632 (2011).
Google ScholarCheviron, ZA & Brumfield, RT Migração-seleção de equilíbrio e adaptação local de haplótipos mitocondriais em pardais rufous-collared ( Zonotrichia capensis ) ao longo de um gradiente de elevação. Evolution 63 , 1593–1605 (2009).
PubMedGoogle ScholarGompert, Z., Forister, ML, Fordyce, JA & Nice, CC Discordância mitonuclear generalizada com evidências de hibridização introgressiva e varreduras seletivas em Lycaeides. Mol. Eco. 17 , 5231–5244 (2008).
CASPubMedGoogle ScholarLi, G., Davis, BW, Eizirik, E. & Murphy, WJ Evidência filogenômica para hibridização antiga nos genomas de gatos vivos (Felidae). Genoma Res. 26 , 1–11 (2016).
PubMedPubMed CentralCASGoogle ScholarJohnson, WE et al. A radiação do Mioceno tardio dos felídeos modernos: uma avaliação genética. Ciência 311 , 73–77 (2006).
CASPubMedPUBLICIDADESGoogle ScholarRand, DM & Kann, LM Excesso de polimorfismo de aminoácidos no DNA mitocondrial: contrastes entre genes de Drosophila, camundongos e humanos. Mol. Biol. Evolução 13 , 735-748 (1996).
CASPubMedGoogle ScholarOliver, JC Processos microevolutivos geram discordância filogenômica em divergências antigas. Evolution 67 , 1823–1830 (2013).
PubMedGoogle ScholarJoyce, WG, Parham, JF, Lyson, TR, Warnock, RCM & Donoghue, PCJ Uma análise de divergência de datação de tartarugas usando calibrações de fósseis: um exemplo de melhores práticas. J. Paleontol. 87 , 612–634 (2013).
Google ScholarKehlmaier, C. et ai. Mitogenômica de espécimes de tipo histórico de tartarugas da Australásia: Esclarecimento da confusão taxonômica e antiga introgressão mitocondrial. ciência Rep. 9 , 1–12 (2019).
CASGoogle ScholarVamberger, M. et ai. A população de tartarugas do Parque Nacional Phnom Kulen (noroeste do Camboja) tem assinaturas genéticas e morfológicas de hibridação. J. Zool. Sist. Evolução Res. 55 , 167–174 (2017).
Google ScholarSpinks, PQ & Shaffer, HB Filogenias mitocondriais e nucleares conflitantes para o complexo de espécies Emys (Testudines: Emydidae) amplamente disjunto, e o que elas nos dizem sobre biogeografia e hibridização. Sist. Biol. 58 , 1–20 (2009).
CASPubMedGoogle ScholarShen, Y.-Y. e outros Evolução adaptativa dos genes do metabolismo energético e a origem do voo em morcegos. Proc. Nacional Acad. ciência EUA 107 , 8666–8671 (2010).
CASPubMedPUBLICIDADESPubMed CentralGoogle ScholarBernardo, PH e cols. Discordância mito-nuclear extrema em um lagarto peninsular: o papel da deriva, seleção e clima. Hereditariedade 123 , 359–370 (2019).
PubMedPubMed CentralGoogle ScholarBetts, MJ & Russell, RB Propriedades de aminoácidos e consequências das substituições. Em Bioinformatics for Geneticists (eds Barnes, MR & Gray, IC) 289–316 (Wiley, Nova York, 2003).
Google ScholarFinch, TM, Zhao, N., Korkin, D., Frederick, KH & Eggert, LS Evidência de seleção positiva em complexos mitocondriais I e V do elefante africano. PLoS ONE 9 , e92587 (2014).
PubMedPubMed CentralPUBLICIDADESCASGoogle Scholarda Fonseca, RR, Johnson, WE, O'Brien, SJ, Ramos, MJ & Antunes, A. A evolução adaptativa do genoma mitocondrial dos mamíferos. BMC Genomics 9 , 119 (2008).
PubMedPubMed CentralCASGoogle ScholarEo, SH & DeWoody, JA As taxas evolutivas dos genomas mitocondriais correspondem às taxas de diversificação e à riqueza de espécies contemporâneas em aves e répteis. Proc. Biol. ciência 277 , 3587–3592 (2010).
PubMedPubMed CentralGoogle ScholarAvise, JC, Bowen, BW, Lamb, T., Meylan, AB & Bermingham, E. Evolução do DNA mitocondrial no ritmo de uma tartaruga: evidências de baixa variabilidade genética e taxa microevolutiva reduzida nos Testudines. Mol. Biol. Evolução 9 , 457–473 (1992).
PubMedGoogle ScholarHays, GC Tartarugas marinhas: uma revisão de algumas das principais descobertas recentes e questões remanescentes. J. Exp. Mar. Bio. Eco. 356 , 1–7 (2008).
Google ScholarHatefi, Y. O transporte de elétrons mitocondrial e sistema de fosforilação oxidativa. Annu. Rev. Biochem. 54 , 1015–1069 (1985).
CASPubMedGoogle ScholarRand, DM, Haney, RA & Fry, AJ Coevolução citonuclear: a genômica da cooperação. Tendências Eco. Evolução 19 , 645-653 (2004).
PubMedGoogle ScholarPopadin, KY, Nikolaev, SI, Junier, T., Baranova, M. & Antonarakis, SE A seleção purificadora em genes de codificação de proteínas mitocondriais de mamíferos é altamente eficaz e congruente com a evolução de genes nucleares. Mol. Biol. Evolução 30 , 347–355 (2013).
CASPubMedGoogle ScholarSunnucks, P., Morales, HE, Lamb, AM, Pavlova, A. & Greening, C. Abordagens integrativas para estudar a coevolução do genoma mitocondrial e nuclear na fosforilação oxidativa. Frente. Genet. 8 , 25 (2017).
PubMedPubMed CentralCASGoogle ScholarLevin, L., Blumberg, A., Barshad, G. & Mishmar, D. Mito-nuclear co-evolução: os lados positivos e negativos de antigas mutações funcionais. Frente. Genet. 5 , 448 (2014).
PubMedPubMed CentralCASGoogle ScholarGershoni, M. et al. A interrupção da coevolução mitocondrial-nuclear afeta a integridade do complexo OXPHOS I e afeta a saúde humana. Genoma Biol. Evolução 6 , 2665-2680 (2014).
CASPubMedPubMed CentralGoogle ScholarJames, MC, Martin, K. & Dutton, PH Hibridização entre uma tartaruga verde, Chelonia mydas , e uma tartaruga cabeçuda, Caretta caretta , e o primeiro registro de uma tartaruga verde no Canadá atlântico. Posso. Campo-Nat. 118 , 579-582 (2004).
Google ScholarKarl, SA, Bowen, BW & Avise, JC Hibridização entre os antigos marinheiros: Caracterização de híbridos de tartarugas marinhas com ensaios genéticos moleculares. J. Hered. 86 , 262–268 (1995).
CASPubMedGoogle ScholarLara-Ruiz, P., Lopez, GG, Santos, FR & Soares, LS Hibridação extensiva em tartarugas-de-pente ( Eretmochelys imbricata ) nidificando no Brasil revelada por análises de mtDNA. Conserva Genet. 7 , 773–781 (2006).
CASGoogle ScholarKelez, S., Velez-Zuazo, X. & Pacheco, AS Primeiro registro de hibridação entre tartarugas verdes Chelonia mydas e tartarugas -de-pente Eretmochelys imbricata no sudeste do Pacífico. PeerJ 4 , e1712 (2016).
PubMedPubMed CentralCASGoogle ScholarSoares, LS e cols. Comparação da produção reprodutiva de tartarugas marinhas híbridas e espécies parentais. Mar. Biol. 164 , 9 (2016).
CASGoogle ScholarReis, EC, Soares, LS & Lôbo-Hajdu, G. Evidências da introgressão do genoma mitocondrial olive ridley em viveiros de tartaruga cabeçuda de Sergipe, Brasil. Conserva Genet. 11 , 1587-1591 (2010).
CASGoogle ScholarBowen, BW & Karl, SA Genética populacional e filogeografia de tartarugas marinhas. Mol. Eco. 16 , 4886–4907 (2007).
CASPubMedGoogle ScholarVilaça, ST, Lara-Ruiz, P., Marcovaldi, MA, Soares, LS & Santos, FR Origem da população e demografia histórica de agregados de alimentação e nidificação de bico-de-pente ( Eretmochelys imbricata ) do Brasil. J. Exp. Mar. Biol. Eco. 446 , 334–344 (2013).
Google ScholarKatoh, K., Misawa, K., Kuma, K.-I. & Miyata, T. MAFFT: Um novo método para alinhamento rápido de múltiplas sequências baseado na transformada rápida de Fourier. Res. de Ácidos Nucleicos. 30 , 3059–3066 (2002).
CASPubMedPubMed CentralGoogle ScholarKatoh, K., Rozewicki, J. & Yamada, KD Serviço online MAFFT: Alinhamento de múltiplas sequências, escolha interativa de sequências e visualização. Apresentação. bioinform. https://doi.org/10.1093/bib/bbx108 (2017).
PubMed CentralGoogle ScholarSuyama, M., Torrents, D. & Bork, P. PAL2NAL: Conversão robusta de alinhamentos de sequências de proteínas nos alinhamentos de códons correspondentes. Res. de Ácidos Nucleicos. 34 , W609–W612 (2006).
CASPubMedPubMed CentralGoogle ScholarRussell, RD & Beckenbach, AT Recodificação da tradução em genomas mitocondriais de tartarugas: mutações programadas de mudança de quadro e evidências de um código genético modificado. J. Mol. Evolução 67 , 682–695 (2008).
CASPubMedPubMed CentralPUBLICIDADESGoogle ScholarLuo, H. et al. O genoma mitocondrial completo de Platysternon megacephalum peguense e análise filogenética molecular. Genes 10 , 487 (2019).
CASPubMed CentralGoogle ScholarRozas, J. et ai. DnaSP 6: Análise de polimorfismo de sequência de DNA de grandes conjuntos de dados. Mol. Biol. Evolução 34 , 3299–3302 (2017).
CASPubMedGoogle ScholarNguyen, L.-T., Schmidt, HA, von Haeseler, A. & Minh, BQ IQ-TREE: Um algoritmo estocástico rápido e eficaz para estimar filogenias de probabilidade máxima. Mol. Biol. Evolução 32 , 268–274 (2015).
CASPubMedGoogle ScholarHoang, DT, Chernomor, O., von Haeseler, A., Minh, BQ & Vinh, LS UFBoot2: Melhorando a aproximação de inicialização ultra-rápida. Mol. Biol. Evolução 35 , 518–522 (2018).
CASPubMedGoogle ScholarRonquist, F. et ai. MrBayes 3.2: Inferência filogenética bayesiana eficiente e escolha de modelo em um grande espaço de modelo. Sist. Biol. 61 , 539–542 (2012).
PubMedPubMed CentralGoogle ScholarLanfear, R., Frandsen, PB, Wright, AM, Senfeld, T. & Calcott, B. PartitionFinder 2: Novos métodos para selecionar modelos particionados de evolução para análises filogenéticas moleculares e morfológicas. Mol. Biol. Evolução 34 , 772-773 (2017).
PubMedGoogle ScholarRambaut, A., Drummond, AJ, Xie, D., Baele, G. & Suchard, MA Sumarização posterior em filogenética bayesiana usando traçador 1.7. Sist. Biol. 67 , 901–904 (2018).
CASPubMedPubMed CentralGoogle ScholarPond, SLK, Frost, SDW & Muse, SV HyPhy: Teste de hipóteses usando filogenias. Bioinformática 21 , 676-679 (2005).
CASPubMedGoogle ScholarYang, Z. PAML 4: Análise filogenética por máxima verossimilhança. Mol. Biol. Evolução 24 , 1586–1591 (2007).
CASPubMedGoogle ScholarGuindon, S., Rodrigo, AG, Dyer, KA & Huelsenbeck, JP Modelando a variação site-specific de padrões de seleção ao longo de linhagens. Proc. Nacional Acad. ciência EUA 101 , 12957–12962 (2004).
CASPubMedPUBLICIDADESPubMed CentralGoogle ScholarWertheim, JO, Murrell, B., Smith, MD, Kosakovsky Pond, SL & Scheffler, K. RELAX: Detectando seleção relaxada em uma estrutura filogenética. Mol. Biol. Evolução 32 , 820–832 (2015).
CASPubMedGoogle ScholarKosakovsky Pond, SL & Frost, SDW Afinal, não é tão diferente: uma comparação de métodos para detectar locais de aminoácidos sob seleção. Mol. Biol. Evolução 22 , 1208-1222 (2005).
PubMedCASGoogle ScholarSpielman, SJ et al. Evolução dos genomas virais: Interação entre seleção, recombinação e outras forças. Métodos Mol. Biol. 1910 , 427–468 (2019).
CASPubMedGoogle ScholarYang, Z. Testes de razão de verossimilhança para detectar seleção positiva e aplicação para evolução de lisozimas de primatas. Mol. Biol. Evolução 15 , 568–573 (1998).
CASPubMedGoogle ScholarYang, Z. & Nielsen, R. Modelos de substituição de códons para detectar a adaptação molecular em locais individuais ao longo de linhagens específicas. Mol. Biol. Evolução 19 , 908–917 (2002).
CASPubMedGoogle ScholarZhang, J., Nielsen, R. & Yang, Z. Avaliação de um método aprimorado de probabilidade de local de ramificação para detectar seleção positiva no nível molecular. Mol. Biol. Evolução 22 , 2472–2479 (2005).
CASPubMedGoogle ScholarSmith, MD et ai. Menos é mais: um modelo adaptativo de efeitos aleatórios de filial para detecção eficiente de seleção diversificada episódica. Mol. Biol. Evolução 32 , 1342-1353 (2015).
CASPubMedPubMed CentralGoogle ScholarPond, SLK & Frost, SDW Datamonkey: Detecção rápida de pressão seletiva em locais individuais de alinhamentos de códons. Bioinformática 21 , 2531–2533 (2005).
CASPubMedGoogle ScholarMurrell, B. et al. Detecção de sítios individuais sujeitos a seleção diversificada episódica. PLoS Genet. 8 , e1002764 (2012).
CASPubMedPubMed CentralGoogle ScholarMurrell, B. et al. FUBAR: Uma aproximação bayesiana rápida e irrestrita para inferir a seleção. Mol. Biol. Evolução 30 , 1196–1205 (2013).
CASPubMedPubMed CentralGoogle ScholarWoolley, S., Johnson, J., Smith, MJ, Crandall, KA & McClellan, DA TreeSAAP: Seleção de propriedades de aminoácidos usando árvores filogenéticas. Bioinformática 19 , 671-672 (2003).
CASPubMedGoogle ScholarMitchell, AL et al. InterPro em 2019: Melhorando a cobertura, classificação e acesso a anotações de sequências de proteínas. Res. de Ácidos Nucleicos. 47 , D351–D360 (2019).
CASPubMedGoogle Scholar
Reconhecimentos
Este estudo foi parcialmente financiado pela Coordenação de Aperfeiçoamento de Pessoal de Nível Superior—Brasil (CAPES)—Código Financeiro 001 e FAPESP (2015/18269-1 e 2018/01236-1). LF foi financiado por bolsa de pós-doutorado FAPESP (2017/25058-2). Agradecemos aos cientistas que disponibilizaram as sequências do mitogenoma utilizadas neste estudo.
Informação sobre o autor
Autores e Afiliações
Contribuições
EKSR e MFN conceberam a hipótese de pesquisa; EKSR e LF analisaram os dados e redigiram o manuscrito; todos os autores contribuíram igualmente na versão final.
autor correspondente
Declarações éticas
Interesses competitivos
Os autores declaram não haver interesses conflitantes.
Informação adicional
Nota do editor
A Springer Nature permanece neutra em relação a reivindicações jurisdicionais em mapas publicados e afiliações institucionais.
Nenhum comentário:
Postar um comentário
Observação: somente um membro deste blog pode postar um comentário.