Neolítico do Noroeste da África iniciado por migrantes da Península Ibérica e do Levante
Natureza 618 , 550–556 ( 2023 )
Resumo
No noroeste da África, o estilo de vida passou da coleta de alimentos para a produção de alimentos há cerca de 7.400 anos, mas o que desencadeou essa mudança permanece obscuro.
Dados arqueológicos apoiam visões conflitantes: (1) que os agricultores neolíticos europeus migrantes trouxeram o novo modo de vida para o norte da África 1 , 2 , 3 ou (2) que os caçadores-coletores locais adotaram inovações tecnológicas 4 , 5 . Esta última visão também é apoiada por dados arqueogenéticos 6 .
Aqui, preenchemos lacunas cronológicas e arqueogenéticas importantes para o Magreb, do Epipaleolítico ao Neolítico Médio, sequenciando os genomas de nove indivíduos (para uma cobertura genômica entre 45,8 e 0,2 vezes maior). Notavelmente, rastreamos 8.000 anos de continuidade populacional e isolamento do Paleolítico Superior, via Epipaleolítico, até alguns grupos agrícolas neolíticos magrebinos. No entanto, vestígios dos primeiros contextos neolíticos mostraram principalmente ancestralidade neolítica europeia. Sugerimos que a agricultura foi introduzida por migrantes europeus e foi então rapidamente adotada por grupos locais. Durante o Neolítico Médio, uma nova ancestralidade do Levante aparece no Magreb, coincidindo com a chegada do pastoreio na região, e todas as três ancestralidades se misturam durante o Neolítico Tardio. Nossos resultados mostram mudanças de ancestralidade na neolitização do noroeste da África que provavelmente refletiam uma paisagem econômica e cultural heterogênea, em um processo mais multifacetado do que o observado em outras regiões.
Conteúdo semelhante sendo visualizado por outros
Principal
A localização geográfica do Norte da África, centrada entre o vasto deserto do Saara, o fértil Oriente Próximo e a Europa Mediterrânea, resultou em uma história humana complexa na área 7 , 8 . O registro fóssil sugere presença hominídea e humana de longo prazo 9 , embora a continuidade ao longo dos últimos 100.000 anos não possa ser deduzida devido à natureza fragmentada do registro. No Pleistoceno Superior, 15.000 anos atrás, os restos de forrageadores escavados no Marrocos mostram uma composição genética distinta intermediária entre os forrageadores levantinos contemporâneos e as populações da África subsaariana 10 . Os norte-africanos atuais são amplamente relacionados às populações eurasianas, o que provavelmente foi causado por migrações de "retorno à África" 7 .
Tanto os registros arqueológicos quanto os dados arqueogenômicos mostram que os agricultores neolíticos (geneticamente distintos dos forrageadores europeus) se dispersaram do norte do Levante e da Anatólia para as ilhas do Mediterrâneo, península Itálica e Ibéria 11 , 12 , 13 , 14 , 15 , 16 , 17 , 18 . As rotas costeiras do Mediterrâneo são há muito reconhecidas no registro arqueológico como uma parte importante da expansão neolítica na Europa. No Mediterrâneo ocidental, a tecnologia de cerâmica impressa — e mais adiante o horizonte cardial — se espalhou ao longo da costa continental europeia e ilhas para chegar à península ibérica, onde ambos os fenômenos estão presentes em 7.550 anos calibrados antes do presente (cal BP ) (refs. 19 , 20 ).
Enquanto alguns estudos apoiam uma aparição simultânea do Neolítico no noroeste da África (Rif Oriental, sítio Ifri Oudadane) e na Península Ibérica por volta de 7.550 cal A.P. (ref. 21 ), a evidência mais antiga de cerâmica, cereais domésticos e criação de animais é encontrada no norte do Marrocos aproximadamente dois séculos depois em Kaf Taht el-Ghar (KTG) por volta de 7.350 cal A.P. (refs. 2 , 3 , 22 , 23 ). Embora a cultura material do Neolítico Inicial e os primeiros mamíferos e leguminosas domésticas sugiram uma conexão com a Península Ibérica 1 , 2 , 3 , a extensão e o legado dessas conexões permanecem obscuros. No entanto, a primeira análise genômica de agricultores do Neolítico Inicial do noroeste da África (do sítio Ifri n'Amr o'Moussa (IAM) no centro do Marrocos) não mostra vestígios de mistura com agricultores do Neolítico Europeu. Em vez disso, ele mostra continuidade populacional de longo prazo desde o Paleolítico Superior na região 6 . Este resultado se alinha com a hipótese de que a transição neolítica no noroeste da África foi iniciada por comunidades epipaleolíticas locais adotando inovações tecnológicas 4 , 5 , como aquelas encontradas no IAM: cerâmicas impressas semelhantes às de Cardial, semelhantes às presentes em toda a Europa neolítica do Mediterrâneo ocidental, e cereais domésticos (por exemplo, um grão de Hordeum vulgare datado em torno de 7.050 cal A.P. ) 2 . Este padrão implica um processo de neolitização que contrasta marcadamente com o da Europa, onde foi estabelecido que a agricultura foi introduzida pela difusão demica para o oeste e norte dos primeiros agricultores da Anatólia 11 , 12 . O desenvolvimento local, ou aculturação, do Neolítico do Norte da África é ainda apoiado por sinais de grupos epipaleolíticos cada vez mais sedentários desenvolvendo estratégias para o gerenciamento de recursos, como a exploração de plantas selvagens e cerâmica 1 , 4 , 24 , 25 , 26 . Mudanças climáticas rápidas favoreceram o pastoreio móvel 27 e, embora tenha sido levantada a hipótese de que o gado foi domesticado independentemente no Saara 28 , dados de radiocarbono sugerem uma introdução gradual do pastoreio no Saara em uma direção sudoeste de 7.000 a 6.000 cal A.P. , possivelmente do Oriente Próximo 29 , 30 .
Enquanto estudos paleogenômicos sobre a transição neolítica mediterrânea europeia são abundantes 15 , 31 , 32 , 33 , o norte da África tem sido o foco de apenas um único estudo que gerou dados genéticos humanos de um sítio neolítico inicial e um tardio 6 , deixando lacunas substanciais na cronologia dos eventos. É evidente que o sítio do IAM mostra um estilo de vida neolítico e uma ausência de ancestralidade neolítica europeia, mas se este foi um desenvolvimento independente ou se a inspiração veio de outros grupos no noroeste da África ou do outro lado do Mar Mediterrâneo permanece incerto. Portanto, a linha do tempo e os processos envolvidos na neolitização da região, a natureza e a dinâmica de diferentes economias no norte da África e o papel que elas podem ter desempenhado no neolítico europeu mais amplo permanecem pouco estudados e controversos.
Neste estudo, investigamos uma série temporal de restos humanos de quatro sítios arqueológicos que abrangem o Epipalaeolítico ao Neolítico Médio no atual Marrocos: o sítio Epipalaeolítico de Ifri Ouberrid (OUB), os sítios do Neolítico Inicial de IAM e KTG e o cemitério do Neolítico Médio de Skhirat-Rouazi (SKH), coanalisados com dados genéticos publicados anteriormente daquela região 6 , 10 . Ao sequenciar os genomas de nove indivíduos escavados desses quatro sítios arqueológicos, podemos demonstrar que a transição neolítica no noroeste da África foi iniciada pela migração de agricultores neolíticos da Europa Mediterrânea.
Geramos dados de sequência genômica de nove indivíduos antigos do Marrocos moderno (Tabela 1 ), variando em cobertura genômica de 45,75 a 0,017 vezes, incluindo cinco indivíduos com cobertura de mais de uma vez e três com mais de nove vezes. Cronologicamente, os dados abrangem mais de 1.000 anos, cobrindo o Epipaleolítico Tardio ( n = 1), Neolítico Inferior ( n = 5) e Neolítico Médio ( n = 3). Dois sítios do Neolítico Inferior foram estudados — KTG ( n = 4) e IAM — onde coanalisamos os dados genômicos recém-gerados de um indivíduo e aqueles relatados anteriormente 6 (Fig. 1a,b ). Bibliotecas de DNA foram geradas a partir de extratos de DNA obtidos de ossos e dentes e posteriormente sequenciados shotgun em uma plataforma Illumina. Todas as bibliotecas apresentaram os padrões de degradação esperados do DNA antigo, incluindo tamanhos de fragmentos curtos e desaminação de citosina nas extremidades de leitura (Fig. 1 Suplementar ). As estimativas de contaminação foram geralmente baixas tanto para o genoma nuclear quanto para as mitocôndrias, exceto para o indivíduo skh003, que mostrou 10–16% de contaminação nuclear (Tabela 1 ). Para avaliar a relação dos antigos indivíduos do noroeste da África com outras populações antigas e atuais da Eurásia Ocidental e da África, coanalisamos nossos dados com grupos relevantes antigos (Dados Suplementares 2 ) e atuais da África, Oriente Médio e Europa 34 .
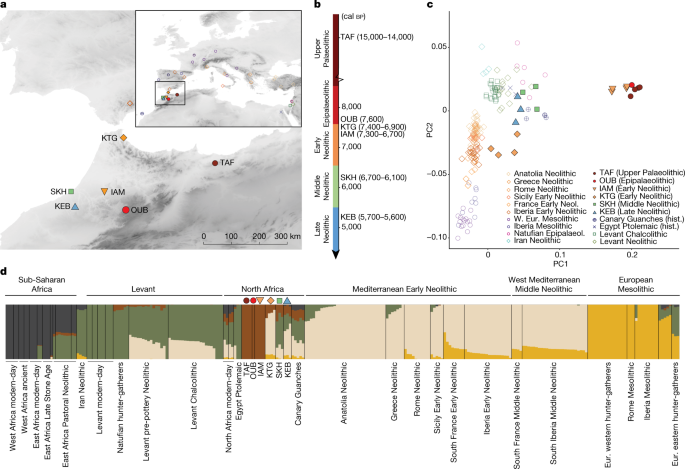
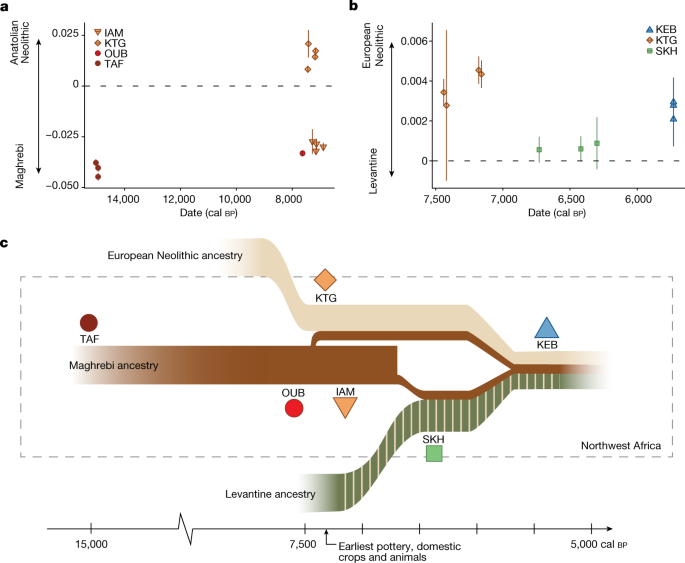
a , Localização geográfica dos sítios arqueológicos investigados. Legenda de símbolos dada em c . O mapa foi gerado usando o Sistema de Informação Geográfica QGIS de código aberto, http://qgis.osgeo.org . b , Representação cronológica dos períodos arqueológicos investigados do noroeste da África, com a linha do tempo datada por radiocarbono de cada sítio indicada. c , Visão ampliada de um gráfico PCA (Fig. Suplementar 3 ) com foco nos indivíduos antigos analisados. Cada indivíduo antigo projetado é representado por um símbolo colorido. W. Eur., Europa Ocidental; hist., histórico. d , Proporções de ancestralidade estimadas para indivíduos africanos, do Oriente Médio e europeus (Eur.) modernos e antigos relevantes (assumindo cinco componentes de ancestralidade; resultados adicionais são apresentados na Fig. Suplementar 4 ). Populações/indivíduos pré-neolíticos e neolíticos do noroeste da África são destacados pelos mesmos símbolos usados em a e c .
Oito mil anos de continuidade populacional
Do povo do Paleolítico Superior de Taforalt (TAF) através do Epipalaeolítico em OUB até o Neolítico Inicial em IAM, observamos a persistência da composição genética única que existia nos habitantes do noroeste da África há 15.000 anos (Fig. 1c,d e Fig. Suplementar 5 ), e possivelmente ainda mais para trás no tempo. O indivíduo Epipalaeolítico oub002, datado de 7.660–7.506 cal BP , é geneticamente muito semelhante aos indivíduos de TAF (15.086–14.046 cal BP ) 35 e aos indivíduos do Neolítico Inicial de IAM (7.316–6.679 cal BP ; Fig. 1 ) 6 , 36 . O genoma de Oub002 demonstra uma continuidade populacional marcante no noroeste da África, sem fluxo genético substancial através do Mar Mediterrâneo por pelo menos 7.000 anos durante o Epipaleolítico (Fig. 1c,d ), ligando a ancestralidade genética magrebina encontrada no Paleolítico Superior aos indivíduos do Neolítico Inferior no IAM.
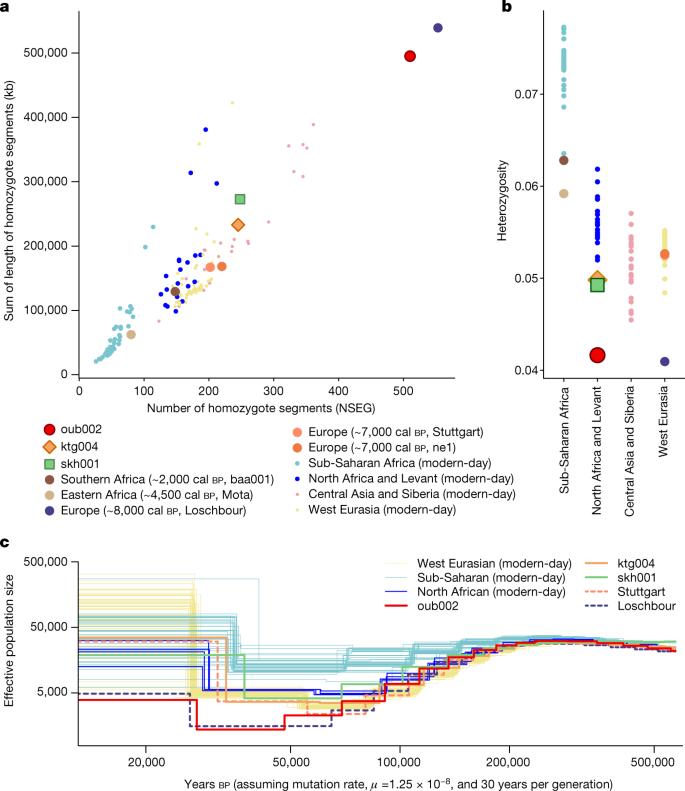
A linhagem Maghrebi mostra uma diversidade genética excepcionalmente baixa 6 , 10 (Fig. 2b e Fig. Suplementar 9 ) e longas e frequentes séries de homozigose (RoH) (Fig. 2a ), provavelmente como consequência de isolamento duradouro. Por meio da investigação do genoma de 45,8 vezes de oub002, mostramos que os antigos africanos do noroeste passaram por um grave gargalo populacional. Até cerca de 70.000–60.000 anos atrás, as mudanças no tamanho efetivo da população ( N e ) de oub002 seguem um padrão semelhante ao das populações eurasianas com um tamanho efetivo populacional relativamente pequeno alcançado há 50.000 anos (Fig. 2c ), o que é consistente com a linhagem Maghrebi sendo relacionada às populações que migraram para fora da África. Curiosamente, o tamanho efetivo da população dos eurasianos e norte-africanos modernos, bem como dos eurasianos neolíticos, permaneceu em torno de 5.000 até cerca de 30.000 anos atrás, mas o tamanho efetivo da população da linhagem magrebina continua a diminuir e atingiu seu ponto mais baixo ( N e ≈ 1.400) entre 50.000 e 27.000 anos atrás, durante o pico da Última Glaciação. Padrões notavelmente semelhantes são observados para os caçadores-coletores ocidentais (WHG) do Mesolítico da Europa (representados por Loschbour na Fig. 2c ), para os quais medidas de baixa diversidade foram atribuídas a altos níveis de parentesco de fundo e autozigosidade devido ao pequeno tamanho da população 37 .
a – c , Indivíduos antigos (incluindo oub002, ktg004 e skh001) são comparados com indivíduos modernos de regiões geograficamente correspondentes. a , Execuções de homozigose. ne1, Neolítico Europeu 1; NSEG, número de segmentos homozigotos. b , Heterozigosidade, calculada a partir do número de posições variáveis por indivíduo dividido pelo número de sítios de polimorfismo de nucleotídeo único (SNP) por indivíduo. c , Tamanho efetivo da população ao longo do tempo, conforme inferido pela coalescência Markoviana sequencial em pares, para três antigos africanos do noroeste com cobertura genômica de mais de nove vezes, bem como para um indivíduo europeu mesolítico (Loschbour) e um indivíduo europeu neolítico (Stuttgart), e indivíduos modernos para comparação.
Agricultores europeus induzem a neolitização
No sítio do IAM, uma multidão de artefatos representando o pacote neolítico foi identificada. No entanto, foi demonstrado que as pessoas que vivem no IAM apresentam ancestralidade magrebina autóctone 6 e eram descendentes de grupos anteriores (Paleolítico Superior e Epipaleolítico) do noroeste da África (Fig. 1c,d ). Essas duas observações apoiam a visão de que o primeiro estágio da transição neolítica no Marrocos foi impulsionado por populações locais adotando inovações tecnológicas com base em contatos através do Mediterrâneo 2 .
O sítio neolítico inicial de KTG, localizado na costa mediterrânea do norte da África, perto do estreito de Gibraltar (Fig. 1a ), é anterior e se sobrepõe parcialmente no tempo ao IAM 2 (Tabela 1 ). No KTG, uma assembleia neolítica completa é encontrada, incluindo uma diversidade de cereais cultivados, mamíferos domésticos e cerâmicas cardiais 38 , 39 . Em contraste com as pessoas no IAM, aquelas no KTG são geneticamente semelhantes às populações neolíticas iniciais europeias (Figs. 1c,d e 3a ). Curiosamente, todos os quatro indivíduos KTG mostram mistura (15,4–27,4%) com grupos locais do norte da África (Fig. 1d ), consistente com valores significativamente positivos para o teste f 4 de mistura (KTG, EN Mediterrâneo; TAF, Mbuti) (Dados Suplementares 7 ). Além disso, identificamos uma pequena proporção de ancestralidade WHG em KTG (Fig. 1d ), consistente com a observação de europeus do Neolítico Inicial carregando ancestralidade WHG 14 , 15 , 31 , 33 , 40 . Um modelo de história populacional para o povo KTG com ancestralidade Neolítica Anatólia de 72 ± 4,4%, ancestralidade WHG de 10 ± 2,6% e ancestralidade Magrebina de 18 ± 3,3% é consistente com os dados (qpAdm, P = 0,193). Tomados em conjunto, esses resultados sugerem uma origem Neolítica Europeia de fazendeiros KTG cujos ancestrais se dispersaram da Anatólia por toda a Europa, misturando-se com caçadores-coletores europeus em seu caminho para o sudoeste da Europa 33 , 40 antes de cruzar o Mediterrâneo para o Norte da África. A presença de ancestrais caçadores-coletores europeus exclui a possibilidade de que migrantes do Neolítico Inferior tenham seguido exclusivamente as costas do Mediterrâneo Norte-Africano a partir da Anatólia ou do Levante.
a , Afinidade genética de indivíduos analisados do noroeste da África da Idade da Pedra, polarizada entre a ancestralidade Neolítica da Anatólia e Magrebina, usando o teste f 4 da forma f 4 (Neolítico da Anatólia, TAF011; indivíduos do noroeste da África da Idade da Pedra, Mbuti). b , Afinidade genética de indivíduos do noroeste da África da Idade da Pedra, polarizada entre a ancestralidade Neolítica Ibérica e Levantina usando o teste f 4 da forma f 4 (Neolítico Inicial da Ibéria, Calcolítico do Levante; indivíduos Neolíticos do noroeste da África, Mbuti). a , b , Cada símbolo representa um único valor f 4 individual . As barras de erro indicam ±2 se, computado com uma abordagem de bloco jack-knife (blocos de 5 Mb ponderados pelo número de SNPs). c , Resumo da história populacional inferida do Magrebe da Idade da Pedra.
O Neolítico Inicial Ibérico (tanto como um todo quanto regionalmente) foi considerado a melhor população fonte para a ancestralidade europeia no KTG, seguido pelo Neolítico Inicial Sicília Stentinello (Dados Suplementares 9 ). Isso é consistente com baixos níveis de diferenciação genética em grupos associados à cerâmica Cardial ao longo das costas europeias do Mar Mediterrâneo 41 , confirmado por datações diretas de radiocarbono mostrando que os fazendeiros de cerâmica impressa se expandiram rapidamente pelo Mediterrâneo ocidental 3 , 19 , 42 .
Tem sido debatido se os agricultores europeus cruzaram da Península Ibérica para o Marrocos 2 , 3 ou se travessias anteriores do Mediterrâneo teriam acontecido, através do Estreito Siciliano-Tunísia seguido por uma rota de expansão Magrebi 4 , 43 . Comparações diretas de agricultores do Neolítico Inicial da Sicília e da Península Ibérica como ancestrais de agricultores KTG fornecem evidências mais fortes para uma origem Neolítica Ibérica (Dados Suplementares 9 e Informações Suplementares 8 ), mas não podemos excluir alguma contribuição de agricultores sicilianos. Dados genéticos são consistentes com a explicação mais parcimoniosa para evidências arqueológicas sobre a transição Neolítica no noroeste da África: a travessia do sul da Península Ibérica por agricultores Neolíticos Ibéricos 2 , 23 . A proximidade geográfica entre o sul da Península Ibérica e a Península Tangitana acrescenta força a esta observação, enquanto a falta de evidências arqueológicas confiáveis de elementos domésticos antigos em sítios relevantes ao longo do Magreb oriental e Túnis, incluindo sítios com cerâmica e obsidiana da Ilha de Pantelleria, enfraquece a hipótese do cruzamento Sicília-Túnis 3 . Curiosamente, o fluxo gênico do Norte da África foi encontrado apenas em indivíduos europeus mediterrâneos muito mais tarde, por volta de 4.500 anos atrás 31 , 44 .
Diferentes indivíduos do KTG datam de períodos de tempo ligeiramente diferentes. Encontramos uma proporção duas vezes maior de ancestralidade magrebina em indivíduos KTG anteriores (aproximadamente 25%, ktg001 e ktg005, aproximadamente 7.429–7.267 cal BP ) do que nos posteriores (cerca de 13%, ktg004 e ktg006, em torno de 7.247–6.945 cal BP ) (Fig. 1d ). Isso coincide com um aumento na ancestralidade neolítica europeia, mostrado pelo resultado significativamente negativo para f 4 (KTG anterior, KTG posterior, Neolítico Inicial da Ibéria, Mbuti; pontuação z = −5,01). Aproximadamente um quarto da ancestralidade magrebina no KTG inicial sugere que eles representam pelo menos a segunda geração de cruzamento entre os grupos. Estimamos o tempo de mistura usando duas abordagens baseadas em padrões de covariância de ancestralidade e decaimento de desequilíbrio de ligação, usando o Neolítico Inicial da Ibéria ou Sicília e TAF como fontes de mistura. Ambos os métodos datam o contato nas últimas seis a 13 gerações (Informações Suplementares 8 ), sugerindo que a mistura entre grupos ocorreu por algumas centenas de anos, o que é consistente com a análise do estilo de cerâmica que aponta para o primeiro contato em 7.500–7.400 cal BP (ref. 23 ).
Os agricultores de Kaf Taht el-Ghar tinham níveis de diversidade genética ligeiramente mais baixos e maior RoH do que a maioria das populações europeias do Neolítico Inicial (Fig. 2a,b e Fig. Suplementar 9 ). A ancestralidade magrebina carregada pelo povo KTG mostra uma diversidade marcadamente menor e RoH mais extensa, e é provavelmente a causa da redução na diversidade geral. Evidências arqueológicas sugerem que a agricultura do Neolítico Inicial era restrita a enclaves no Magreb mais ocidental, possivelmente devido a restrições climáticas ao sul 4 , 22 . Isso pode ter limitado o potencial desses grupos de se recuperarem de um efeito fundador inicial.
No geral, os padrões genéticos de interação local entre diferentes grupos no noroeste da África são comparáveis aos encontrados na Europa: os agricultores assimilaram a ancestralidade dos forrageadores locais em um processo de mistura unidirecional. Casos de comunidades de caçadores-coletores adotando certos elementos do Neolítico foram descritos na Europa 11 , 14 , 45 . No entanto, o processo de neolitização do noroeste da África envolveu a notável sobrevivência de populações locais geneticamente não misturadas (representadas por IAM), apesar de coexistirem por pelo menos 300 anos com comunidades agrícolas estrangeiras (KTG), e ainda adotaram vários elementos dos modos de vida neolíticos delas. Enquanto as descobertas arqueológicas em IAM e KTG apontam para a troca de ideias entre grupos e apoiam um processo de aculturação de comunidades forrageiras 1 , 4 , nossos dados genéticos mostram que a troca de genes foi unidirecional.
Influxo de ascendência levantina
Outra ancestralidade distinta foi introduzida no noroeste da África durante o Neolítico Médio. Todos os indivíduos do SKH mostram grandes proporções de um componente genético maximizado em indivíduos do Levante Neolítico e Calcolítico, Egito Ptolomaico e populações modernas do Oriente Próximo (Fig. 1d ). A ancestralidade no SKH pode ser modelada como uma mistura bidirecional entre populações do Neolítico do Levante (aproximadamente 76,4 ± 4,0%) e africanos locais do noroeste (representados por TAF; 23,6 ± 4,0%). Se uma população fonte adicional do Neolítico Europeu (por exemplo, da Ibéria) for adicionada, o modelo é rejeitado.
Como essa ancestralidade levantina neolítica não foi observada no lado europeu do Mediterrâneo durante o Neolítico, ela provavelmente representa uma expansão independente de pessoas do Levante para o norte da África. Migrações do Levante para o leste da África foram identificadas para indivíduos pastores neolíticos há cerca de 4.000 anos, que são presumivelmente descendentes de populações não amostradas do nordeste da África associadas à disseminação do pastoreio saariano 46 . Tanto no SKH quanto nos pastores neolíticos do leste da África, a ancestralidade levantina é misturada com ancestralidades locais (Fig. 1d , Informações Suplementares 8 e Dados Suplementares 12 ). A chegada dessa ancestralidade levantina coincide com o surgimento de uma nova tradição cerâmica no norte do Marrocos, frequentemente caracterizada por motivos impressos em cordão ('roleta' ou linha ondulada), como os bens funerários em Skhirat pertencentes à cerâmica Ashakar Ware 47 , 48 . Paralelamente, a pastorícia de gado expandiu-se no actual território do Saara 30 , 47 e os grupos de línguas afro-asiáticas espalharam-se por todo o Norte de África 22 .
Nossas análises mostram que o componente associado ao Levante também permanece no Magreb durante o Neolítico Tardio em indivíduos de Kehf el Baroud (KEB) e nos Guanches das Ilhas Canárias (cerca de 1.000 cal A.P.; Fig. 1c,d ) 6 , 49. Indivíduos desses locais são deslocados para populações antigas do Levante no espaço de análise de componentes principais (PCA) (Fig. 1c ). Isso destaca os processos demográficos complexos que ocorreram no noroeste da África, em contraste com o aumento gradual na ancestralidade caçadora-coletora descrita na Europa do Neolítico Médio e Tardio 32 , 33 , 40 .
Os indivíduos do Neolítico Tardio do KEB podem ser modelados como uma mistura de ancestrais já presentes no noroeste da África durante o Neolítico Inferior e o Neolítico Médio, sugerindo que não houve ondas de migração substancial para esta região entre o Neolítico Médio e o Neolítico Tardio (Informações Suplementares 8 e Dados Suplementares 13 ).
Conclusão
A complexa estrutura populacional no noroeste da África moderna tem sido associada a vários eventos históricos, como a expansão árabe 7 , 8 . No entanto, nossa cronologia detalhada e dados genômicos de alta resolução fornecem uma nova compreensão desses processos pré-históricos no Magreb e revelam um substrato genético rico e diversificado com origem neolítica. Primeiro, as populações humanas no noroeste da África mostram continuidade genética e isolamento desde o Paleolítico Superior, de pelo menos 15.000 a cerca de 7.500 anos atrás, quando esse período de isolamento foi interrompido pela migração de grupos europeus do Neolítico Inicial que introduziram práticas agrícolas. Portanto, apesar de uma distância geográfica relativamente pequena entre o sul da Península Ibérica e o noroeste da África (a distância hoje é de apenas 13 km através do estreito de Gibraltar) e do fato de que ambas as regiões foram povoadas por forrageadores por muitos milênios antes do Neolítico, o fluxo gênico através do Mar Mediterrâneo não foi estabelecido até o Neolítico Inicial. Os recém-chegados trouxeram novos modos de vida, práticas agrícolas, domesticação e tradições de cerâmica que foram posteriormente adotadas pelas populações locais. Nossos resultados mostram que o processo de neolitização no noroeste da África foi iniciado por europeus neolíticos migrantes, mas que grupos locais (pelo menos os indivíduos analisados no IAM) adotaram algumas dessas práticas sem se misturarem com os recém-chegados. Dois grupos geneticamente distintos coexistiram em estreita proximidade na região. Curiosamente, o conhecimento cultural e tecnológico parece ter sido transferido principalmente de agricultores neolíticos europeus para grupos locais (por exemplo, no IAM), enquanto a ancestralidade genética fluiu apenas de grupos locais para os agricultores que chegavam, como a população de KTG. Além disso, no Neolítico Médio, uma nova ancestralidade com origem oriental é detectada no noroeste da África. Essa ancestralidade indica novos grupos migrantes, potencialmente associados a pastores do Saara, que se misturaram com grupos locais (Fig. 3c ).
As várias ondas de migração e mistura para o noroeste da África durante o Neolítico possivelmente resultaram em uma paisagem econômica e cultural heterogênea naquela região — um mosaico de grupos que incluía agricultores vindos da Península Ibérica, forrageadores adotando práticas agrícolas e pastores orientais se misturando com pessoas locais. A maioria desses grupos mostrou tamanho populacional efetivo reduzido e menor diversidade do que as populações contemporâneas na Europa (Fig. 2 ), sugerindo que os tamanhos populacionais permaneceram modestos durante todo o Neolítico. Esses padrões foram provavelmente causados por períodos de isolamento, o que pode ter contribuído para a ancestralidade genética distinta vista no Magreb hoje. Um estudo recente da Idade do Ferro sugere que o noroeste da África permaneceu como lar de um conjunto diversificado de grupos ao longo da pré-história 50 , tornando esta parte do mundo um dos lugares mais exclusivos a serem estudados com o kit de ferramentas arqueogenômicas.
Métodos
Descrições detalhadas de cada seção podem ser encontradas em Informações Suplementares .
Amostragem arqueológica
Os restos humanos antigos analisados neste estudo derivam de um acordo de cooperação científica entre as Universidades INSAP, La Trobe e Uppsala. Elementos completos de ossos e dentes foram trazidos para a antiga instalação de DNA em Uppsala, Suécia, para posterior limpeza e amostragem.
Datação por radiocarbono
Todos os indivíduos investigados foram datados diretamente por radiocarbono no Tandem Laboratory, Uppsala, exceto ktg001, que foi datado no laboratório de datação por carbono analítico Beta, e iam004, cuja data foi obtida da referência 6. A calibração de radiocarbono para datas recentemente relatadas e datas relevantes publicadas anteriormente foi realizada usando Oxcal v.4.4 e o conjunto de dados IntCal20 41 .
Recuperação de DNA antigo
Restos humanos foram coletados em laboratórios dedicados de sala limpa na Universidade de Uppsala, Suécia, após uma série de procedimentos rigorosos visando minimizar a contaminação da superfície óssea e dentária. Trinta a sessenta miligramas de pó ósseo ou pedaços sólidos de material ósseo foram usados para extração de DNA seguindo a ref. 51 , com adaptações conforme descrito na ref. 15 , ou seguindo a ref. 52 , com adaptações ao tampão de ligação e uma etapa inicial de pré-digestão com 1 ml de 0,5 M EDTA pH 8,0 por 30 min a 37 °C 53. A digestão da amostra foi realizada durante a noite com 1 ml de 0,45 M EDTA pH 8,0 e 0,2 mg ml –1 de proteinase K. Bibliotecas de DNA de fita dupla e extremidade romba reparada foram construídas com adaptadores P5 e P7 ligados 54 . Após a avaliação da autenticidade, qualidade e quantidade do DNA (por estimativa do conteúdo endógeno de DNA, padrões de desaminação post mortem e distribuição do tamanho do fragmento), o extrato de DNA restante (para amostras com mais de 1% de proporção de DNA humano) foi usado para construir quatro a seis bibliotecas adicionais de DNA fita dupla; para extratos com aproximadamente 5% de conteúdo humano endógeno ou mais, 15–20 µl de extrato de DNA foram tratados com uracil DNA glicosilase (UDG) para construção de biblioteca fita dupla 55 . As bibliotecas foram amplificadas por PCR usando um primer indexado exclusivo de 7 pb 54 , 56 em quatro reações de 25 µl ou duas de 50 µl, com a aplicação de 12–20 ciclos de PCR dependendo da indicação do ciclo de quantificação qPCR anterior. Dois controles negativos de extração, dois controles negativos de biblioteca e um controle negativo de PCR foram incluídos por lote de amostra. As reações de PCR foram reunidas e purificadas com esferas AMPure XP (Agencourt, Beckman Coulter). A qualidade da biblioteca foi verificada por eletroforese em Tapestation (Agilent High Sensitivity D1000 ScreenTape, Agilent) e a concentração de DNA foi quantificada usando um Qubit dsDNA HS (High Sensitivity) Assay Kit (Invitrogen). Pools equimolares de bibliotecas amplificadas e purificadas foram sequenciadas em Illumina HiSeq X na SNP & SEQ Technology Platform em Uppsala. Para atingir uma cobertura maior, entre quatro e dez bibliotecas foram reunidas equimolarmente e sequenciadas até a depleção.
Processamento e autenticação de dados de bioinformática
Os dados foram demultiplexados de acordo com a sequência de primer indexada e os adaptadores foram aparados com MergeReadsFastQ_cc.py 57 ou Adapter Removal v.2.1.7 (ref. 58 ). Leituras de extremidade pareada direta e reversa foram mescladas quando uma sobreposição de pelo menos 11 pb foi encontrada. O mapeamento contra a construção 37 do genoma de referência humano (hs37d5) foi feito usando o alinhador Burrows–Wheeler 0.7.13 (ref. 59 ). Para cada biblioteca, mesclamos os arquivos bam resultantes de todas as rodadas de resequenciamento usando o SAMtools merge v.1.5 (ref. 60 ). Em seguida, mesclamos separadamente os dados das bibliotecas tratadas e não tratadas com UDG para cada indivíduo e usamos dados das primeiras para análise subsequente, exceto para os indivíduos ktg001 (para os quais apenas dados não UDG foram gerados) e iam004 (para os quais os dados tratados e não tratados foram mesclados e processados como não UDG tratados para análise posterior). Usamos uma versão modificada de FilterUniqSAMCons_cc.py 57 para garantir a escolha aleatória de bases para colapsar leituras com posições iniciais e finais idênticas em um consenso, removendo assim duplicatas de PCR. Leituras menores que 35 pb e mais de 10% de incompatibilidades com o genoma de referência humano foram filtradas.
Contaminação, determinação sexual, marcadores uniparentais e análises de parentesco
As estimativas de contaminação da amostra foram obtidas usando três métodos diferentes com base no genoma mitocondrial 61 , no cromossomo X em machos 62 e em dados nucleares 63 (Dados Suplementares 3 ). A proporção de cobertura dos cromossomos X e Y em relação aos autossomos foi usada para determinar o sexo biológico de cada indivíduo 64 . Geramos sequências de consenso mitocondrial usando SAMtools 1.5 mpileup e vcfutils.pl 60 , 65 . As pontuações de qualidade de base (BQ) e de mapeamento (MAPQ) foram definidas como MAPQ > 30 e BQ > 30, e apenas locais com cobertura pelo menos tripla foram usados. Os haplogrupos foram atribuídos usando Haplogrep 2.1.16 (ref. 66 ) e PhyloTree mtDNA tree Build 17 (18 de fevereiro de 2016) 67 (Dados Suplementares 4 ). Para a inferência do haplogrupo Y, chamamos SNPs da Sociedade Internacional de Genealogia Genética ( http://isogg.org ; v.11.110, 21 de abril de 2016)) de arquivos bam usando o SAMtools mpileup com a opção -B. Extraímos locais com mapeamento e qualidade de base maiores que 30. Inserções e deleções, e locais mostrando múltiplos alelos, foram excluídos (Dados Suplementares 5 ).
Executamos análise de parentesco com READ 68 dentro de cada sítio arqueológico (mínimo de três indivíduos; Fig. Suplementar 2 ). Quando um par de indivíduos com parentesco próximo foi encontrado, como relacionamentos de primeiro grau (pai-filho ou um irmão completo), excluímos o indivíduo com menos SNPs cobertos das análises. Isso resultou na remoção da análise de iam4 (mesmo indivíduo que iam5), keb8 (mesmo indivíduo que keb1), iam6 (primeiro grau relativo a iam004) 6 e TAF012 (primeiro grau relativo a TAF011) 10 .
Análise genômica populacional de dados pseudohaplóides
Dados de mais de 300 indivíduos antigos da Eurásia, do Norte da África e da África Subsaariana, organizados de acordo com a geografia e a cronologia (Dados Suplementares 2 ), foram baixados, mapeados e processados pelo mesmo pipeline usado para dados recém-gerados. O conjunto completo de dados de DNA antigo foi mesclado com indivíduos modernos publicamente acessíveis amostrados em todo o mundo do conjunto de dados do Simons Genome Diversity Project (SGDP) 34 para um painel de 2,2 milhões de SNP 64 . Os alelos foram amostrados de arquivos bam, extraindo aleatoriamente uma leitura com MAPQ > 30 e BQ > 30 por local SNP para cada indivíduo antigo (usando SAMtools v.1.5.0 mpileup com a opção -B), e essa posição foi tratada como (pseudo)haploide. Para dados não tratados com UDG (ktg001) ou dados mesclados de UDG e não UDG (iam004), cortamos 10 pb das extremidades da sequência para evitar a integração de substituições de C para T e G para A com codificação incorreta. Para os dados tratados com UDG parciais publicados (metade UDG), 2 pb foram cortados das extremidades da sequência. Os SNPs que mostravam mais de dois alelos foram excluídos dos dados, deixando 1.379.466 SNPs para análise.
A análise de componentes principais foi realizada usando smartpca v.10210 (ref. 69 ). Os componentes principais foram calculados com base em indivíduos de 18 populações modernas do Mediterrâneo, Eurásia ou Norte da África do SGDP. Indivíduos antigos foram projetados no espaço PCA com as opções shrinkmode: YES e lsqproject: YES. Um algoritmo de agrupamento baseado em modelo não supervisionado, implementado em ADMIXTURE v,1.3.0 (ref. 70 ), foi realizado para K = 3–5 (30 execuções) em um conjunto de dados totalmente pseudo-haploidizado, com desequilíbrio de ligação podado de indivíduos modernos e antigos de populações do Mediterrâneo, Eurásia ou Norte da África, deixando 812.092 SNPs para análise. Os resultados foram analisados, alinhados e plotados com pong 71 .
Popstats 72 foi usado para calcular estatísticas f- 73 , com Mbuti definido como o grupo externo (Dados Suplementares 6 e 7 ). Estatísticas f 3 do grupo externo foram computadas com a opção –f3vanilla. Erros-padrão (EPs) foram calculados com uma abordagem de bloco ponderado jack-knife.
A modelagem de mistura foi realizada com qpAdm 74 usando ADMIXTOOLS v.5.0, por meio de uma versão adaptada do qpAdm_wrapper ( https://github.com/pontussk/qpAdm_wrapper ) que percorre todos os subconjuntos possíveis da lista de populações de origem fornecida (selecionada com base em resultados anteriores), para testar modelos de mistura de uma, duas, três e quatro vias. Os SEs foram computados com jack-knife de bloco de 5 cM. Usamos um conjunto de 11 populações de referência cujo poder de desembaraçar linhagens divergentes de ancestralidade presentes na Europa, Norte da África e Oriente Próximo foi descrito anteriormente e que são diferentes relacionadas às fontes testadas 10 , 31 , 75 . Fontes distantemente relacionadas foram exploradas e, quando possível, também grupos mais próximos (geograficamente, cronologicamente ou de acordo com evidências arqueológicas existentes). Tentamos encontrar os modelos mais parcimoniosos consistentes com os dados ( P > 0,05) verificando o menor número possível de fontes de ancestralidade necessárias para explicar a ancestralidade em cada população de teste (Dados Suplementares 8 – 13 ). O evento de mistura em KTG foi datado usando ALDER 76 e DATES 77 (Informações Suplementares 8 ). Calculamos a diversidade condicional de nucleotídeos 78 pela estimativa do número médio de incompatibilidades entre dois indivíduos da mesma população. Os SEs foram estimados usando uma abordagem de bloco jack-knife e um tamanho de bloco de 2.000 SNPs (Fig. Suplementar 9 ).
Análise genômica populacional de dados diplóides
Chamadas de genótipo diploide para um painel de 49.791.572 SNPs foram realizadas para indivíduos antigos do noroeste da África com cobertura genômica de pelo menos nove vezes (oub002, ktg004 e skh001), bem como indivíduos antigos relevantes publicados anteriormente com genomas de alta cobertura sequenciados. Antes da chamada do genótipo, a qualidade da base nas extremidades de leitura foi reduzida e o realinhamento de indel foi conduzido com GATK 3.5.0. Os genótipos diploides foram chamados usando dbSNP v.142 como SNPs conhecidos, com UnifiedGenotyper 79 do GATK . Calculamos a profundidade média de sequenciamento (avg.DP) sobre todas as posições chamadas para cada indivíduo e filtramos para QUAL > 30 e um intervalo de profundidade de cinco vezes a 3× avg.DP por indivíduo, usando a visualização BCFtools. Este conjunto de dados foi mesclado com dados de indivíduos modernos do conjunto de dados SGDP.
A heterozigosidade individual foi calculada a partir do número de posições variáveis dividido pelo de SNPs sequenciados, usando o comando –het no PLINK 1.9 (ref. 80 ). Estimamos o comprimento e o número de execuções de homozigosidade após a filtragem com o comando PLINK –geno 0. Os arquivos de entrada do MSMC 81 foram gerados a partir de arquivos VCF. Filtros para MAPQ > 30, qualidade mínima do genótipo de 50 e profundidade de sequenciamento foram usados. Os locais que não passaram por esses filtros foram mascarados por indivíduo. O MSMC 0.1.0 foi então executado para cada indivíduo.
Declaração de ética e inclusão
A amostragem para este estudo surgiu de projetos de arqueologia que envolveram universidades e pesquisadores locais, incluindo YB, cujo envolvimento no design da pesquisa incluiu a seleção de material arqueológico para análises, bem como a supervisão da amostragem. A relevância local desta pesquisa está vinculada à história da região, e é localmente relevante no que diz respeito à descrição do passado humano no noroeste da África. O estudo foi realizado com os mais altos padrões de pesquisa arqueogenômica, e pesquisas relevantes de acadêmicos locais foram citadas.
Resumo do relatório
Mais informações sobre o desenho da pesquisa estão disponíveis no Resumo do Relatório de Portfólio da Nature vinculado a este artigo.
Disponibilidade de dados
Os dados de sequência gerados para este estudo estão disponíveis no Arquivo Europeu de Nucleotídeos sob o número de acesso PRJEB59008 .
Referências
Linstädter, J., Medved, I., Solich, M. & Weniger, G.-C. Processo de neolitização dentro do território alborano: modelos e possível impacto africano. Quat. Int. 274 , 219–232 (2012).
Martínez-Sánchez, RM, Vera-Rodríguez, JC, Pérez-Jordà, G., Peña-Chocarro, L. & Bokbot, Y. O início do Neolítico no noroeste de Marrocos. Quat. Internacional 470 , 485–496 (2018).
Zilhão, J. Navegação pré-histórica inicial no Mediterrâneo Ocidental: implicações para a transição neolítica na Península Ibérica e no Magreb. Isl. Archaeol. Orig. Seafaring East. Mediterr. 11 , 185–200 (2014).
Mulazzani, S. et al. O surgimento do Neolítico no Norte da África: um novo modelo para o Magreb Oriental. Quat. Int. 410 , 123–143 (2016).
Linstädter, J. A transição epipalaeolítica-neolítica na região mediterrânica do noroeste da África. Quartär. Anuário Internacional para Pesquisa da Idade do Gelo e da Idade da Pedra 55 , 41–62 (2008).
Fregel, R. et al. Genomas antigos do Norte da África evidenciam migrações pré-históricas para o Magreb, tanto do Levante quanto da Europa. Proc. Natl Acad. Sci. EUA 115 , 6774–6779 (2018).
Henn, BM et al. A ancestralidade genômica dos norte-africanos apoia migrações de volta à África. PLoS Genet. 8 , e1002397 (2012).
Arauna, LR et al. Migrações históricas recentes moldaram o pool genético de árabes e berberes no norte da África. Mol. Biol. Evol. 34 , 318–329 (2017).
Hublin, J.-J. et al. Novos fósseis de Jebel Irhoud, Marrocos e a origem pan-africana do Homo sapiens . Nature 546 , 289–292 (2017).
van de Loosdrecht, M. et al. Genomas do norte da África do Pleistoceno ligam populações humanas do Oriente Próximo e da África Subsaariana. Science 360 , 548–552 (2018).
Skoglund, P. et al. Origens e legado genético de agricultores e caçadores-coletores neolíticos na Europa. Science 336 , 466–469 (2012).
Omrak, A. et al. Evidências genômicas estabelecem a Anatólia como a fonte do pool genético neolítico europeu. 26 , 270–275 (2016).
Antonio, ML et al. Roma Antiga: uma encruzilhada genética da Europa e do Mediterrâneo. Science 366 , 708–714 (2019).
Yu, H. et al. Descontinuidades genômicas e alimentares durante o Mesolítico e Neolítico na Sicília. iScience 25 , 104244 (2022).
Günther, T. et al. Genomas antigos ligam os primeiros agricultores de Atapuerca, na Espanha, aos bascos modernos. Proc. Natl Acad. Sci. EUA 112 , 11917–11922 (2015).
Barnett, WK Cerâmica cardial e a transição agrícola na Europa Mediterrânea. Em Europe's First Farmers 93–116 (Cambridge Univ. Press, 2000).
Manen, C. et al. A transição neolítica no Mediterrâneo Ocidental: um processo de difusão complexo e não linear — o registro de radiocarbono revisitado. Radiocarbon 61 , 531–571 (2019).
Natali, E. & Forgia, V. O início do Neolítico no sul da Itália e na Sicília. Quat. Int. 470 , 253–269 (2018).
Guilaine, J. Uma visão pessoal da neolitização do Mediterrâneo Ocidental. Quat. Int. 470 , 211–225 (2018).
Bernabeu Auban, J. & Pardo-Gordó, S. A impressão na Península Ibérica: ¿Espejismo ou realidade? Uma reflexão a partir do binómio radiocarbono-cerâmico (2020)
Linstädter, J., Broich, M. & Weninger, B. Definindo o Neolítico Inicial do Rif Oriental, Marrocos – distribuição espacial, estrutura cronológica e impacto das mudanças ambientais. Quat. Int. 472 , 272–282 (2018).
Broodbank, C. & Lucarini, G. A dinâmica da África Mediterrânea, ca. 9600–1000 A.C .: uma síntese interpretativa de conhecidos e desconhecidos. J. Mediterr. Arqueol. https://doi.org/10.17863/CAM.49028 (2020).
Martínez-Sánchez, RM et al. Reflexões sobre o outro lado. Uma origem do sul da Península Ibérica para a primeira produção de cerâmica do norte de Marrocos? Open Archaeol. 7 , 1054–1065 (2021).
Huysecom, E. et al. O surgimento da cerâmica na África durante o décimo milênio cal a.C.: novas evidências de Ounjougou (Mali). Antiquity 83 , 905–917 (2009).
Garcea, EAA Forrageadores semipermanentes em ambientes semiáridos do Norte da África. World Archaeol. 38 , 197–219 (2006).
Dunne, J., Mercuri, AM, Evershed, RP, Bruni, S. & di Lernia, S. Primeira evidência direta de processamento de plantas em cerâmica pré-histórica do Saara. Nature Plants 3 , 16194 (2016).
Marshall, F. & Hildebrand, E. Gado antes das colheitas: os primórdios da produção de alimentos na África. J. World Prehist. 16 , 99–143 (2002).
Português Gautier, A. Em Secas, Alimentação e Cultura: Mudança Ecológica e Segurança Alimentar na Pré-História Tardia da África (ed. Hassan, FA) 195–207 (Springer US, 2002); https://doi.org/10.1007/0-306-47547-2_12 .
David, B. & McNiven, IJ Manual de Oxford de Arqueologia e Antropologia da Arte Rupestre (Oxford Univ. Press, 2018).
Smith, AB Origens e propagação do pastoralismo na África. Annu. Rev. Antropol. 21 , 125–141 (1992).
Olalde, I. et al. A história genômica da Península Ibérica nos últimos 8000 anos. Science 363 , 1230–1234 (2019).
Valdiosera, C. et al. Quatro milênios de pré-história biomolecular ibérica ilustram o impacto das migrações pré-históricas no extremo da Eurásia. Proc. Natl Acad. Sci. EUA 115 , 3428–3433 (2018).
Villalba-Mouco, V. et al. Sobrevivência da ancestralidade caçadora-coletora do Pleistoceno tardio na Península Ibérica. Curr. Biol. 29 , 1169–1177 (2019).
Mallick, S. et al. The Simons Genome Diversity Project: 300 genomas de 142 populações diversas. Nature 538 , 201–206 (2016).
Humphrey, LT et al. Evidências mais antigas de cáries e exploração de alimentos vegetais ricos em amido em caçadores-coletores do Pleistoceno do Marrocos. Proc. Natl Acad. Sci. EUA 111 , 954–959 (2014).
Turek, J. & Vintr, J. Neolit Maghrebu e vários novos radiocarbonos dados. Zivá Archeol. 18 , 10–15 (2016).
Ringbauer, H., Novembre, J. & Steinrücken, M. Parentalidade ao longo do tempo revelada por séries de homozigose em DNA antigo. Nat. Commun. 12 , 5425 (2021).
Morales, J. et al. A introdução de plantas domesticadas do sudoeste asiático no noroeste da África: uma contribuição arqueobotânica do Marrocos neolítico. Quat. Int. 412 , 96–109 (2016).
Martínez Sánchez, RM et al. Revisitando a transição epipalaeolítica-neolítica no extremo noroeste da África: os últimos resultados da sequência cronológica da caverna de Kaf Taht el-Ghar (Tétouan, Marrocos). Rev. 38 , 251–274 (2021).
Lipson, M. et al. Transectos paleogenômicos paralelos revelam história genética complexa dos primeiros agricultores europeus. Nature 551 , 368–372 (2017).
Reimer, PJ et al. A curva de calibração da idade do radiocarbono do hemisfério norte IntCal20 (0–55 cal kBP). Radiocarbon 62 , 725–757 (2020).
Martins, H. et al. Datação por radiocarbono do início do Neolítico na Península Ibérica: novos resultados, novos problemas. J. Mediterr. Archaeol. 28 , 105–131 (2015).
García Borja, P., Aura Tortosa, JE, Bernabeu Aubán, J. & Jordá Pardo, JF Novas perspectivas sobre a neolitização na cueva de Nerja (Málaga-Espanha): a cerâmica da sala do vestíbulo. Zephyrvs https://revistas.usal.es/uno/index.php/0514-7336/article/view/7979/8431 (2010).
Marcus, JH et al. História genética do Neolítico Médio até o presente na ilha mediterrânea da Sardenha. Nat. Commun. 11 , 939 (2020).
Mathieson, I. et al. A história genômica do sudeste da Europa. Nature 555 , 197–203 (2018).
Prendergast, ME et al. DNA antigo revela uma disseminação multietapa dos primeiros pastores na África subsaariana. Science 365 , eaaw6275 (2019).
Martínez Sánchez, RM et al. O Neolítico Médio da Faixa Atlântica Noroeste de Marrocos: novas evidências das Cavernas El-Khil (Tânger). Afr. Rev. 35 , 417–442 (2018).
Gilman, A. A pré-história posterior de Tânger, Marrocos (Escola Americana de Pesquisa Pré-histórica, Museu Peabody de Arqueologia e Etnologia, Universidade de Harvard, 1975).
Rodríguez-Varela, R. et al. Análises genômicas de restos humanos pré-conquista europeia das Ilhas Canárias revelam afinidade próxima com os modernos norte-africanos. 27 , 3396–3402 (2017).
Moots, HM et al. Uma história genética de continuidade e mobilidade no Mediterrâneo Central da Idade do Ferro. Pré-impressão em bioRxiv https://doi.org/10.1101/2022.03.13.483276 (2022).
Yang, DY, Eng, B., Waye, JS, Dudar, JC & Saunders, SR Extração de DNA aprimorada de ossos antigos usando colunas de centrifugação baseadas em sílica. 105 , 539–543 (1998).
Dabney, J. et al. Sequência completa do genoma mitocondrial de um urso-das-cavernas do Pleistoceno Médio reconstruído a partir de fragmentos de DNA ultracurtos. Proc. Natl Acad. Sci. EUA 110 , 15758–15763 (2013).
Svensson, E. et al. Genoma do crânio de Peştera Muierii mostra alta diversidade e baixa carga mutacional na Europa pré-glacial. Curr. Biol. 31 , 2973–2983 (2021).
Meyer, M. & Kircher, M. Preparação da biblioteca de sequenciamento Illumina para captura e sequenciamento de alvos altamente multiplexados. Cold Spring Harb. Protoc. 2010 , pdb-prot5448 (2010).
Günther, T. et al. Genômica populacional da Escandinávia Mesolítica: investigando rotas de migração pós-glaciais iniciais e adaptação a altas latitudes. PLoS Biol. 16 , e2003703 (2018).
Gansauge, M.-T. & Meyer, M. Preparação de biblioteca de DNA fita simples para sequenciamento de DNA antigo ou danificado. Nat. Protoc. 8 , 737–748 (2013).
Kircher, M. Análise de dados de sequenciamento de DNA antigo de alto rendimento. Métodos Mol. Biol . 840 , 197–228.
Schubert, M., Lindgreen, S. & Orlando, L. AdapterRemoval v2: corte rápido do adaptador, identificação e fusão de leitura. BMC Res. Notes 9 , 88 (2016).
Li, H. & Durbin, R. Alinhamento de leitura curta rápido e preciso com a transformada de Burrows–Wheeler. Bioinformatics 25 , 1754–1760 (2009).
Li, H. et al. O formato de alinhamento/mapa de sequência e SAMtools. Bioinformatics 25 , 2078–2079 (2009).
Fu, Q. et al. Uma escala de tempo revisada para a evolução humana com base em genomas mitocondriais antigos. Curr. Biol. 23 , 553–559 (2013).
Rasmussen, M. et al. Um genoma aborígene australiano revela dispersões humanas separadas na Ásia. Science 334 , 94–98 (2011).
Jun, G. et al. Detectando e estimando contaminação de amostras de DNA humano em dados de genótipos baseados em sequenciamento e array. Am. J. Hum. Genet. 91 , 839–848 (2012).
Fu, Q. et al. A história genética da Europa da era glacial. Nature 534 , 200–205 (2016).
Danecek, P. et al. O formato de chamada variante e VCFtools. Bioinformatics 27 , 2156–2158 (2011).
Weissensteiner, H. et al. HaploGrep 2: classificação de haplogrupos mitocondriais na era do sequenciamento de alto rendimento. Nucleic Acids Res. 44 , W58–W63 (2016).
Van Oven, M. PhyloTree Build 17: crescendo a árvore de DNA mitocondrial humana. Forensic Sci. Int. Genet. Suppl. Ser. 5 , e392–e394 (2015).
Monroy Kuhn, JM, Jakobsson, M. & Günther, T. Estimativa de relações genéticas de parentesco em populações pré-históricas. PLoS ONE 13 , e0195491 (2018).
Patterson, N., Price, AL & Reich, D. Estrutura populacional e autoanálise. PLoS Genet. 2 , e190 (2006).
Alexander, DH, Novembre, J. & Lange, K. Estimativa rápida baseada em modelo de ancestralidade em indivíduos não relacionados. Genome Res. 19 , 1655–1664 (2009).
Behr, AA, Liu, KZ, Liu-Fang, G., Nakka, P. & Ramachandran, S. Pong: análise rápida e visualização de clusters latentes em dados genéticos populacionais. Bioinformatics 32 , 2817–2823 (2016).
Skoglund, P. et al. Evidência genética para duas populações fundadoras das Américas. Nature 525 , 104–108 (2015).
Patterson, N. et al. Mistura antiga na história humana. Genética 192 , 1065–1093 (2012).
Haak, W. et al. Migração massiva da estepe foi uma fonte para línguas indo-europeias na Europa. Nature 522 , 207–211 (2015).
Harney, É. et al. DNA antigo do Israel Calcolítico revela o papel da mistura populacional na transformação cultural. Nat. Commun. 9 , 3336 (2018).
Loh, P.-R. et al. Inferindo histórias de mistura de populações humanas usando desequilíbrio de ligação. Genetics 193 , 1233–1254 (2013).
Narasimhan, VM et al. A formação de populações humanas na Ásia do Sul e Central. Science 365 , eaat7487 (2019).
Skoglund, P. et al. Diversidade genômica e mistura diferem para forrageadores e fazendeiros escandinavos da Idade da Pedra. Science 344 , 747–750 (2014).
DePristo, MA et al. Uma estrutura para descoberta de variação e genotipagem usando dados de sequenciamento de DNA de próxima geração. Nat. Genet. 43 , 491–498 (2011).
Purcell, S. et al. PLINK: um conjunto de ferramentas para associação de genoma completo e análises de ligação baseadas em população. Am. J. Hum. Genet. 81 , 559–575 (2007).
Schiffels, S. & Durbin, R. Inferindo o tamanho da população humana e o histórico de separação a partir de múltiplas sequências de genoma. Nat. Genet. 46 , 919–925 (2014).
Agradecimentos
Agradecemos a AR Munters pelo suporte em bioinformática e a RP Stjerna e F. Sánchez-Quinto pelas discussões úteis sobre análise de dados. O sequenciamento foi realizado na The National Genomics Infrastructure, Uppsala. Os cálculos e o manuseio de dados foram habilitados por recursos fornecidos pela Swedish National Infrastructure for Computing no Uppsala Multidisciplinary Center for Advanced Computational Science, parcialmente financiado pelo Swedish Research Council por meio do acordo de subsídio n.º 2018-05973. Este projeto foi apoiado pela Knut and Alice Wallenberg Foundation (para MJ), Vetenskapsrådet (subsídios n.º 2018-05537 e 2022-04642 para MJ e 2017-05267 para TG) e Ramón y Cajal (subsídio n.º RYC2018-025223-I para CV). A equipe arqueológica hispano-marroquina foi apoiada pelo Conselho Europeu de Pesquisa (nº ERC AdG 230561).
Financiamento
Financiamento de acesso aberto fornecido pela Universidade de Uppsala.
Informações do autor
Autores e Afiliações
Contribuições
CV, JCV-R. e MJ conceberam o estudo. RM-S., JCV-R., EI, RR-V., YB e CV selecionaram e amostraram material arqueológico. LGS realizou trabalho de laboratório de DNA. RM-S., JCV-R. e CV forneceram interpretações arqueológicas. LGS, TG e MJ analisaram dados genéticos. LGS, TG, RM-S., JCV-R., CV e MJ escreveram o artigo, com a contribuição de todos os autores.
Autores correspondentes
Declarações de ética
Interesses concorrentes
Os autores declaram não haver conflitos de interesse.
Revisão por pares
Informações de revisão por pares
A Nature agradece aos revisores anônimos por sua contribuição à revisão por pares deste trabalho.
Informações adicionais
Nota do editor A Springer Nature permanece neutra em relação a reivindicações jurisdicionais em mapas publicados e afiliações institucionais.
Informações suplementares
Informações suplementares
Uma descrição dos sítios arqueológicos do noroeste da África estudados e uma descrição detalhada dos métodos usados para análises, resultados complementares e discussões complementares para esses resultados.
Dados suplementares
Dados suplementares 1–15.
Direitos e permissões
Acesso aberto Este artigo está licenciado sob uma Licença Internacional Creative Commons Atribuição 4.0, que permite uso, compartilhamento, adaptação, distribuição e reprodução em qualquer meio ou formato, desde que você dê o devido crédito ao(s) autor(es) original(ais) e à fonte, forneça um link para a licença Creative Commons e indique se foram feitas alterações. As imagens ou outros materiais de terceiros neste artigo estão incluídos na licença Creative Commons do artigo, a menos que indicado de outra forma em uma linha de crédito para o material. Se o material não estiver incluído na licença Creative Commons do artigo e seu uso pretendido não for permitido por regulamentação estatutária ou exceder o uso permitido, você precisará obter permissão diretamente do detentor dos direitos autorais. Para visualizar uma cópia desta licença, visite http://creativecommons.org/licenses/by/4.0/ .
Sobre este artigo
Citar este artigo
Simões, LG, Günther, T., Martínez-Sánchez, RM et al. Neolítico do noroeste da África iniciado por migrantes da Península Ibérica e do Levante. Nature 618 , 550–556 (2023). https://doi.org/10.1038/s41586-023-06166-6
Recebido
Aceito
Publicado
Data de emissão
DOIhttps://doi.org/10.1038/s41586-023-06166-6
Este artigo é citado por
A modelagem da história demográfica dos genomas humanos do Norte da África aponta para uma recente divergência de divisão suave entre populações
Biologia do Genoma (2024)
Compreendendo a heterogeneidade genômica dos Imazighen do Norte da África: de perspectivas amplas a microgeográficas
Relatórios Científicos (2024)
Melhorando as práticas de arquivamento de dados em genômica antiga
Dados científicos (2024)
Evidência isotópica de alta dependência de alimentos vegetais entre caçadores-coletores da Idade da Pedra Tardia em Taforalt, Marrocos
Ecologia da Natureza e Evolução (2024)
Estrutura genética humana no noroeste da França fornece novos insights sobre a demografia histórica da Europa Ocidental
Comunicações da Natureza (2024)

Nenhum comentário:
Postar um comentário
Observação: somente um membro deste blog pode postar um comentário.