Genoma de um caçador-coletor do Holoceno médio de Wallacea
Natureza volume 596 , Páginas 543–547 ( 2021 )
Resumo
Muito permanece desconhecido sobre a história da população dos primeiros humanos modernos no sudeste da Ásia, onde o registro arqueológico é esparso e o clima tropical é hostil à preservação do DNA humano antigo 1 .
Até agora, apenas dois genomas humanos pré-neolíticos de baixa cobertura foram sequenciados nesta região. Ambos são de locais de caçadores-coletores de Hòabìnhian no continente: Pha Faen em Laos, datado de 7939-7751 anos calibrados antes do presente (ano cal bp; presente tomado como ad 1950), e Gua Cha na Malásia (4,4-4,2 kyr cal bp ) 1.
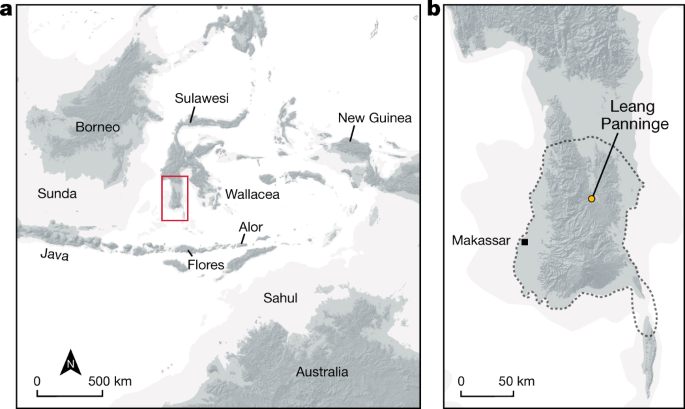
Aqui relatamos, até onde sabemos, o primeiro genoma humano antigo de Wallacea, a zona da ilha oceânica entre a plataforma Sunda (compreendendo o sudeste da Ásia continental e as ilhas continentais do oeste da Indonésia) e o Pleistoceno Sahul (Austrália-Nova Guiné).
Extraímos DNA do osso petroso de uma jovem caçadora-coletora enterrada 7,3-7,2 kyr cal bp na caverna de calcário de Leang Panninge 2 em South Sulawesi, Indonésia. Análises genéticas mostram que esta forrageira pré-neolítica, que está associada ao tecnocomplexo 'Toalean' 3 , 4, compartilha a maioria dos desvios genéticos e semelhanças morfológicas com os grupos atuais da Papuásia e da Austrália indígena, mas representa uma linhagem humana divergente anteriormente desconhecida que se ramificou na época da divisão entre essas populações há aproximadamente 37.000 anos 5 . Também descrevemos ancestrais denisovanos e asiáticos profundos no genoma de Leang Panninge e inferimos seu deslocamento em grande escala da região hoje.
Principal
Os humanos modernos cruzaram a Wallacea (Fig. 1a ) para Sahul 5 , 6 , 7 , 8 pelo menos 50 mil anos atrás (kya) 9 , e possivelmente por até 65 kya 10 . Atualmente, no entanto, as primeiras evidências arqueológicas de nossa espécie em Wallacea datam de pelo menos 45,5 kya para a arte figurativa em Sulawesi 11 e 47-43 kyr cal bp para uma mudança comportamental em Liang Bua (Flores, Indonésia) 12 . Os mais antigos vestígios do esqueleto do Homo sapiens datam de 13 kya 13 . A rota que os humanos modernos usaram para entrar em Sahul não é conhecida 14. Os modelos demográficos inferem uma população dividida entre os ancestrais dos grupos da Oceania e da Eurásia de aproximadamente 58 kya, enquanto os grupos da Papua e da Austrália aborígene se separaram em torno de 37 kya 5 . Nesse intervalo de tempo, os humanos modernos se misturaram várias vezes com grupos relacionados aos Denisovans 15 , 16 , 17 , 18 , 19 , 20 , 21 , 22 , 23 e potencialmente outros hominíneos desconhecidos 24 , 25 .
A ancestralidade genética das duas forrageadoras associadas a Hòabìnhian de Pha Faen e Gua Cha 1mostra a maior semelhança com os povos andamaneses modernos. Esses povos antigos e atuais carecem de quantidades substanciais de ancestralidade relacionada a Denisovan, sugerindo que a linhagem associada a Hòabìnhian e relacionada a Onge divergiu antes dos principais eventos de introgressão humana arcaica 1 . Os indivíduos atuais da Wallacean carregam proporções maiores de ancestralidade relacionada a Denisovan, mas em frequências substancialmente mais baixas do que no caso de indivíduos Papuanos e Indígenas australianos 20 . Isso provavelmente se deve à mistura com os fazendeiros neolíticos do Leste Asiático ('povos austronésios') que chegaram a Wallacea por volta de 4 kya 20 , 26 .
O enterro Toalean de Leang Panninge
As assembleias arqueológicas mais distintas associadas aos caçadores-coletores do Holoceno em Wallacea pertencem ao tecnocomplexo Toalean (8-1,5 kya) 3 , 4 , 27 , 28 . Encontrado apenas em uma área de 10.000 km 2 de South Sulawesi 3 (Fig. 1b ), assembléias culturais Toalean são geralmente caracterizadas por micrólitos apoiados e pequenos projéteis de pedra ('pontos de Maros') 4 (Dados Estendidos Fig. 1a-c ). Em 2015, as escavações em Leang Panninge no distrito de Mallawa de Maros, Sulawesi do Sul (Fig. 1b ), descobriram o primeiro sepultamento humano relativamente completo de um contexto Toalean seguro (Figs de dados estendidos.1 - 5 , Informações suplementares ). O indivíduo foi enterrado em uma posição fletida 29 em um rico estrato acerâmico de Toalean. Exposto a uma profundidade de cerca de 190 cm, o sepultamento tem uma idade inferida de 7,3-7,2 kyr cal bp obtida a partir da datação por 14 C de um Canarium sp. semente (Dados Estendidos Figs. 2 , 3 , Tabela Suplementar 1 ). Caracteres morfológicos indicam que esta forrageira Toaleana era uma fêmea de 17-18 anos com uma afinidade amplamente australo-melanésia, embora a morfologia não caia fora da faixa de variação recente do sudeste asiático ( informações suplementares ).
Análise genômica
Extraímos DNA antigo do pó de osso obtido da porção petrosa do osso temporal do indivíduo Leang Panninge. Após a preparação da biblioteca, usamos uma abordagem de captura de hibridização de DNA para enriquecer cerca de 3 milhões de polimorfismos de nucleotídeo único (SNPs) em todo o genoma humano (1240K e painéis de captura de mistura arcaica 30 ), bem como para todo o genoma mitocondrial (captura de mtDNA 31 ) . Recuperamos 263.207 SNPs no painel 1240K, 299.047 SNPs no painel de mistura arcaico e a sequência de mtDNA quase completa. A autenticidade do DNA antigo analisado foi confirmada pelo comprimento médio curto do fragmento, padrões elevados de danos nas extremidades da molécula e estimativas baixas de contaminação autossômica e de mtDNA (Fig. 1 Suplementar) Confirmamos que o indivíduo era geneticamente do sexo feminino. A análise dos polimorfismos presentes na sequência reconstruída do mtDNA sugere um posicionamento profundamente divergente dentro do haplogrupo M do mtDNA (Tabela suplementar 17 , Fig. 2 suplementar ).
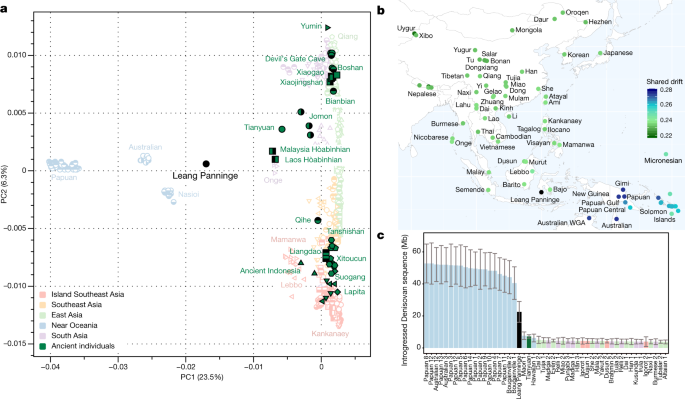
Iniciamos nossa investigação genômica por análises de componentes principais (PCAs), comparando o genoma de Leang Panninge com indivíduos atuais do Leste Asiático, sudeste da Ásia e perto da Oceania (incluindo Austrália Indígena, Papua Nova Guiné e Bougainville) genotipados no painel SNP de Origens Humanas 18 , 32 , 33 , 34 . O genoma recém-gerado e os genomas relevantes publicados de indivíduos antigos da Eurásia oriental foram então projetados no PCA 1 , 34 , 35 , 36 , 37 , 38. Leang Panninge cai no espaço do PCA não ocupado por nenhum indivíduo atual ou antigo, mas está amplamente localizado entre os povos indígenas australianos e os Onge (Fig. 2a , Dados estendidos, Fig. 6 ). As estatísticas F 3 33 da forma f 3 (Mbuti; Leang Panninge, X), em que X é substituído pelos grupos atuais da Ásia-Pacífico, indicaram que o novo genoma compartilha a maior parte da deriva genética com indivíduos próximos da Oceania (Fig. 2b ) . Confirmamos esses resultados com as estatísticas f 4 33, sugerindo afinidade semelhante de indivíduos Leang Panninge e Papuan para indivíduos asiáticos de hoje, apesar dos grupos da Quase Oceania formarem um clado com a exclusão de Leang Panninge (Dados Estendidos Fig. 7a, b ). Todos os grupos atuais da região, com exceção do Mamanwa e do Lebbo 26 , carregam apenas uma pequena contribuição de ancestralidade papua (Fig. Suplementar 4 ).
a , PCA calculado em indivíduos atuais da Eurásia oriental e perto da Oceania, projetando os principais indivíduos antigos da região 1 , 34 , 35 , 36 , 37 , 38 . b , Deriva genética compartilhada dos grupos atuais com o indivíduo Leang Panninge, calculada usando f 3 (Mbuti; Leang Panninge, X) mapeado na posição geográfica do grupo testado. WGA, amplificação do genoma completo. c, A quantidade de sequência Denisovan introgressada em fragmentos maiores que 0,05 cM em indivíduos atuais (Projeto de Diversidade do Genoma Simons) e maiores que 0,2 cM em indivíduos antigos (medido com Admixfrog). Cada barra representa a estimativa média posterior de um único genoma e os bigodes indicam 2 sd (estimado a partir de 200 amostras da decodificação posterior).
Para investigar a presença e distribuição de contribuições genéticas atribuíveis a grupos 39 relacionados a Denisovan , calculamos a estatística f 4 (Mbuti, Denisova; Leang Panninge, X), em que X são grupos da atual Ilha do Sudeste Asiático, Perto da Oceania e o Andamans, bem como antigos indivíduos do Pacífico Asiático 1 , 37 , 38 . Valores positivos calculados para grupos próximos à Oceânia sugerem maiores proporções de ancestralidade relacionada a Denisovan do que o indivíduo Leang Panninge ( z- escores de> 3,19), enquanto Onge e os demais indivíduos antigos retornaram valores negativos, indicando uma proporção menor de ancestralidade relacionada a Denisovan (Dados estendidos Fig. 7c, Suplementar Fig. 3 ). Também calculamos as estatísticas de f 4 -ratio para estimar a proporção Denisovan usando SNPs do painel de captura de 1240K e indivíduos Han do Leste Asiático como linha de base 18 . Confirmámos que indígena australiana e indivíduos Papuan transportar uma quantidade semelhante de ascendência Denisovan (aproximadamente 2,9%) 18 , 21 , 40 , ao passo que o indivíduo Leang Panninge tem um valor inferior de cerca de 2,2 ± 0,5% (Tabelas complementares 18 - 20 ). A proporção de mistura Denisovan no indivíduo Leang Panninge é maior do que nos indivíduos Hòabìnhian de Pha Faen e Gua Cha 1, sugerindo que grupos ancestrais de caçadores-coletores de Wallacea e Sunda estavam envolvidos em diferentes eventos de introgressão com hominídeos arcaicos. Além disso, realizamos estatísticas D em um conjunto de SNPs projetados para medir a contribuição da ancestralidade arcaica em humanos modernos (painel de captura de mistura arcaica). O indivíduo Leang Panninge compartilha menos alelos relacionados a Denisovan com indivíduos da Papua, mas tem mais alelos desse tipo do que a maioria dos grupos testados, incluindo o indivíduo Tianyuan do Pleistoceno Superior da China 38 . Neanderthal alelo partilha é semelhante em todos os grupos testados actual não africanos (Tabelas complementares 21 - 23 ). Finalmente, executamos o Admixfrog 41no conjunto de SNPs de mistura arcaica e mediu 22,4 Mb (± 1,9 Mb) de ancestralidade relacionada a Denisovan em 33 fragmentos distribuídos pelo genoma Leang Panninge. Essa contribuição é responsável por cerca de metade do que é encontrado em grupos de Papua, mas há uma correlação significativa entre os fragmentos Denisovan no genoma de Leang Panninge e aqueles nos grupos atuais da região próxima à Oceânia, sugerindo eventos de introgressão compartilhados (Fig. 2c , Dados estendidos Fig. 8 , Fig. 5 complementar ).
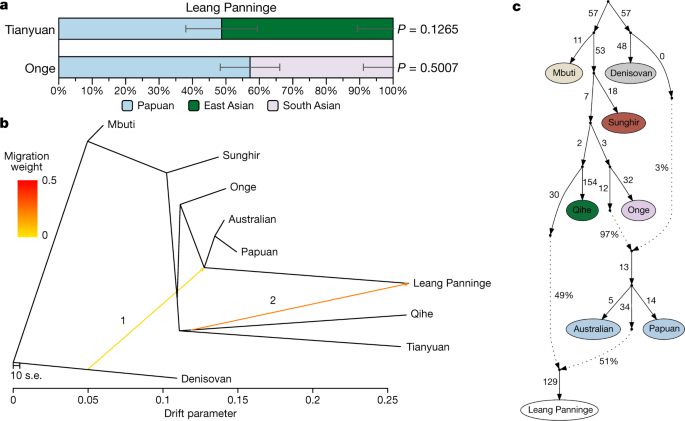
Para investigar se o deslocamento aparente do PCA de Leang Panninge para longe dos grupos da Oceania é devido apenas à deriva genética, realizamos um gráfico de escala multidimensional com base nas semelhanças genéticas medidas como 1 - f 3 (Mbuti; Leang Panninge, X). O posicionamento de escala multidimensional do indivíduo Leang Panninge recapitula o PCA com um posicionamento intermediário entre indivíduos da Papua e Ásia (dados estendidos Fig. 9 ). Em seguida, usamos estatísticas f 4 e qpWave 33 para testar formalmente a presença de fontes genéticas adicionais em Leang Panninge além da ancestralidade papua. Isso identificou uma afinidade marginal para os genomas asiáticos antigos (dados estendidos Fig. 7d), e um mínimo de duas correntes de ancestralidade quando Denisova 37 e / ou antigos grupos asiáticos 1 , 37 , 38 foram incluídos nos grupos de referência qpWave (Tabela Suplementar 24 ). Com base nesses resultados, usamos qpAdm 33 para identificar fontes potenciais para um ancestral relacionado à Ásia no genoma ao lado do componente relacionado à Papua (Tabela Suplementar 25 ). Usando uma abordagem rotativa entre diferentes grupos asiáticos 1 , 37 , 38 , fomos capazes de modelar o genoma de Leang Panninge como uma mistura entre Papuan e Tianyuan (51 ± 11%) ou Onge (43 ± 9%) (Fig. 3a, Tabela complementar 26 ). Explorações adicionais com gráficos de mistura construídos em qpGraph 33 e TreeMix 42 , incluindo grupos atuais e indivíduos antigos relevantes 37 , 38 , 40 , 43 , forneceram evidências para a presença de ancestrais asiáticos profundos (Fig. 3b, c , Figs. 6 Suplementares - 11) Em TreeMix, a primeira borda de mistura representa a introgressão arcaica de grupos relacionados a Denisovan no ancestral comum de Leang Panninge e dos povos atuais da região próxima da Oceania. Isto é seguido por um fluxo gênico relacionado ao Leste Asiático em Leang Panninge partindo basicamente da linhagem Qihe, um genoma neolítico inicial do sudeste da China 37 (Fig. 3b , Dados Estendidos Fig. 10 , Fig. 6 Suplementar ). A análise qpGraph confirmou este padrão de ramificação, com o indivíduo Leang Panninge ramificando-se do clado Próximo da Oceania após o fluxo gênico de Denisovan, embora com a topologia mais suportada indicando cerca de 50% de um componente basal do Leste Asiático contribuindo para o genoma de Leang Panninge (Fig. . 3c, Figs suplementares. 7 - 11 ).
a , Proporções de mistura modelando Leang Panninge como uma combinação de grupos Papuan 49 e Tianyuan 38 ou Onge 49 conforme estimado por qpAdm 33 usando Mbuti, Denisovan 39 , Kostenki 14 (ref. 50 ) e indivíduos asiáticos antigos 1 , 37 como grupos de referência rotativos ( Tabela complementar 26 ). As barras de erro denotam erros padrão calculados com block jacknife no software qpAdm. b , c , Gráficos de mistura colocando Leang Panninge no galho com o atual clado Próximo da Oceania 41e mostrando a mistura com uma ascendência-relacionada asiática profunda em TreeMix 42 ( b ) (Dados estendidos Fig. 10 , complementar Fig. 6 ) e qpGraph ( c ) (pior z -Score de -2,194;. Suplementar Figs 7 - 11 ) 33 , 37 , 38 , 39 , 43 . Em b , '1' e '2' referem-se à ordem em que o software TreeMix adicionou 'eventos de migração' (indicados pelas setas) ao gráfico. Ao plotar os resultados qpGraph ( c ), as setas pontilhadas indicam bordas de mistura.
Discussão
As análises de todo o genoma do indivíduo Leang Panninge mostram que a maior parte da deriva genética é compartilhada com grupos atuais da Nova Guiné e da Austrália aborígene (Fig. 2b , Dados estendidos Fig. 7a ). No entanto, este genoma associado a Toalean representa um perfil de ancestralidade não descrito anteriormente, que se ramificou após linhagens relacionadas a Onge e Hòabìnhian, mas na época em que os grupos da Papua e da Austrália indígena se dividiram (Fig. 3b, c , Dados estendidos Fig. 8 , Figs. 6 - 9 complementares ). É possível que este indivíduo Toaleano carregue uma ancestralidade local que estava presente em Sulawesi antes do povoamento inicial de Sahul pelo menos 50 kya 9, embora não se saiba se essa população produziu a arte rupestre do Pleistoceno Superior no sul da ilha 11 , 44 , 45 .
O indivíduo Toalean transporta ascendência Denisovan-relacionada substancial, provavelmente partilha o evento mistura arcaico com atuais grupos Perto Oceanian (Fig. 2c , Extended Data Figs. 7 C, 10 , complementar a Fig. 5 , Tabelas complementares 21 - 23 ). Isso fornece um forte suporte para o principal fluxo gênico relacionado a Denisovan que acontecia antes que os humanos modernos chegassem a Sahul, tornando Wallacea e Sunda locais igualmente prováveis para esse evento arcaico de introgressão. No entanto, previamente publicados genomas de caçadores-coletores de Sunda realizar ascendência quase nenhuma Denisovan-relacionada (quadros suplementares 18 - 20), sugerindo uma propagação relacionada ao Hòabìnhian no sudeste da Ásia após o fluxo gênico mencionado ou que Wallacea era de fato o ponto de encontro crucial entre humanos arcaicos e modernos. A aparente presença de uma população estabelecida de longa data de hominíneos arcaicos no sudoeste de Sulawesi 46 fornece uma possível fonte para o evento de introgressão. Dois estudos anteriores sugeriram que duas linhagens profundamente divergentes de Denisovan se misturaram aos ancestrais de indivíduos de Papua 17 , 22 , mas nossos dados genômicos atualmente não têm resolução suficiente para distinguir entre um ou vários pulsos de introgressão.
A menor quantidade de ancestralidade Denisovan no indivíduo Leang Panninge do que em indivíduos Papuanos e Indígenas australianos pode resultar de: (1) uma mistura adicional com ancestralidade Denisovana nos ancestrais comuns de grupos da Quase Oceania, ou (2) uma diluição do Denisovan- ancestralidade relacionada no genoma de Leang Panninge por meio da mistura com linhagens que carregam menos ou nenhuma ancestralidade. Nossas análises baseadas em frequência de alelo não suportam o primeiro cenário (Fig. Suplementar 11), mas favorecem o último. A escassez de genomas pré-neolíticos em toda a Ásia nos impede de definir a fonte exata e as proporções de mistura desse evento de fluxo gênico. É digno de nota, no entanto, que apesar das árvores populacionais reconstruídas (TreeMix e qpGraph) sugerirem uma influência genética no Holoceno médio Sulawesi do leste da Ásia continental, nossa modelagem qpAdm não pode descartar uma contribuição do sudeste asiático de um grupo relacionado aos atuais povos andamaneses (Fig. 3 , Figs. 6 - 11 suplementares, Tabela 26 suplementar ). Isso é consistente com um estudo recente que descreve misturas generalizadas em toda a Ásia entre populações antigas relacionadas a Onge e Tianyuan 47. No entanto, a presença desse tipo de ancestralidade em uma forrageira do Holoceno médio de Wallacea sugere que a mistura relacionada à Ásia poderia ter ocorrido muito antes da expansão das sociedades austronésias na região.
Não foi possível detectar evidências da ancestralidade Leang Panninge em nenhum dos grupos atuais testados (Fig. 4 suplementar ). Isso pode ser devido à proporção limitada geral de ancestrais parentes da Quase Oceania em Wallacea ou à descontinuidade genética em grande escala entre os primeiros caçadores-coletores e os grupos modernos. O último cenário sugeriria que qualquer sinal genético relacionado ao indivíduo Leang Panninge foi obscurecido por processos demográficos posteriores, incluindo a expansão austronésica 1 , 20 , 26 , 48. Dados genéticos de cobertura mais alta das populações atuais em Sulawesi e genomas antigos toaleanos adicionais são necessários para investigar mais a fundo esse perfil de ancestralidade único e a diversidade genética de caçadores-coletores de Wallacea de forma mais geral.
Métodos
Arqueologia
Leang Panninge foi identificado pela primeira vez como um local com alto potencial arqueológico durante uma pesquisa de 2013 por AB, BH e BB. Desde então, a caverna de calcário tem sido o foco de várias escavações realizadas por diferentes equipes (Dados estendidos Fig. 2 , Informações complementares ) . O primeiro, a escavação de um poço de teste de 1 m 2 (rotulado como TP1), foi conduzido por Balai Arkeologi Sulawesi Selatan (Balar Sulsel) em 2014 (ref. 2 ). Isso foi seguido em 2015 por três escavações (de 1 m 2poços de teste MLP / A.1 '/ 13, MLP / A.2' / 13 e MLP / B.3 ′ / 1) espalhados pela caverna, incluindo um fora da boca, por Balai Pelestarian Cagar Budaya (BPCB) Sulawesi Selatan. O objetivo dessas escavações foi avaliar a importância do local (o relatório resultante concluiu que ele constava do banco de dados do patrimônio cultural da BPCB). Mais tarde, no mesmo ano, Balar Sulsel retornou em colaboração com Universitas Hasanuddin (UNHAS) e Universiti Sains Malaysia para escavar uma trincheira na extremidade norte da caverna (unidades contíguas S8T5 e S8T6) e na área do piso central (unidades contíguas S16T6 e S17T6 ) Parte de um crânio humano em um contexto de sepultamento foi descoberta no final desta escavação. Devido a limitações financeiras e de tempo, o cemitério foi coberto com folhas de plástico e a trincheira preenchida para protegê-la para as escavações subsequentes.Balar Sulsel continuou a trabalhar na caverna em 2016 (unidade de escavação S30T9). Os quadrados S16T6 e S17T6 foram reabertos em 2018 para recuperar o esqueleto humano encontrado na base da escavação de 2015. Em 2019, esta trincheira foi estendida para a parte de trás da caverna (formando unidades contíguas S16T7 e S17T7) por uma equipe conjunta da Indonésia-Austrália da Griffith University e Pusat Penelitian Arkeologi Nasional (ARKENAS), UNHAS e Balar Sulsel. Os principais objetivos do trabalho de campo de 2019 eram avaliar esses depósitos adjacentes para outros restos de esqueletos humanos, bem como obter amostras de carbono vegetal e outros materiais com os quais determinar com mais precisão a idade do sepultamento humano exposto pela primeira vez em 2015.As investigações de 2019 foram conduzidas sob uma licença de pesquisa estrangeira emitida pelo Ministério de Pesquisa e Tecnologia do Estado da Indonésia (licença nº: 154 / SIP / FRP / E5 / Dit.KI / VII / 2017). As investigações anteriores lideradas pela Indonésia em Leang Panninge foram realizadas sob os termos de notificações formais para conduzir pesquisas (Surat Pemberitahuan ou Surat Penyampaian ) apresentado às autoridades governamentais locais em vários níveis de administração, da regência / município ( kabupaten ) ao distrito ( kecamatan ) à aldeia ( desa ).
As escavações de 2015 foram conduzidas em espetos arbitrários de 10 cm de espessura e peneirados por via úmida através de uma malha de 3 mm, a uma profundidade de aproximadamente 190 cm, ponto em que os restos de esqueleto humano foram encontrados no canto sudoeste da escavação (espetos 19 e 20, camada 4). Em 2019, os depósitos foram escavados com o mesmo método, mas desta vez em espetos de 5 cm; consequentemente, os nomes de cuspe em S16T7 ou S17T7 se originam de uma profundidade metade daquela de um cuspe com o mesmo número em S16T6 ou S17T6 (por exemplo, cuspe 18 tem 170-180 cm no primeiro caso ou 85-90 cm no último; ver Dados estendidos Fig. 3) Como observado, o esqueleto foi recuperado do local em 2018. Devido à fragilidade dos restos do esqueleto, os elementos visíveis que compreendem o crânio e as áreas pélvicas foram primeiro consolidados com uma solução de endurecimento (resina acrílica Paraloid B72) e depois removidos do depósito. bloco (dados estendidos Fig. 4e) O 'bloqueio craniano' e o 'bloqueio pélvico' foram ambos submetidos à tomografia computadorizada (TC) em hospital de Makassar, Indonésia (Balai Pengamanan Fasilitas Kesehatan Makassar), utilizando os seguintes parâmetros tomográficos: colimação: 0,625 mm; pitch: 1 / 0,625; miliamperes e quilovolts: deixado sozinho; kernel: osso; reconstrução retro: interslice de 0,3 mm. Após a tomografia computadorizada, os dois blocos de sedimentos foram escavados em condições de laboratório para remover os restos do esqueleto. O bloco de sedimentos contendo o crânio consistia em uma porção intacta do aterro original localizado imediatamente adjacente e abaixo do crânio, mandíbula e elementos dentários. A parte mais espessa deste bloco de sedimentos mede aproximadamente 100 mm. Durante a escavação do 'bloco de crânio',recuperamos a porção petrosa direita do osso temporal humano e depois a submetemos para análise de DNA no Instituto Max Planck de Ciência da História Humana (MPI-SHH) em Jena, Alemanha. Também recuperamos artefatos de pedra e restos faunísticos, bem como uma queimadaCanarium sp. semente localizada a poucos centímetros do aglomerado principal de ossos cranianos ( Informações Suplementares ). Esta semente rendeu um acelerador de espectrometria de massa (AMS) 14 C idade de 7264-7165 anos cal bp (Wk-48639) (Tabela Suplementar 1 ).
Documentação morfológica
Os restos mortais humanos de Leang Panninge (Tabela Complementar 2) estão armazenados no Laboratório de Arqueologia do Departamento de Arqueologia (Departemen Arkeologi Fakultas Ilmu Budaya) em UNHAS, Makassar, South Sulawesi, Indonésia. Em 2019, DB reconstruiu e descreveu os restos mortais sob a supervisão de MN e as junções IS foram feitas usando o Tarzan's Grip junto com a plasticina para ossos perdidos. Pesos esqueléticos foram medidos com uma balança com precisão de 1 g. As medições foram feitas com uma pinça eletrônica Kincrome com precisão de 0,01 mm (geralmente arredondada para o décimo de milímetro mais próximo). Os dentes foram medidos em seus diâmetros mesiodistal e vestíbulo-lingual máximos e também esses diâmetros na junção amelocementária. As características morfológicas dentais registradas foram aquelas do sistema da Arizona State University 51, incluindo referência a placas padrão ilustradas nessa obra, e na ref. 52 para fotografias de algumas outras placas padrão. Outras fontes para registrar medições e características anatômicas são descritas nas Informações Suplementares .
Processamento de DNA antigo
Amostragem, extração, preparação de biblioteca e indexação foram realizadas em uma sala limpa dedicada para DNA antigo no MPI-SHH. Obtivemos pó de osso da pars petrosa direita do indivíduo Leang Panninge, cortando ao longo da margem superior e perfurando próximo à cóclea 53 . O DNA foi extraído usando uma versão modificada do antigo protocolo de DNA descrito na ref. 54 . A partir do extrato, construímos uma biblioteca de fita dupla após tratamento parcial com uracila-DNA glicosilase 55 para reduzir as transições C> T para os primeiros dois pares de bases e uma biblioteca de fita simples em um sistema automatizado de manipulação de líquidos 56 . Após dupla indexação com combinações de índice exclusivas 57, as bibliotecas foram sequenciadas rapidamente para uma profundidade de aproximadamente 4 milhões de leituras em uma Illumina HiSeq 4000 no MPI-SHH usando uma configuração de leitura única de 75 bp para avaliações de qualidade iniciais.
Após a amplificação adicional, as bibliotecas foram hibridizadas em solução para enriquecer o mitogenoma completo (captura de mtDNA 31 ) e duas vezes para um conjunto direcionado de 2.986.592 SNPs em todo o genoma humano (duas rodadas de '1240K' e 'ancestralidade arcaica' 30 capturas) . Os produtos de captura foram então sequenciados em uma Illumina HiSeq 4000 no MPI-SHH usando uma configuração de leitura única de 75 bp. Após AdapterRemoval conforme implementado no EAGER v.1.92.56 58 , as leituras enriquecidas com mtDNA foram alinhadas ao genoma de referência mitocondrial (rCRS) e as leituras das capturas de todo o genoma para o genoma de referência humano (hg19) usando um filtro de qualidade de mapeamento de 30 para o mapeador circular v.1.93.5 e BWA 59alinhador, respectivamente. As duplicatas foram removidas com DeDup v.0.12.2 ( https://github.com/apeltzer/DeDup ). A contaminação das sequências de fita simples foi avaliada com AuthentiCT v.1.0 60 .
Reconstruímos a sequência de consenso mitocondrial e estimamos a contaminação mitocondrial em 2 ± 1% usando schmutzi 61 . O haplogrupo mitocondrial foi verificado com Haplofind 62 . Após mesclar com os dados publicados usando o mafft v.7.305 63 , construímos uma árvore de parcimônia máxima no MEGA X 64 . Com base no padrão de incorporação incorreta determinado pelo mapDamage v.2.0.9 conforme implementado no EAGER v.1.92.56 58 , aparamos 2 bp dos dados da biblioteca de fita dupla capturados em 1240K e genotipamos as sequências aparadas e não aparadas individualmente para o Painel de 1240K usando samtools v.1.3 ( https://github.com/samtools/samtools ) e pileupCaller v.1.4.0.2 (https://github.com/stschiff/sequenceTools ), que chama aleatoriamente um alelo por site SNP. Os genótipos não aparados e aparados foram então combinados, retendo apenas transversões do genótipo não aparado e transições do genótipo aparado para maximizar a informação das extremidades aparadas. Em vez disso, os dados da biblioteca de fita simples foram genotipados usando o modo de fita simples de pileupCaller e os dois genótipos mesclados usando um script personalizado. A cobertura resultante foi adequada para análises de genética populacional com 263.207 SNPs no 1240K e 135.432 SNPs no painel de origens humanas (HO). Também genotipamos dados de fita simples e dupla individualmente após a filtragem com PMDtools v.0.6 65 .
Análises genéticas populacionais
PCAs foram realizados usando smartpca com shrinkmode e lsqmode habilitado 66 , calculando os componentes principais dos indivíduos atuais do leste e sudeste da Ásia e da Oceania genotipados no painel Origens Humanas 18 , 32 , 33 , 34 e projetando todos os genomas antigos.
Todas as estatísticas f 3 ef 4 foram calculadas usando qp3pop v.420 (endogamia: SIM) e qpDstat v.721, respectivamente 33 . Para estatísticas f 3 , usamos grupos do Leste e Sudeste Asiático e da Oceania do conjunto de dados Origens Humanas para incluir populações mais comparativas, enquanto para estatísticas f 4 , usamos um conjunto de dados mais restrito contendo dados do Projeto Simons Genome Diversity (SGDP 49 ) genotipado no painel 1240K para maximizar o número de SNPs sobrepostos com o indivíduo Leang Panninge. Os resultados de f 3-estatísticas foram plotadas na localização geográfica do grupo de teste usando ggplot2 v.3.3.3 no RStudio v.1.2.1335. Para investigar a proporção de ancestralidade relacionada a Denisovan (α), calculamos a estatística f 4 -ratio usando qpF4Ratio 18 , 20 , 33 , admixfrog 41 e D- statistics 33 com um script personalizado. Usando qpWave 33 , investigamos se poderíamos distinguir entre a ancestralidade parecida com a da Papuásia presente no indivíduo Leang Panninge em comparação com os indivíduos da Papua atual. As proporções de mistura foram estimadas com qpAdm (allsnps: YES) 33 . Após a conversão do arquivo com PLINK v.1.9 67, executamos TreeMix v.1.12 42 definindo o genoma Denisovan 39 como a raiz e utilizando os parâmetros -k 150 e -global. Os modelos foram plotados usando RColorBrewer v.1.1.2 no RStudio v.1.2.1335 e o ajuste foi avaliado por inspeção residual após cada borda de migração adicional ter sido adicionada. Gráficos de mistura com qpGraph 33foram construídos (outpop: NULL, useallsnps: YES, blgsize: 0,05, forcezmode: YES, lsqmode: YES, diag: 0,0001, bigiter: 15, hires: YES, lambdascale: 1, initmix: 1,000, endogamia: YES) adicionando um grupo após o outro, passando de humanos arcaicos sobre grupos atuais para amostras antigas e testando todas as misturas unilaterais e bidirecionais possíveis usando um script personalizado. A decisão sobre qual modelo foi escolhido para progredir com a adição de outro grupo foi feita com base na pior pontuação z mais baixa calculada para cada árvore proposta. A estimativa do tempo de mistura foi calculada com DATES v.753 68 .
Resumo do relatório
Mais informações sobre o desenho da pesquisa estão disponíveis no Nature Research Reporting Summary, vinculado a este artigo.
Disponibilidade de dados
As sequências brutas e alinhadas estão disponíveis no Arquivo Europeu de Nucleotídeos com o número de acesso PRJEB43715 .
Disponibilidade de código
Os scripts personalizados usados neste estudo estão disponíveis mediante solicitação aos autores correspondentes.
Referências
- 1
McColl, H. et al. O povoamento pré-histórico do Sudeste Asiático. Science 361 , 88–92 (2018).
- 2
Hasanuddin. Gua Panningnge di Mallawa, Maros: kajian tentang gua hunian berdasarkan artefak batu dan sisa fauna. Naditira Widya 11 , 81–96 (2017).
- 3
Bulbeck, D., Pasqua, M. & Di Lello, A. História da cultura de Toalean de South Sulawesi, Indonésia. Perspectiva asiática . 39 , 71–108 (2000).
- 4
Perston, YL et al. Um esquema de classificação padronizado para os artefatos Toalean do Holoceno médio de Sulawesi do Sul, Indonésia. PLoS ONE 16 , e0251138 (2021).
- 5
Malaspinas, A.-S. et al. Uma história genômica da Austrália aborígine. Nature 538 , 207–214 (2016).
- 6
Pugach, I., Delfin, F., Gunnarsdóttir, E., Kayser, M. & Stoneking, M. Dados de todo o genoma substanciam o fluxo gênico do Holoceno da Índia para a Austrália. Proc. Natl Acad. Sci. USA 110 , 1803–1808 (2013).
- 7
Rasmussen, M. et al. Um genoma aborígine australiano revela dispersões humanas separadas na Ásia. Science 334 , 94-98 (2011).
- 8
Teixeira, JC & Cooper, A. Using hominin introgression to trace modern human dispersals. Proc. Natl Acad. Sci. USA 116 , 15327–15332 (2019).
- 9
O'Connell, JF et al. Quando o Homo sapiens alcançou o sudeste da Ásia e Sahul pela primeira vez? Proc. Natl Acad. Sci. USA 115 , 8482–8490 (2018).
- 10
Clarkson, C. et al. Ocupação humana do norte da Austrália por 65.000 anos atrás. Nature 547 , 306–310 (2017).
- 11
Brumm, A. et al. A arte rupestre mais antiga encontrada em Sulawesi. Sci. Adv . 7 , eabd4648 (2021).
- 12
Sutikna, T. et al. A distribuição espaço-temporal de achados arqueológicos e faunísticos em Liang Bua (Flores, Indonésia) à luz da cronologia revisada do Homo floresiensis . J. Hum. Evol . 124 , 52–74 (2018).
- 13
Samper Carro, SC et al. Em algum lugar além do mar: restos cranianos humanos das ilhas Lesser Sunda (Ilha de Alor, Indonésia) fornecem informações sobre o povoamento do Pleistoceno Superior da Ilha do Sudeste Asiático. J. Hum. Evol . 134 , 102638 (2019).
- 14
Bird, MI et al. O assentamento humano inicial de Sahul não foi um acidente. Sci. Rep . 9 , 8220 (2019).
- 15
Browning, SR, Browning, BL, Zhou, Y., Tucci, S. & Akey, JM A análise de dados de sequência humana revela dois pulsos de mistura Denisovan arcaica. Cell 173 , 53-61.e9 (2018).
- 16
Consórcio GenomeAsia100K. O Projeto GenomeAsia 100K permite descobertas genéticas em toda a Ásia. Nature 576 , 106-111 (2019).
- 17
Jacobs, GS et al. Múltiplas linhagens denisovanas profundamente divergentes em papuas. Cell 177 , 1010–1021.e32 (2019).
- 18
Ascendência de Qin, P. & Stoneking, M. Denisovan nas populações da Eurásia oriental e dos nativos americanos. Mol. Biol. Evol . 32 , 2665–2674 (2015).
- 19
Reich, D. et al. História genética de um grupo de hominíneos arcaico da caverna Denisova na Sibéria. Nature 468 , 1053–1060 (2010).
- 20
Reich, D. et al. Adição de Denisova e as primeiras dispersões humanas modernas no Sudeste Asiático e na Oceania. Sou. J. Hum. Genet . 89 , 516-528 (2011).
- 21
Vernot, B. et al. Escavando DNA de Neandertal e Denisovan de genomas de indivíduos da Melanésia. Science 352 , 235–239 (2016).
- 22
Choin, J. et al. Informações genômicas sobre a história da população e adaptação biológica na Oceania. Nature 592 , 583–589 (2021).
- 23
Teixeira, JC et al. Ascendência denisovana difundida na Ilha do Sudeste Asiático, mas nenhuma evidência de uma mistura substancial de hominídeos superarcaicos. Nat. Ecol. Evol . 5 , 616–624 (2021).
- 24
Mondal, M., Bertranpetit, J. & Lao, O. Computação Bayesiana aproximada com aprendizado profundo suporta uma terceira introgressão arcaica na Ásia e Oceania. Nat. Comun . 10 , 246 (2019).
- 25
Mondal, M. et al. A análise genômica de Andamanese fornece insights sobre a antiga migração humana para a Ásia e sua adaptação. Nat. Genet . 48 , 1066–1070 (2016).
- 26
Lipson, M. et al. Reconstruindo a história da população austronésica na Ilha do Sudeste Asiático. Nat. Comun . 5 , 4689 (2014).
- 27
Suryatman, et al. Artefak batu preneolitik situs Leang Jarie: bukti teknologi Maros Point tertua de Kawasan budaya Toalean, Sulawesi Selatan. AMERTA 37 , 1-17 (2019).
- 28
Anggraeni, A., Simanjuntak, T., Bellwood, P. & Piper, P. Fundações neolíticas no vale de Karama, West Sulawesi, Indonésia. Antiquity 88 , 740-756 (2014).
- 29
Pawlik, A., Croxier, R., Fuentes, R., Wood, R. & Piper, P. Tradições funerárias no início do meio-Holoceno Ilha Sudeste Asiático: novas evidências de Bubog-1, Ilha Ilin, Mindoro Occidental. Antiquity 93 , 901–918 (2019).
- 30
Fu, Q. et al. Um ser humano moderno da Romênia com um ancestral Neandertal recente. Nature 524 , 216–219 (2015).
- 31
Fu, Q. et al. Uma escala de tempo revisada para a evolução humana com base em genomas mitocondriais antigos. Curr. Biol . 23 , 553–559 (2013).
- 32
Lazaridis, I. et al. Genomas humanos antigos sugerem três populações ancestrais para os europeus atuais. Nature 513 , 409-413 (2014).
- 33
Patterson, N. et al. Mistura antiga na história humana. Genetics 192 , 1065–1093 (2012).
- 34
Skoglund, P. et al. Informações genômicas sobre o povoamento do sudoeste do Pacífico. Nature 538 , 510-513 (2016).
- 35
Kanzawa-Kiriyama, H. et al. Genoma nuclear parcial dos Jomons que viveram há 3.000 anos em Fukushima, Japão. J. Hum. Genet . 62 , 213–221 (2017).
- 36
Sikora, M. et al. A história da população do nordeste da Sibéria desde o Pleistoceno. Nature 570 , 182-188 (2019).
- 37
Yang, MA et al. O DNA antigo indica mudanças e mistura da população humana no norte e no sul da China. Science 369 , 282-288 (2020).
- 38
Yang, MA et al. Um indivíduo de 40.000 anos da Ásia fornece uma visão sobre a estrutura da população inicial na Eurásia. Curr. Biol . 27 , 3202–3208.e9 (2017).
- 39
Meyer, M. et al. Uma sequência de genoma de alta cobertura de um indivíduo Denisovan arcaico. Science 338 , 222-226 (2012).
- 40
Bergström, A. et al. Insights sobre a variação genética humana e história populacional de 929 genomas diversos. Science 367 , eaay5012 (2020).
- 41
Peter, BM 100.000 anos de fluxo gênico entre neandertais e denisovanos nas montanhas Altai. Pré-impressão em https://doi.org/10.1101/2020.03.13.990523 (2020).
- 42
Pickrell, JK & Pritchard, JK Inferência de divisões e misturas de populações a partir de dados de frequência de alelos em todo o genoma. PLoS Genet . 8 , e1002967 (2012).
- 43
Sikora, M. et al. Genomas antigos mostram o comportamento social e reprodutivo de forrageadoras do início do Paleolítico Superior. Science 358 , 659–662 (2017).
- 44
Aubert, M. et al. Arte em cavernas do Pleistoceno de Sulawesi, Indonésia. Nature 514 , 223-227 (2014).
- 45
Aubert, M. et al. A cena de caça mais antiga da arte pré-histórica. Nature 576 , 442–445 (2019).
- 46
van den Bergh, GD et al. Primeira ocupação hominínea de Sulawesi, Indonésia. Nature 529 , 208-211 (2016).
- 47
Wang, C.-C. et al. Informações genômicas sobre a formação de populações humanas no Leste Asiático. Nature 591 , 413–419 (2021).
- 48
Bellwood, P. First Islanders: Prehistory and Human Migration in Island Southeast Asia (Wiley Blackwell, 2017).
- 49.
Mallick, S. et al. The Simons Genome Diversity Project: 300 genomas de 142 populações diversas. Nature 538 , 201–206 (2016).
- 50
Fu, Q. et al. A história genética da Idade do Gelo na Europa. Nature 534 , 200–205 (2016).
- 51
Scott, GR & Turner, CG II. A antropologia dos dentes humanos modernos: morfologia dentária e sua variação nas populações humanas recentes (Cambridge Univ. Press, 1997).
- 52
Hillson, S. Dental Anthropology (Cambridge Univ. Press, 1996).
- 53
Pinhasi, R. et al. O DNA antigo ideal é obtido da parte do ouvido interno do osso petroso humano. PLoS ONE 10 , e0129102 (2015).
- 54
Dabney, J. et al. Sequência completa do genoma mitocondrial de um urso da caverna do Pleistoceno Médio reconstruída a partir de fragmentos de DNA ultracurtos. Proc. Natl Acad. Sci. USA 110 , 15758–15763 (2013).
- 55
Rohland, N., Harney, E., Mallick, S., Nordenfelt, S. & Reich, D. Partial uracil-DNA-glycosylase treatment for screening of Ancient DNA. Phil. Trans. R. Soc. Lond. B 370 , 20130624 (2015).
- 56
Gansauge, M.-T., Aximu-Petri, A., Nagel, S. & Meyer, M. Preparação manual e automatizada de bibliotecas de DNA de fita simples para o sequenciamento de DNA de restos biológicos antigos e outras fontes de DNA altamente degradado . Nat. Protoc . 15 , 2279-2300 (2020).
- 57
Kircher, M., Sawyer, S. & Meyer, M. A indexação dupla supera imprecisões no sequenciamento multiplex na plataforma Illumina. Nucleic Acids Res . 40 , e3 (2012).
- 58
Peltzer, A. et al. EAGER: eficiente reconstrução do genoma antigo. Genome Biol . 17 , 60 (2016).
- 59.
Li, H. & Durbin, R. Alinhamento de leitura curta rápido e preciso com transformada de Burrows-Wheeler. Bioinformatics 25 , 1754–1760 (2009).
- 60
Peyrégne, S. & Peter, BM AuthentiCT: um modelo de dano de DNA antigo para estimar a proporção de contaminação de DNA atual. Genome Biol . 21 , 246 (2020).
- 61
Renaud, G., Slon, V., Duggan, AT & Kelso, J. Schmutzi: estimativa de contaminação e consenso mitocondrial endógeno pedindo DNA antigo. Genome Biol . 16 , 224 (2015).
- 62
Vianello, D. et al. HAPLOFIND: um novo método para atribuição de haplogrupo de mtDNA de alto rendimento. Zumbir. Mutat . 34 , 1189–1194 (2013).
- 63
Katoh, K., Misawa, K., Kuma, K. & Miyata, T. MAFFT: um novo método para alinhamento de sequência múltipla rápido com base na transformada rápida de Fourier. Nucleic Acids Res . 30 , 3059–3066 (2002).
- 64
Kumar, S., Stecher, G., Li, M., Knyaz, C. & Tamura, K. MEGA X: análise genética evolutiva molecular em plataformas de computação. Mol. Biol. Evol . 35 , 1547–1549 (2018).
- 65
Skoglund, P. et al. Separando o DNA endógeno antigo da contaminação moderna em um Neandertal siberiano. Proc. Natl Acad. Sci. USA 111 , 2229–2234 (2014).
- 66
Patterson, N., Richter, DJ, Gnerre, S., Lander, ES & Reich, D. Evidência genética para especiação complexa de humanos e chimpanzés. Nature 441 , 1103-1108 (2006).
- 67
Chang, CC et al. PLINK de segunda geração: enfrentando o desafio de conjuntos de dados maiores e mais ricos. Gigascience 4 , 7 (2015).
- 68
Narasimhan, VM et al. A formação de populações humanas na Ásia do Sul e Central. Science 365 , eaat7487 (2019).
Reconhecimentos
A pesquisa de 2019 em Leang Panninge foi financiada por um Australian Research Council Future Fellowship concedido à AB (FT160100119), juntamente com o apoio financeiro da Griffith University. A pesquisa foi autorizada pelo Ministério de Pesquisa e Tecnologia do Estado (RISTEK) e foi conduzida em colaboração com Pusat Penelitian Arkeologi Nasional (ARKENAS), Jacarta, Indonésia. Outras organizações participantes importantes na Indonésia incluem Departemen Arkeologi Fakultas Ilmu Budaya, Universitas Hasanuddin (UNHAS, Makassar) e Balai Arkeologi Sulawesi Selatan (Makassar). Os seguintes estudantes de arqueologia da UNHAS participaram do trabalho de campo: FN Shalawat, AA Qalam, KM Prayoga, M. Sura, MN Taufik e MA Oka. Assistentes de campo locais incluem Irwan, Amar e Hardin. A equipe que auxilia o DBA análise morfológica de restos de esqueletos humanos incluiu Fahkri (Balai Arkeologi Sulawesi Selatan), Cika e M. Hafdal. A. Crowther ajudou com identificações paleobotânicas e R. Wood aconselhou sobre calibração de radiocarbono. A escavação de 2015 foi realizada pela UNHAS em colaboração com o Centro de Pesquisa Arqueológica Global da Universiti Sains Malaysia. Agradecemos à equipe computacional e de laboratório do Instituto Max Planck para a Ciência da História Humana (Jena), em particular C. Freund, R. Radzeviciute e A. Wissgott. Aspectos dessa pesquisa também foram apoiados financeiramente pela Bolsa da Universidade de Pesquisa da Universiti Sains Malaysia de S. Chia (1001 / PARKEO / 870002), a bolsa inicial ERC 'Waves' (ERC758967) e a Sociedade Max Planck. Os modelos de elevação digital na Fig. 1a,b foram criados no ArcGIS (QGIS) da Shuttle Radar Topography Mission (SRTM 1) Dados do Arc-Second Global cortesia do US Geological Survey.
Financiamento
Financiamento de acesso aberto fornecido pela Max Planck Society.
Informação sobre o autor
Afiliações
Contribuições
AB, JK, CP e AD conceberam e conduziram o estudo. A escavação de 2015 foi liderada pela AD em colaboração com S. Chia e envolveu contribuições importantes de MN, IS, Supriadi e DATPAB dirigida a escavação de 2019, análise adicional e conceituação em colaboração com AAO, MR, BH e a equipe sênior Universitas Hasanuddin (AD , MN e IS). A FAS supervisionou as atividades de campo de 2019 com BB, que recuperou os restos mortais do local em 2018 com AMSDPM conduziu a escavação laboratorial dos restos mortais humanos e forneceu observações e análises em campo e pós-escavação. DB descreveu e analisou os restos mortais do esqueleto humano. YLP e Suryatman analisaram os artefatos de pedra. K. Newman conduziu análises chave pós-escavação, processamento de dados e preparação de figuras com BBHasanuddin conduziu escavações iniciais importantes no local. S. Carlhoff gerou dados genômicos. S. Carlhoff, LS, BMP e KP realizaram análises genéticas com a contribuição crítica de CP, K. Nägele, CJ e JKS Carlhoff, K. Nägele, CP e AB escreveram o manuscrito com a contribuição crítica de JK e dos autores restantes.
Autores correspondentes
Declarações de ética
Interesses competitivos
Os autores declaram não haver interesses conflitantes.
Informações adicionais
Informações da revisão por pares A Nature agradece a Richard Roberts, Martin Sikora e o (s) outro (s) revisor (es) anônimo (s) por sua contribuição para a revisão por pares deste trabalho.
Nota do editor A Springer Nature permanece neutra em relação a reivindicações jurisdicionais em mapas publicados e afiliações institucionais.
Figuras e tabelas de dados estendidos
Dados estendidos Fig. 1 Tipos de artefatos Toalean e escavações em Leang Panninge.
a , b , Tipos de artefatos típicos do Holoceno tardio a partir de assembléias Toalean em South Sulawesi; pedra lascada por pressão 'pontos de Maros' (ambos os artefatos são de Leang Pajae, Maros) ( a ) e micrólitos apoiados (superior: Leang Pajae; inferior: Leang Bulu Bettue, Maros) ( b ). c , pontos ósseos (à esquerda: da camada 4, sepultamento humano Toalean, Leang Panninge; à direita: Leang Rakkoe, Maros). d - i , Escavação em Leang Panninge em 2019. Escavação de uma trincheira colocada adjacente à parede traseira na entrada da caverna voltada para oeste ( d , e): visto de noroeste a sudeste (observe que o nome do local da caverna está incorretamente transcrito neste banner produzido localmente) ( d ); visto de norte a sul ( e ); noroeste a sudeste ( f ); detalhe da escavação ( g ). Faces de trincheiras escavadas no final da temporada de campo de 2019, seguindo amostragem de coluna de solo ( h , i ): trincheira vista de nordeste a sudoeste, mostrando a intersecção entre as paredes ocidentais das praças de escavação S16T6 e S17T6 e paredes sul de S17T6 e S17T7 ( h ); trincheira vista de norte a sul, com a seção estratigráfica principal voltada para o observador mostrando as paredes do norte de S16T6 e S16T7 ( i) Créditos da imagem: Y. Perston ( a - c ); Equipe de pesquisa Leang Panninge ( d - i ).
Dados estendidos Fig. 2 Planta do local, estratigrafia e evidências de datação em Leang Panninge.
a - c , Planos de vista do local da caverna mostrando as localizações das unidades de escavação anteriores e das praças S16T6 e S17T6, onde o cemitério Toalean foi exposto em 2015. Escavações na entrada voltada para oeste (que tem 10 m de altura e 28 m de largura) produziu densas assembléias arqueológicas Toalean. Cinza claro, rocha calcária; cinza escuro, coluna / pilar espeleotema; cinza com x, estalagmite. Os níveis de contorno estão em centímetros. Siglas e iniciais que denotam as instituições arqueológicas e departamentos de patrimônio da Indonésia são os seguintes: BALAR, Balai Arkeologi Sulawesi Selatan; BPCB, Balai Pelestarian Cagar Budaya Makassar; UNHAS, Universitas Hasanuddin. A vala de 2015 foi ampliada em 2019, formando os quadrados S16T7 e S17T7. d , Seção da boca da caverna. e, Estratigrafia e datação. L1, camada 1; L2, camada 2 e assim por diante. Pontos pretos denotam as posições plotadas em 3D de artefatos de pedra, itens faunísticos (ossos, dentes e conchas), restos paleobotânicos, conchas e outras descobertas escavadas em 2019. Oito camadas arqueológicas distintas de ocupação humana são evidentes: o estrato cultural superior, camada 1 (aproximadamente 1,5 kyr cal bp ), contém cerâmica 'Neolítica' e micrólitos com fundo de Toalean; as camadas 2-4 compreendem depósitos acerâmicos de ocupação Toalean. Maros aponta micrólitos anteriores à data e aparecem no nível Toalean mais antigo (ver também a ref. 27 ); a camada 5 (aproximadamente 9,4–8,7 kyr cal bp ) e os estratos subjacentes (camadas 6–8) são pré-Toaleanas. O horizonte Toalean mais profundo, camada 4, uma argila siltosa (7,9-5,1 kyr calbp ), continha o único sepultamento definido associado a Toalean da região. O sepultamento foi revelado em 2015 dentro dos quadrados contíguos S16T6 e S17T6. Em 2019, uma trincheira de 2 x 1 m escavada adjacente a esses quadrados expôs estratos não perturbados a uma profundidade de 3 m, sem atingir o leito rochoso (Dados estendidos Figs. 2 , 3 ). AMS 14 C calibrados (95% de probabilidade) são mostrados à direita (as informações suplementares contêm detalhes sobre os métodos de datação). Os materiais datados compreendem amostras de carvão vegetal ( N = 8) e Canarium sp. Carbonizado . sementes ( N = 3). A inversão de idade na camada 4 sugere um grau de mistura pós-deposição que não afeta a idade de sepultamento inferida. f , sepultamento Toalean da camada 4 (imagem 3D gerada usando PhotoScan). O indivíduo foi enterrado em uma posição flexionada e coberto com grandes pedras (indicadas 1–5). g , Crânio fragmentado. h , i , pontos de Maros recuperados com os restos humanos. Um chert atípico ponto de Maros ( h ) foi encontrado logo abaixo do crânio. Um ponto de Maros clássico ( i ) com base oca e margens serrilhadas (falta a ponta devido à quebra) foi encontrado aproximadamente 40 mm abaixo da incisura ciática maior. Créditos da imagem: equipe de pesquisa Leang Panninge ( a - g); Y. Perston ( h , i ).
Dados estendidos Fig. 3 Seção estratigráfica em Leang Panninge.
As distribuições de dados apresentadas nas tabelas à esquerda e as datas calibradas de radiocarbono AMS ( 14 C) (mostradas projetadas no perfil estratigráfico) são baseadas nos resultados das escavações de 2019. As idades AMS 14 C calibradas são relatadas com 95% de probabilidade (as informações suplementares contêm detalhes sobre os métodos de datação). O esqueleto humano Toalean foi exposto em 2015 por uma equipe da Universitas Hasanuddin (UNHAS) em colaboração com Balar Sulsel e Universiti Sains Malaysia (ver Informações Suplementares ).
Dados estendidos Fig. 4 Enterro humano Toalean de Leang Panninge.
a , Inumação de adulto solteiro. A fêmea de 17 a 18 anos foi enterrada em uma posição flexionada e parcialmente coberta por vários paralelepípedos grandes. b , Detalhe dos agrupamentos de fragmentos que compõem a área do crânio. O esqueleto é fragmentário (Tabela Complementar 2 ), mas o crânio, embora esmagado post-mortem, é bem representado pela dentição mandibular e mandibular, dentição maxilar e maxilar, osso frontal, temporais, osso parietal direito e osso occipital. O crânio foi posicionado entre duas grandes pedras (indicadas como 1 e 2). c , Detalhe do recurso de sepultamento; A pedra # 1 foi colocada sobre uma das mãos, a pedra # 4 foi posicionada sobre o pé esquerdo e a pedra # 5 cobriu a região pélvica. d, Pelves semi-completas e pé esquerdo (face plantar voltada para cima). e , Bloco de sedimentos contendo o crânio, após retirada da vala arqueológica e antes da escavação em laboratório. Créditos da imagem: equipe de pesquisa Leang Panninge ( a - g ).
Dados estendidos Fig. 5 Tipos de ferramentas de pedra Toalean do cemitério Toalean e contextos associados em Leang Panninge.
a , b , Posição do ponto de Chert Maros (mostrado na inserção em a ) encontrado abaixo da pelve direita durante a escavação do bloco de sedimentos contendo as pelves semi-completas. As setas vermelhas em um e b indicar a posição do ponto de Maros em meio a detritos-descamação pedra e outros artefatos culturais (no momento estas fotografias foram tiradas, os ossos da região pélvica já havia sido removido). c , Posição do ponto de Maros chert não clássico (inserido na ilustração, localização original destacada pela seta azul) encontrada abaixo do crânio entre outros artefatos de pedra e vestígios culturais (dentes humanos ainda são visíveis no topo do bloco de sedimentos sob escavação) . d, Ponto de Maros exposto durante a escavação. e - h , Exemplos de pontos de Maros da camada 4, acima do nível do sepultamento humano Toalean: quadrado S16T6, cuspo 18 (Encontre # 62) ( e ); quadrado S16T6, espeto 18 (Encontre # 77) ( f ); quadrado S17T6, espeto 18 (Find # 54) ( g ); quadrado S17T6, cuspo 16 (Encontre # 95) ( h ). i - k , Exemplos de pontos de Maros dos espetos 19 e 20 dentro dos quadrados S16T6 e S17T6, onde o túmulo humano de Toalean estava localizado. l - p , Exemplos de pontos de Maros de outras praças escavadas em Leang Panninge. Créditos da imagem: equipe de pesquisa Leang Panninge ( a - p)
Dados estendidos Fig. 6 Análise de componentes principais.
Calculado nos grupos atuais 18 , 32 , 33 , projetando todos os genomas antigos 1 , 34 , 35 , 36 , 37 , 38 incluindo todos os SNPs disponíveis ou apenas fragmentos de DNA danificados após a filtragem de PMD das bibliotecas de fita simples e dupla do indivíduo Leang Panninge. a , Componentes principais calculados em indivíduos do leste e sudeste da Ásia, onde os grupos papua, australiano indígena e nasioi (de Bougainville) são projetados. b , Componentes principais calculados em indivíduos do Leste, Sudeste Asiático e Perto da Oceania.
Dados estendidos Fig. 7 A posição do genoma de Leang Panninge na história genética da população regional.
Testado com estatísticas f 4 , em que X denota o grupo testado. a , A afinidade dos grupos 49 do Leste Asiático, do Sul da Ásia, do Sudeste Asiático e da Oceânia com indivíduos de Leang Panninge e de Papua. b , Afinidades entre os grupos atuais da Oceania próxima e o genoma de Leang Panninge 49 . c , A afinidade do genoma de Leang Panninge com o genoma Denisovan em comparação com os indivíduos atuais e antigos da região 26 , 36 , 37 , 38 . d , A afinidade do genoma de Leang Panninge com genomas antigos da região 26 , 36, 37 , 38 em comparação com os grupos da Papua 49 . Os dados são apresentados como valores exatos de f 4 ± um e três erros padrão indicados como linhas cinza claro e escuro, respectivamente; estatísticas com z- scores acima | 3 | são exibidos em verde.
Dados estendidos Fig. 8 Teste de permutação na sobreposição da sequência Denisovan intrograda no indivíduo Leang Panninge e nos grupos atuais.
Após 500 permutações onde a sequência Denisovan é distribuída aleatoriamente pelo genoma. A linha vertical pontilhada é a sobreposição observada nos dados não permutados. O valor p empírico indica quantas permutações mostram mais sobreposição do que a sobreposição observada nos dados não permutados.
Dados estendidos Fig. 10 A relação genética de Leang Panninge com outros grupos da Ásia-Pacífico.
Dados para outros grupos da Ásia-Pacífico das refs. 37 , 38 , 39 , 43 , 49 . a - d , Árvores de máxima verossimilhança após adicionar incrementalmente um evento de migração (de zero a quatro) conforme inferido por TreeMix 42 ; os resíduos correspondentes de modelos individuais são apresentados à direita (Fig. 6 complementar ).
Informação suplementar
Informação suplementar
Este arquivo contém texto suplementar, tabelas suplementares 1 a 27, figuras suplementares 1 a 11 e referências suplementares.
Direitos e permissões
Acesso aberto Este artigo está licenciado sob uma Licença Internacional Creative Commons Atribuição 4.0, que permite o uso, compartilhamento, adaptação, distribuição e reprodução em qualquer meio ou formato, desde que você dê o devido crédito ao (s) autor (es) original (is) e à fonte, fornecer um link para a licença Creative Commons e indicar se foram feitas alterações. As imagens ou outro material de terceiros neste artigo estão incluídos na licença Creative Commons do artigo, a menos que indicado de outra forma em uma linha de crédito para o material. Se o material não estiver incluído na licença Creative Commons do artigo e seu uso pretendido não for permitido por regulamentação legal ou exceder o uso permitido, você precisará obter permissão diretamente do detentor dos direitos autorais. Para ver uma cópia desta licença, visitehttp://creativecommons.org/licenses/by/4.0/ .
Nenhum comentário:
Postar um comentário
Observação: somente um membro deste blog pode postar um comentário.