Padrões Filogeográficos Globais e de Mistura em Lobos Cinzentos e Legado Genético de uma Antiga Linhagem Siberiana
Abstrato
As relações evolutivas entre linhagens extintas e existentes fornecem informações importantes sobre a resposta das espécies às mudanças ambientais.
O lobo cinzento está entre os poucos grandes carnívoros holárticos que sobreviveram às extinções da megafauna do Pleistoceno Superior, respondendo às profundas mudanças ambientais daquele período com perda de linhagens distintas e mudanças filogeográficas, e passando pela domesticação. Reconstruímos padrões filogeográficos globais do genoma em lobos modernos, incluindo lobos siberianos anteriormente sub-representados, e avaliamos suas relações evolutivas com um lobo previamente genotipado de Taimyr, na Sibéria, datado de 35 Kya.
A estrutura filogeográfica inferida foi afetada pela mistura com cães, coiotes e chacais dourados, enfatizando a importância de contabilizar esse processo em estudos filogeográficos. A linhagem Taimyr era distinta dos lobos siberianos modernos e constituía uma linhagem irmã dos lobos eurasianos modernos e cães domésticos, com uma posição ambígua em relação aos lobos norte-americanos. Detectamos o fluxo gênico da linhagem Taimyr para as raças de cães do Ártico, mas os métodos de agrupamento populacional indicaram uma semelhança mais próxima do lobo Taimyr com os lobos modernos do que com os cães, implicando relações pós-divergência complexas entre essas linhagens. Nosso estudo mostra que a introgressão de populações ecoespecíficas e congenéricas ecologicamente diversas foi comum na história evolutiva dos lobos e pode ter facilitado sua adaptação às mudanças ambientais.
Detectamos o fluxo gênico da linhagem Taimyr para as raças de cães do Ártico, mas os métodos de agrupamento populacional indicaram uma semelhança mais próxima do lobo Taimyr com os lobos modernos do que com os cães, implicando relações pós-divergência complexas entre essas linhagens. Nosso estudo mostra que a introgressão de populações ecoespecíficas e congenéricas ecologicamente diversas foi comum na história evolutiva dos lobos e pode ter facilitado sua adaptação às mudanças ambientais.
Detectamos o fluxo gênico da linhagem Taimyr para as raças de cães do Ártico, mas os métodos de agrupamento populacional indicaram uma semelhança mais próxima do lobo Taimyr com os lobos modernos do que com os cães, implicando relações pós-divergência complexas entre essas linhagens. Nosso estudo mostra que a introgressão de populações ecoespecíficas e congenéricas ecologicamente diversas foi comum na história evolutiva dos lobos e pode ter facilitado sua adaptação às mudanças ambientais.
Introdução
O Pleistoceno tardio foi um período de numerosas extinções de grandes mamíferos, resultantes do efeito combinado das mudanças climáticas e do impacto humano 1 . As espécies que sobreviveram às extinções da megafauna experimentaram gargalos demográficos, extinções locais e mudanças filogeográficas 2 , 3 , 4 – os eventos que também precederam extinções em várias espécies 5 , 6 , 7 , 8 . Estudos paleogenômicos revelaram a presença de fragmentos genômicos originários de espécies extintas em pools gênicos de seus parentes existentes 9 , 10 , 11.
Isso pode apontar para a inundação genética como um fator contribuinte dessas extinções, embora algumas populações carreguem variantes de genes derivados de hibridação mostrando assinaturas de seleção positiva 9 , 12 , 13 . Isso sugere que a contribuição genética de espécies extintas pode ter facilitado a adaptação de seus parentes existentes às condições ambientais em rápida mudança. A reconstrução das relações evolutivas entre linhagens extintas e existentes pode, assim, fornecer uma visão direta dos caminhos evolutivos complexos para a extinção ou sobrevivência de uma espécie.
O lobo cinzento ( Canis lupus ) está entre os poucos grandes carnívoros que sobreviveram às extinções da megafauna do Pleistoceno Superior. Semelhante a muitas outras espécies da megafauna, o lobo experimentou um declínio populacional global no final do Pleistoceno 14 , 15 , que foi associado a extinções de ecomorfos distintos e mudanças filogeográficas 16 , 17 , 18 , 19 . Nesse período, uma das linhagens dos lobos evoluiu para o cão doméstico ( Canis lupus familiaris ; revisado em 20). A domesticação garantiu o sucesso evolutivo dessa linhagem por meio da expansão para um novo nicho ecológico, afetando substancialmente as populações não domesticadas por meio da competição por recursos, transferência de doenças e mistura 21 , 22 . As populações de lobos também carregam assinaturas de misturas passadas e atuais com chacais dourados ( Canis aureus ) na Eurásia 23 , 24 , 25 e coiotes ( Canis latrans ) na América do Norte 26 , 27 , 28 .
Todos os lobos existentes têm um ancestral comum recente estimado em cerca de 32 Kya – uma data que coincide com o início do declínio demográfico global da espécie 15 . Estudos baseados em dados de toda a sequência do genoma nuclear revelaram que os lobos eurasianos modernos formam um clado monofilético distinto dos lobos norte-americanos 15 , 28 , enquanto os genomas mitocondriais apresentam um padrão filogeográfico mais complexo 18 .
Uma análise recente dos genomas mitocondriais de lobos antigos e modernos inferiu que as populações de lobos modernos podem ser rastreadas até um único evento de expansão originário de cerca de 25 Kya em Beringia (a região que inclui o leste da Sibéria, Alasca e parte do Yukon, todos anteriormente conectados por terra ) 19. Lobos antigos de outras partes da distribuição do lobo cinzento carregavam linhagens mitocondriais distintas não representadas nas populações de lobos contemporâneas, implicando extinção e substituição dessas linhagens 18 , 19 , 29 .
Assim, o genoma nuclear de um lobo da Península Taimyr na Sibéria, datado de 34,9 Kya, era filogeneticamente distinto dos genomas de lobos e cães existentes, o que implica que este indivíduo representa uma linhagem divergente extinta 10 . Em contraste, o genoma mitocondrial desse indivíduo agrupado com lobos modernos 10 , o que pode resultar do fluxo gênico pós-divergência. Além disso, foi demonstrado que as raças de cães do Ártico carregam assinaturas de misturas anteriores com a linhagem de lobos Taimyr 10 . No entanto, as raças de cães do Ártico também mostram assinaturas de mistura com lobos modernos 30, implicando episódios de mistura independentes ocorrendo em momentos diferentes ou continuidade da linhagem Taimyr em lobos siberianos. Uma melhor compreensão do legado genético da linhagem do lobo Taimyr requer comparação com dados abrangentes sobre lobos modernos, mas a representação de lobos asiáticos modernos em estudos genéticos publicados é muito limitada, com a Sibéria sendo particularmente sub-representada.
A origem beringiana proposta dos lobos modernos 19deveria ter resultado em similaridade genética entre as populações modernas da Sibéria Oriental e do Alasca (antigas partes da Beríngia), mas essas populações não foram incluídas em estudos anteriores sobre a filogeografia global do lobo. Portanto, o conhecimento dos padrões filogenéticos em lobos modernos da Ásia, e da Sibéria em particular, é necessário para entender melhor a complexa história desta espécie.
Neste estudo, reconstruímos padrões filogeográficos de todo o genoma em populações modernas de lobos cinzentos em toda a sua distribuição holártica, com base em um extenso conjunto de amostras que inclui lobos siberianos anteriormente sub-representados (Fig. 1 ). Também fornecemos a primeira reconstrução global de padrões de mistura de lobos com cães domésticos, coiotes e chacais dourados. Usamos esses dados para testar as seguintes hipóteses:
- (1)
Não há continuidade entre a linhagem Taimyr do Pleistoceno Superior e as linhagens modernas de lobos na Eurásia 10 , 19 . Se for esse o caso, os lobos siberianos modernos devem se agrupar com outras populações de lobos modernos, e não com o lobo Taimyr.
- (2)
Todos os lobos modernos originaram-se de uma única expansão beringiana 19 . Isso deve resultar em um aumento gradual do desequilíbrio de ligação com o aumento da distância da Sibéria Oriental e do Alasca.
- (3)
A mistura de lobos em raças de cães do Ártico é originária da linhagem Taimyr 10 . Isso deve resultar na inferência do fluxo gênico do lobo Taimyr, e não dos lobos siberianos modernos nas raças árticas.
- (4)
Os lobos cinzentos da Eurásia e os chacais dourados experimentaram uma mistura antiga 23 , 25 . Nesse cenário, os blocos de ancestralidade originários do chacal dourado devem ser detectados nos lobos cinzentos da Eurásia.
- (5)
A inferência filogeográfica em lobos cinzentos modernos é afetada por episódios de mistura com outros canídeos. Nesse caso, esperamos que a topologia das relações de ancestralidade inferidas entre o lobo e outras linhagens de canídeos mude uma vez que os episódios de miscigenação sejam levados em consideração.
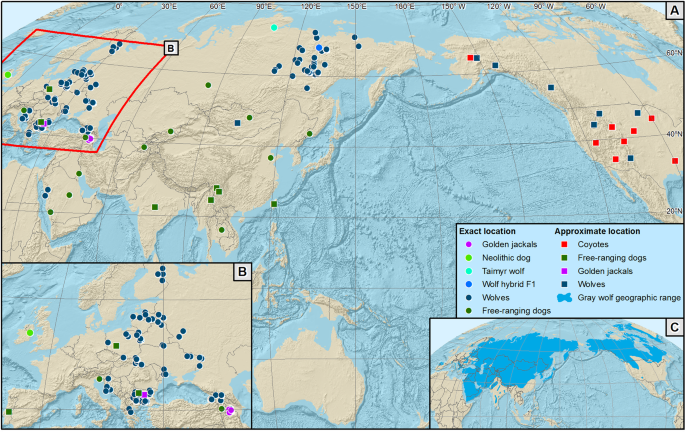
Distribuição de canídeos silvestres e cães de vida livre analisados neste estudo. ( A ) Distribuição de amostras em todo o Holártico, ( B ) distribuição de amostras na Europa, ( C ) faixa de distribuição do lobo cinzento. Cada símbolo representa vários indivíduos amostrados nos mesmos locais. O mapa não inclui amostras de chacais de dorso preto (da Namíbia), cães de vida livre da América do Norte (com locais de amostragem desconhecidos) e cães de raça pura. Para a lista de todas as amostras analisadas no estudo, consulte a Tabela Complementar 1 . Software utilizado para gerar o mapa: ESRI 2014. ArcGIS Desktop: Release 10.2.2. Redlands, CA: Instituto de Pesquisa de Sistemas Ambientais. Relevo sombreado gerado a partir de SRTM DEM 90 m 63. Países marcados usando dados do Natural Earth (dados gratuitos de mapa vetorial e raster @ naturalearthdata.com). Todas as versões dos dados do mapa vetorial e raster do Natural Earth são de domínio público. Eles podem ser usados de qualquer maneira sem licença, incluindo modificação de conteúdo e design, disseminação eletrônica e impressão em offset. Faixa geográfica do lobo cinzento desenhada de acordo com Boitani et al . 64 .
Resultados
Heterozigosidade e padrões de desequilíbrio de ligação em populações mundiais de lobos
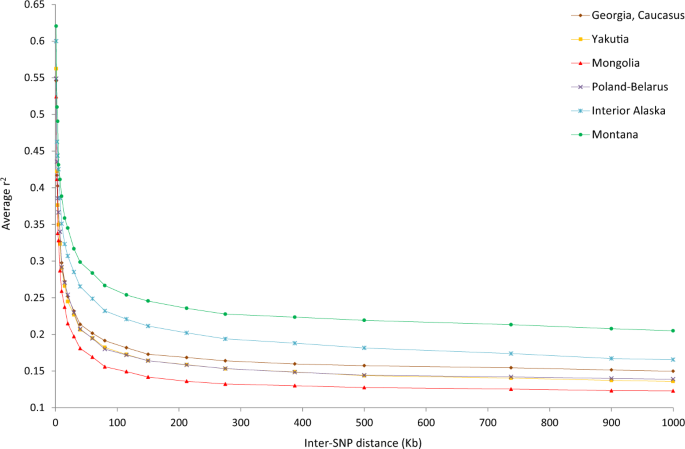
Descobrimos que todas as populações de lobos asiáticos tinham níveis semelhantes de heterozigosidade em todo o genoma (Ho = 0,27), que eram comparáveis aos das populações europeias e superiores aos das populações norte-americanas (tabela complementar 2 ). Os lobos da Mongólia apresentaram menor desequilíbrio de ligação (LD) do que todas as outras populações representando as principais regiões geográficas da Eurásia e América do Norte, em todas as classes de distância avaliadas (Fig. 2). As populações das regiões correspondentes ao leste e oeste da Beríngia (Interior do Alasca e Yakutia, respectivamente) tiveram LD consideravelmente maior em todas as classes de distância em comparação com os lobos da Mongólia. Os níveis de DL nas populações norte-americanas foram maiores do que nas populações da Eurásia e aumentaram ao longo de um gradiente norte-sul em todo o continente norte-americano. Em contraste, as populações da Eurásia não mostraram um único gradiente geográfico de padrões de LD (Fig. 2 , Fig. 1 complementar ).
Padrões de desequilíbrio de ligação em populações de lobos. O desequilíbrio de ligação é representado pelo coeficiente médio de associação genotípica r 2 e é plotado em função da distância inter-SNP. A estimativa foi realizada para 10 indivíduos não aparentados por população. Os dados para lobos mexicanos foram excluídos do gráfico para maior clareza e são apresentados na Fig. 1 complementar .
Padrões de mistura na população global de lobos detectados a partir de análises de agrupamento populacional
Usamos o ADMIXTURE 31 para avaliar a estrutura genética em populações de lobos em todo o mundo, levando em consideração sua mistura com outros canídeos e cães selvagens. Esta análise, bem como outras análises descritas abaixo (salvo indicação em contrário) foram baseadas no conjunto de 42.320 loci SNP, obtidos após a remoção de loci não genotipados no lobo Taimyr, a fim de maximizar a precisão da comparação deste antigo lobo com o moderno populações.
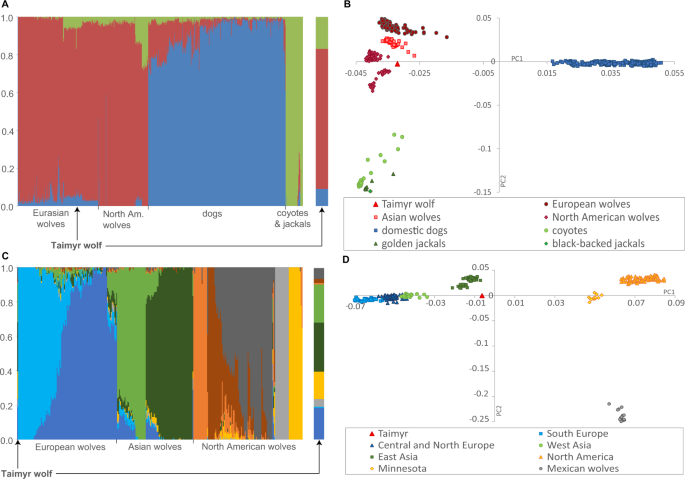
A análise de MISTURA distinguiu cães de canídeos selvagens em K = 2 e lobos de outros canídeos selvagens (coiotes, chacais dourados e chacais de dorso preto) em K = 3. Valores crescentes de K resultaram em subagrupamento dentro das populações de lobos e cães, em vez de separação de as outras espécies de canídeos silvestres. Estas espécies agruparam-se devido ao pequeno tamanho das amostras (ver abaixo). Nenhum K ótimo pode ser identificado entre K = 1 e K = 15, com o erro de validação cruzada diminuindo continuamente com o aumento de K. Isso não implica falta de estrutura, mas reflete subagrupamento dentro das múltiplas espécies de canídeos estudadas.
Em K = 3, o lobo Taimyr agrupou-se com lobos modernos (com valor de atribuição de 0,74), mas apresentou valores de atribuição positivos para o cão (0,09) e agrupamentos de coiote/chacal (0,17). Para valores de K entre 4 e 15, os valores de atribuição do lobo Taimyr mostraram variação limitada: 0,731–0,775 para grupos de lobos cinzentos, 0,075–0,107 para grupos de cães e 0,131–0,168 para grupos de coiotes/chacais (tabela complementar 3 ). Nosso conjunto de dados incluiu populações estruturadas de várias espécies e, portanto, desviou-se das suposições do modelo de MISTURA de que todos os indivíduos são originários de populações homogêneas e cada população está bem representada no conjunto de dados analisado 32. A ancestralidade mista inferida para o lobo Taimyr indica relações complexas entre a linhagem Taimyr e representantes contemporâneos do gênero Canis , mas as proporções estimadas de mistura não devem ser interpretadas como a ancestralidade real desse indivíduo.
Em K = 3, os lobos asiáticos modernos tiveram atribuição positiva ao agrupamento de cães (0,02–0,13) e ao agrupamento de chacal/coiote (0,01–0,13) (Fig. 3A , Fig. Complementar. 2A , Tabela Suplementar 4). Os lobos europeus exibiram grande variação nos valores de atribuição ao agrupamento de cães, e sua atribuição ao agrupamento de chacal/coiote não foi superior a 0,02. Os lobos mexicanos (a população que atualmente habita o Novo México e o Arizona) tiveram valores de atribuição pequenos, mas positivos, para o grupo de cães e mais altos para o grupo de chacais/coiotes (0,13–0,15). Os lobos de Minnesota tiveram valores de atribuição para o agrupamento chacal/coiote superiores a 0,2, enquanto as demais populações norte-americanas não tiveram mais do que 0,05 da ancestralidade inferida do coiote e nenhuma assinatura de mistura de cães, com exceção de dois indivíduos. O termo “lobos norte-americanos” será aplicado posteriormente às populações norte-americanas, excluindo os lobos mexicanos e de Minnesota, que exibem padrões distintos de mistura e estrutura populacional.
Alguns canídeos selvagens individuais tinham níveis de mistura sugerindo que eles são híbridos ou retrocruzados de geração recente. Isso incluiu um indivíduo parecido com um lobo preto de Yakutia (o primeiro caso de coloração negra nesta região conhecido por nós), dois indivíduos de Idaho e da Colúmbia Britânica amostrados como lobos e um chacal dourado de Nagorny Karabakh no Cáucaso, que tinha um pequena mas detectável mistura de lobo (Fig. 3 , Tabela Suplementar 4 ).
Raças de cães e cães de vida livre variaram em seus valores de atribuição ao agrupamento de lobos, com valores mais altos ocorrendo em cães do Leste Asiático e origem do Ártico (Tabela Suplementar 4 ). As raças originárias da mistura recente conhecida com lobos tiveram valores de atribuição para o grupo de lobos de 0,15–0,17 (Czechoslovakian wolfdog) e 0,23–0,34 (Saarloos wolfdog).
Após a avaliação dos padrões globais de miscigenação, realizamos uma análise de MISCIGENAÇÃO incluindo apenas lobos. Esta análise distinguiu os lobos da Eurásia e da América do Norte em K = 2, e os lobos da Eurásia Ocidental, do Leste Asiático e da América do Norte em K = 3. O K ótimo foi 9, distinguindo duas populações regionais na Europa, duas na Ásia e cinco na América do Norte (Fig. 3C , Fig. Complementar. 2B). Em K = 9, o lobo Taimyr foi atribuído com maiores probabilidades a quatro populações de lobos: Leste da Ásia (0,29), Oeste da Ásia (0,22), Nordeste da Europa (0,18) e Minnesota (0,16), com os cinco grupos restantes tendo baixo valores de atribuição (0,15 no total). Em contraste, os valores de atribuição de lobos asiáticos contemporâneos para populações norte-americanas não foram superiores a 0,016.
Estrutura genética da população mundial de lobos inferida a partir da análise de componentes principais
O PCA (realizado usando 42.320 loci) revelou padrões de agrupamento semelhantes como MISTURA , com agrupamentos distintos de lobos cinzentos e cães, e as outras espécies de Canis agrupadas (Fig. 3B , Fig. Complementar. 3A). As posições dos lobos eurasianos no gráfico PCA corresponderam às suas distâncias geográficas, mas as distâncias geográficas entre as populações eurasianas e norte-americanas não se refletiram em sua diferenciação genética. Todos os lobos norte-americanos se agruparam, exceto as populações de Minnesota e México, que mostraram maior proximidade com o grupo de coiote/chacal. Quando apenas os lobos foram incluídos na análise, os lobos eurasianos e norte-americanos foram distinguidos no PC1, e os lobos mexicanos foram distinguidos de outros lobos norte-americanos no PC2 (Fig. 3D , Fig. 4 complementar ).
A análise PCA realizada após a inclusão de amostras adicionais de chacais dourados e chacais de dorso preto (para obter tamanhos de amostra mais equilibrados) resultou em três grupos claramente distintos de coiotes, chacais dourados e chacais de dorso preto (Suplementar Fig. 5 ). Isso implica que essas espécies se agruparam no conjunto de dados original devido a tamanhos amostrais desequilibrados.
A posição do lobo Taimyr no gráfico PCA foi fortemente influenciada pelo conjunto de SNPs analisados e pela composição das populações de referência. No PCA baseado no conjunto de dados incluindo loci SNP com dados ausentes para o lobo Taimyr (90.554 loci), esse indivíduo foi colocado centralmente na parcela, com distâncias semelhantes a grupos de lobos, cães e coiotes (Suplementar Fig. 3B ). A poda dos loci SNP ausentes do lobo Taimyr resultou em seu agrupamento com lobos modernos, embora não dentro de nenhuma população regional (Fig. 3B , Fig. 3A complementar ). Quando apenas os lobos foram incluídos na análise, o lobo Taimyr foi colocado perto, mas não dentro, do grupo de lobos modernos do Leste Asiático (Fig. 3D , Fig. Suplementar 4C ). Para manter a precisão da inferência em relação ao lobo Taimyr, todos os resultados relatados abaixo são baseados no conjunto de dados onde os loci ausentes desse indivíduo foram removidos do conjunto de dados analisado.
Reconstrução das relações de ancestralidade entre as populações de lobos, representando o fluxo gênico interespecífico
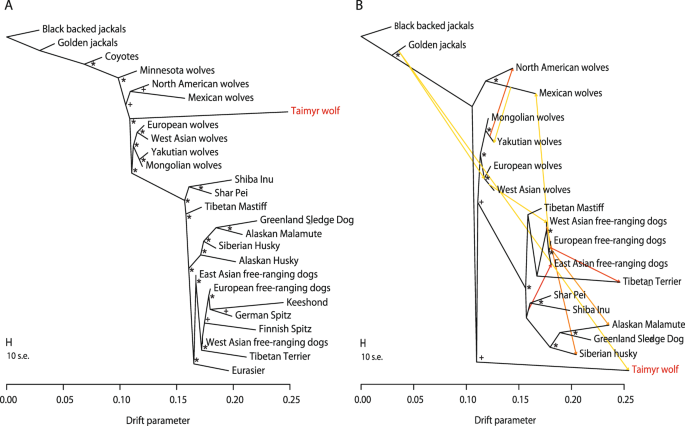
A árvore Maximum Likelihood reconstruída usando TREEMIX 33 mostrou que os lobos norte-americanos divergiram antes da divisão entre os lobos eurasianos modernos e os cães domésticos. O lobo Taimyr foi colocado (com 100% de suporte bootstrap) como a linhagem irmã de todos os lobos eurasianos e cães domésticos, com lobos norte-americanos se ramificando antes da divisão da linhagem Taimyr e lobos eurasianos modernos (Fig. 4A , Suplementar Fig. 6 ) .
Relações de ancestralidade entre o lobo Taimyr e as populações modernas de lobos e cães inferidas em TREEMIX para ( A ) todo o conjunto de populações de canídeos, ( B ) um conjunto populacional reduzido, excluindo aqueles com história de mistura conhecida e assumindo 10 eventos de fluxo gênico. O fluxo gênico é representado por setas, com cores refletindo a intensidade do fluxo gênico, do mais baixo (amarelo) ao mais alto (vermelho). Os nós com suporte de bootstrap de 95 a 100% são marcados com um asterisco e os nós com suporte de bootstrap de 50 a 95% são marcados com um símbolo de mais.
A adição de 10 bordas de migração não alterou as relações de ancestralidade entre as populações de lobos, exceto para os lobos de Minnesota. A inclusão de uma borda de migração de coiotes resultou no agrupamento dos lobos de Minnesota com outros lobos norte-americanos, em vez de formar um ramo separado (Fig. 6B,C complementar ). TREEMIXtambém detectou fluxo gênico pós-divergência de lobos yakutianos para lobos norte-americanos e de coiotes para o ancestral comum dos lobos eurasianos; no entanto, esse último resultado é improvável devido ao longo isolamento geográfico dessas populações. Os restantes eventos de migração refletem a miscigenação entre diferentes raças ou populações de cães, incluindo casos de miscigenação entre raças de cães que são consistentes com a história da formação da raça (por exemplo, cães-lobo da Checoslováquia). A análise jackknife forneceu forte suporte para todos os eventos de migração, com P < 3 × 10 −10 em todos os casos.
Em seguida, repetimos a análise para um conjunto de dados reduzido obtido pela exclusão de populações envolvidas em eventos de miscigenação fora da Eurásia ou entre raças de cães (coiotes, lobos de Minnesota, huskies do Alasca, eurasiers e raças de cães do tipo spitz europeu). Nesta nova análise, o suporte bootstrap para a ramificação do lobo Taimyr após os lobos norte-americanos caiu de 100% para 56% (Fig. 4B ), indicando que as posições relativas dessas duas linhagens podem ser influenciadas pela mistura de lobos norte-americanos com coiotes. Esta nova análise inferiu o fluxo gênico dos chacais dourados para o lobo Taimyr e dos chacais dourados para o ancestral comum dos lobos da Ásia Ocidental/Central e europeus. Também identificamos o fluxo gênico de Yakutian para lobos norte-americanos e de lobos da Ásia Ocidental para cães da Ásia Ocidental (Fig. 4B , Fig. Complementar 7 ). Todos os eventos de fluxo gênico inferido tiveram suporte estatístico de P < 0,03, exceto para o fluxo gênico inferido de cães europeus para Terrier tibetano. Uma análise adicional, onde incluímos coiotes, mas excluímos outras populações e raças mencionadas acima, deu resultados consistentes com os descritos acima, mas o fluxo gênico de chacais dourados não foi mais detectado (Fig. 8 complementar ).
A análise, incluindo apenas canídeos originários da Ásia, e assumindo 10 bordas de migração, revelou o fluxo gênico da linhagem Taimyr para a linhagem ancestral de raças de cães do Ártico: Husky Siberiano, Malamute do Alasca e Greenland Sledge Dog. As raças árticas também receberam fluxo gênico de lobos modernos do Leste Asiático e da linhagem ancestral de cães asiáticos de vida livre (Fig. 9 complementar ).
Mistura inferida a partir da análise de blocos de ancestralidade em LAMP
A proporção de blocos cromossômicos de ancestralidade canina em lobos individuais estimada em LAMP 34 (assumindo 10 gerações desde a mistura) variou entre 0 e 27%, e a proporção de blocos cromossômicos de ancestralidade lupina em cães individuais variou entre 0 e 7% (Tabela 1 , Tabela Complementar 5 , Fig. Complementar 10 ). O cão irlandês neolítico tinha 0,17% da ancestralidade estimada do lobo. As raças de cães-lobo conhecidas por se originarem da recente mistura mediada por humanos entre lobos e cães tinham uma proporção considerável de ancestralidade de lobo: 11-12% no cão-lobo da Checoslováquia e 18-33% no cão-lobo de Saarloos. Cinco canídeos selvagens, amostrados como lobos, tinham proporções de ancestralidade de lobos entre 35% e 73%.
O antigo lobo Taimyr mostrou 100% de ancestralidade de lobo em 36 cromossomos, enquanto a suposta ancestralidade canina foi detectada nos cromossomos 15 e 23 (22% e 100% de ancestralidade canina, respectivamente; ver Resultados Suplementares, p. 2). A ancestralidade canina estimada média em todos os cromossomos autossômicos foi de 3%. O cromossomo 23 tinha apenas 261 loci SNP retidos para a análise de LAMP (com base no limiar LD r 2 < 0,1), enquanto o intervalo de loci retidos nos cromossomos autossômicos restantes foi de 1701 (no cromossomo 1) a 501 (no cromossomo 38) . A inferência de ancestralidade no cromossomo 23 pode ser afetada pelo número reduzido de loci SNP informativos.
A análise de LAMP da mistura de lobo-chacal dourado mostrou que os lobos modernos carregam não mais do que 0,2% de blocos cromossômicos originados de mistura recente com chacais dourados, e chacais - com uma exceção - carregam 0,1-0,3% de blocos cromossômicos originados de mistura recente com lobos (Tabela 1 , Tabela Suplementar 5 , Fig. Suplementar 11). Um chacal dourado do Cáucaso tinha 1,7% de ancestralidade de lobo em cromossomos autossômicos e exibia de 3 a 21% de ancestralidade de lobo em 7 cromossomos. O lobo Taimyr tinha 2% da ascendência estimada de chacal dourado em cromossomos autossômicos, mas isso resultou de uma grande proporção (48%) de ascendência putativa de chacal dourado inferida em apenas um cromossomo - cromossomo 37. Esse cromossomo tinha um número relativamente pequeno de loci SNP retido para a LÂMPADAanálise (510), mas esse número era típico de outros cromossomos com comprimento semelhante, pois os cromossomos 35-38 tinham menos de 600 loci retidos para a análise. Portanto, a ascendência do chacal dourado inferida para o cromossomo 37 não pode ser atribuída apenas ao pequeno número de loci SNP usados na análise, mas não podemos excluir a possibilidade de que a baixa resolução tenha afetado a inferência de LAMP .
Mistura inferida a partir da análise de blocos de ancestralidade em ELAI
Os resultados da análise do bloco ancestral em ELAI 35 (assumindo 100 gerações de mistura) foram consistentes com os resultados de LAMP para a identificação de híbridos de cão-lobo e retrocruzamentos recentes (Tabela Suplementar 6 , Fig. Suplementar. 12 ). OS RESULTADOS DE ELAI também apoiaram os resultados de LAMP mostrando mistura de cães em populações de lobos da Eurásia, mas nenhuma mistura de cães (com poucas exceções) em lobos norte-americanos, exceto lobos mexicanos. ELAI identificou níveis de mistura mais altos do que LAMPem várias raças que experimentaram hibridação histórica, incluindo Saarloos e cães lobo da Checoslováquia, raças do leste asiático e do Ártico, Eurasier e husky do Alasca (Tabela Suplementar 6 ).
Discussão
A principal divisão filogeográfica entre as populações de lobos cinzentos estudadas ocorreu entre as populações da Eurásia e da América do Norte, consistente com sua separação de longo prazo desde a inundação pós-glacial da Bering Land Bridge. Na Eurásia, a principal divisão filogeográfica ocorre entre os lobos da Ásia Oriental e da Eurásia Ocidental, embora sua distinção possa resultar parcialmente de descontinuidades em nossa cobertura geográfica de amostragem. Entre os lobos norte-americanos, as populações mais distintas foram os lobos mexicanos e os lobos dos Grandes Lagos (representados aqui pelos lobos de Minnesota). A distinção dos lobos mexicanos provavelmente resulta do isolamento geográfico e de um gargalo demográfico 26 , 36. A distinção dos lobos dos Grandes Lagos foi explicada pela mistura considerável de coiotes 26 , 27 , 28 , 37 ou por uma origem evolucionária distinta 38 , 39 (consulte o Material Suplementar para uma lista de referência mais abrangente). Nosso estudo não tem cobertura amostral abrangente dos lobos dos Grandes Lagos e, portanto, não podemos tirar conclusões sobre seu status taxonômico.
Nossa análise TREEMIX mostrou que, quando a possibilidade de fluxo gênico entre coiotes e lobos é considerada, todos os lobos norte-americanos, incluindo lobos mexicanos e de Minnesota, formam um único grupo. A monofilia de todos os lobos norte-americanos é consistente com uma inferência baseada em genomas nucleares inteiros 28 . Um estudo baseado nos genomas mitocondriais também concluiu que os lobos norte-americanos se originaram de um único evento de colonização da Ásia que ocorreu de 23 a 30 Kya, após a formação da Bering Land Bridge 18. Nenhuma das populações de lobos da Eurásia parece ser geneticamente mais próxima dos lobos norte-americanos do que qualquer outra população. Isso é consistente com a inferência de dados do genoma nuclear inteiro, mostrando que todas as populações modernas de lobos da Eurásia tiveram um ancestral comum há cerca de 30 Ky, e se diversificaram somente após a divergência dos lobos norte-americanos 15 .
Após um evento de expansão de alcance, espera-se que o desequilíbrio de ligação nas populações aumente com a distância do centro geográfico de expansão 40 . Todas as populações de lobos da América do Norte tiveram níveis de LD mais altos do que os lobos da Eurásia e mostraram um aumento de LD do norte (Alasca) para o sul (Novo México), consistente com um único evento de expansão da Ásia para a América do Norte através da Beringia. Alternativamente, o aumento de LD pode ser explicado pelos recentes declínios da população de lobos nos Estados Unidos contíguos, que foram especialmente graves nos lobos mexicanos. No entanto, nossos resultados são consistentes com outras linhas de evidência que sugerem uma única expansão para o sul na América do Norte 18. Em contraste, os padrões de LD nas populações eurasianas não apontavam claramente para um modelo fundador serial de expansão da Beríngia para a Eurásia. Embora um único gradiente geográfico de LD fosse esperado no caso de um único evento de expansão fora da Beríngia proposto em 19 , os padrões de LD resultantes de uma expansão populacional poderiam ter sido modificados por eventos subsequentes (como eventos de hibridização e perda recente de diversidade devido à caça extensiva), ocultando a origem da expansão geográfica.
O antigo lobo Taimyr não foi claramente atribuído aos lobos siberianos modernos ou a qualquer outra população regional de lobos. Nossa análise TREEMIX colocou o lobo Taimyr como a linhagem irmã dos lobos eurasianos modernos e cães domésticos, consistente com a inferência original 10. Consequentemente, nossa inferência da estrutura genética da população não revelou agrupamento do lobo Taimyr com qualquer população de lobo moderno, demonstrando sua distinção. Deve-se notar, no entanto, que é improvável que as estimativas obtidas para um único indivíduo antigo sejam representativas de toda a população da qual se originou. Portanto, nossos resultados em relação ao lobo Taimyr devem ser interpretados com cautela. No entanto, eles destacam a importância de realizar análises semelhantes para um número maior de lobos do Pleistoceno, uma vez que seus genótipos estejam disponíveis.
A ambigüidade na posição inferida da linhagem dos lobos Taimyr em relação aos lobos norte-americanos modernos pode resultar da mistura entre lobos norte-americanos e coiotes. Assumir divisões de linhagem puramente dicotômicas na presença de fluxo gênico pós-divisão pode levar a uma reconstrução tendenciosa da história evolutiva das linhagens estudadas. No entanto, a ambigüidade na posição do lobo Taimyr também pode sugerir que ambas as linhagens divergiram dos lobos eurasianos modernos em um momento semelhante. A estimativa demográfica do tempo de coalescência para todos os lobos modernos com base em dados completos da sequência do genoma é de cerca de 32,5 Kya 15 , tornando o lobo Taimyr quase contemporâneo do tempo de divergência entre os lobos modernos da Eurásia e da América do Norte. O início da domesticação do cão foi estimado em 33-29 Kya 15, 41 , logo após a divergência entre lobos norte-americanos e eurasianos 15 . Isso sugere que a diversificação entre as principais linhagens de lobos contemporâneos, a linhagem de Taimyr e a linhagem de cães domésticos ocorreu em um curto espaço de tempo próximo ao início do Último Máximo Glacial (33–26,5 Kya 42 ).
Durante esse período, os lobos experimentaram declínios demográficos consideráveis e mudanças filogeográficas 14 , 15 , 19 , com desaparecimento de ecomorfos distintos, provavelmente devido a declínios e extinções de suas espécies de presas 16 , 43 . Mudanças na densidade e na composição de espécies das comunidades de ungulados podem ter levado a extinções locais, mas também à diversificação de linhagens, pois estavam mudando seus nichos alimentares em novas direções, incluindo a dos comensais humanos. As alterações ecológicas e filogeográficas ocorridas nesse período desempenharam um papel crucial na história evolutiva do lobo cinzento e do cão doméstico.
Nossos resultados mostram que a evolução do gênero Canis foi substancialmente afetada pela hibridização. As proporções exatas de mistura inferidas para populações regionais são propensas à falta de precisão, dado que os modelos de inferência são inerentemente mais simples do que a história evolutiva real dessas populações. Apesar dessa limitação, os modelos de mistura são uma ferramenta útil para inferir o efeito de eventos de fluxo gênico nas trajetórias evolutivas das populações. A ANÁLISE DE MISTURA indicou que os lobos asiáticos contemporâneos têm valores de atribuição positivos, mas pequenos, para o agrupamento composto por chacais dourados, chacais de dorso preto e coiotes (que foram agrupados devido a pequenos tamanhos de amostra; consulte a Fig. 5 suplementar). Apenas o chacal dourado poderia estar envolvido na mistura com os lobos asiáticos, uma vez que as outras duas espécies nunca ocorreram na Eurásia. As proporções de mistura entre lobos modernos e chacais dourados inferidas em LAMP foram menores em comparação com os resultados obtidos de ADMIXTURE e TREEMIX , provavelmente porque a inferência de LAMP foi restrita às últimas 10 gerações. Os resultados de ADMIXTURE e TREEMIX provavelmente refletem eventos de hibridação em um passado mais distante. A inferência da mistura antiga entre lobos e chacais dourados é consistente com resultados anteriores baseados em dados completos da sequência do genoma 15 , 23 , 25. Isso não exclui a possibilidade de eventos de fluxo gênico mais recentes entre essas duas espécies, que também já foram relatados antes 24 , 44 . A distinção entre a mistura antiga e recente requer um maior número de amostras de lobo e chacal dourado das regiões onde seus intervalos se sobrepõem, bem como maior densidade de loci SNP.
A análise TREEMIX inferiu múltiplos casos de fluxo gênico entre lobos e cães, consistente com estudos anteriores, por exemplo , 30 , 45 , 46 , 47 , 48 . Consequentemente, a análise de blocos de ancestralidade implementada em LAMP e ELAI identificou assinaturas de mistura de cães em todas as populações de lobos da Eurásia e em lobos mexicanos. A população de lobos mexicanos foi a única população norte-americana que apresentou miscigenação canina (sem contar indivíduos mestiços isolados detectados em algumas outras populações). Como esta população é pequena e altamente endogâmica 36, os alelos derivados de cães podem ter desviado para uma frequência relativamente alta nesta população após um único evento de hibridização. Alternativamente, esse resultado pode ser devido à alta frequência de alelos representando polimorfismos ancestrais compartilhados entre lobos e cães.
Tanto LAMP quanto ELAI inferiram uma mistura limitada de lobos em raças de cães de origem européia e cães europeus de vida livre, mas níveis mais altos de mistura de lobos em cães de vida livre e de raça pura do leste da Ásia e do Ártico, consistente com a inferência de nosso estudo anterior baseado em um conjunto de dados independente 48 . A mistura no cão neolítico irlandês 49 foi baixa e dentro da faixa dos cães europeus modernos. Também inferimos o fluxo gênico da antiga linhagem de lobos Taimyr para a população ancestral de raças árticas, consistente com Skoglund et al . 10. Dada a distinção da linhagem Taimyr dos lobos modernos, isso implica eventos independentes de introgressão de lobos nos cães árticos ocorrendo em vários períodos de tempo.
Espera-se que o lobo Taimyr esteja livre da mistura de cães, pois foi datado em 35 Kya - o tempo que precede ou é contemporâneo do início do processo de domesticação do cão 10. O padrão inferido a partir da análise do bloco de ancestralidade, com 36 cromossomos tendo inteiramente ancestralidade de lobo e uma alta proporção de ancestralidade canina em dois cromossomos restantes, é inconsistente com a introgressão neutra de alelos derivados de hibridação. Nossos resultados também indicam que a linhagem de lobo Taimyr recebeu fluxo gênico de coiotes e/ou chacais dourados. No entanto, a localização ao norte da Península de Taimyr impede a presença de chacais dourados durante o Pleistoceno Superior, e o coiote nunca ocorreu na Eurásia, tornando improvável um evento de hibridização local. A análise do bloco de ancestralidade não mostrou nenhuma evidência de ancestralidade do chacal dourado no lobo Taimyr, exceto em um cromossomo, tendo quase 50% da ancestralidade inferida do chacal. Novamente, tal padrão não poderia ter resultado de introgressão neutra. Isso pode ser atribuído ao baixo número de loci SNP informativos nos cromossomos onde a ancestralidade exógena foi inferida, combinado com a presença de variação ancestral compartilhada entre as linhagens. Isso sugere que é improvável que erros na inferência de mistura resultantes de informações genéticas limitadas usadas na análise produzam padrões de mistura consistentes em vários cromossomos. Portanto, a análise de blocos de ancestralidade dentro dos cromossomos pode fornecer uma maneira eficiente de identificar erros na inferência de mistura média do genoma. Isso sugere que é improvável que erros na inferência de mistura resultantes de informações genéticas limitadas usadas na análise produzam padrões de mistura consistentes em vários cromossomos. Portanto, a análise de blocos de ancestralidade dentro dos cromossomos pode fornecer uma maneira eficiente de identificar erros na inferência de mistura média do genoma. Isso sugere que é improvável que erros na inferência de mistura resultantes de informações genéticas limitadas usadas na análise produzam padrões de mistura consistentes em vários cromossomos. Portanto, a análise de blocos de ancestralidade dentro dos cromossomos pode fornecer uma maneira eficiente de identificar erros na inferência de mistura média do genoma.
A análise da mistura de canídeos deve ser idealmente baseada em dados de sequência do genoma inteiro para maximizar o número de sítios variáveis usados. No entanto, o custo do sequenciamento de todo o genoma de alta cobertura ainda é relativamente alto, limitando o número de indivíduos que podem ser analisados (por exemplo, Fan et al . 15 incluiu 34 indivíduos e Sinding et al . 28incluiu 40 indivíduos). Nosso estudo mostrou grande variação nas proporções de miscigenação dentro das populações, o que implica que a inferência de miscigenação para indivíduos individuais não é representativa de padrões populacionais amplos. A genotipagem de SNP em todo o genoma permite a geração de dados para um grande número de indivíduos e fornece densidade suficiente de locais variáveis para realizar a análise de blocos de ancestralidade. A aplicação de chips SNP a espécies diferentes daquela usada para projetar o chip carrega o potencial inevitável de viés de apuração. Embora não possamos excluir que isso esteja afetando nossos resultados, nossas análises sugerem que o viés provavelmente é baixo (consulte Informações complementares para uma discussão mais detalhada sobre o viés de apuração). Portanto, a genotipagem de SNP em todo o genoma é atualmente o método ideal para gerar os dados em nível populacional necessários para estimativas de mistura,
Conclusões
Os padrões filogeográficos globais nas populações modernas de lobos foram moldados pela grande mudança filogeográfica que ocorreu perto do final do Pleistoceno e subsequentes eventos de mistura local. As populações de lobos da Eurásia carregam assinaturas de mistura geograficamente difundida com cães e mistura rara com chacais dourados, enquanto as populações de lobos da América do Norte exibem níveis variados de mistura com coiotes e mistura rara com cães. A hibridação introgressiva de populações con-específicas e congenéricas ecologicamente diversas pode ser mal-adaptativa em muitas circunstâncias. No entanto, em períodos de rápida mudança ambiental, tal introgressão poderia ter facilitado a adaptação do lobo às condições ambientais em rápida mudança. Isso também foi sugerido no caso do urso pardo ( Ursus arctos ) e do urso polar (Ursus maritimus ) mistura 50 . A mistura afetou as relações inferidas entre as populações regionais de lobos, enfatizando a importância de contabilizar este processo em estudos filogeográficos, se espécies intimamente relacionadas estão ou estiveram presentes na região de estudo.
Ambos os padrões de desequilíbrio de ligação e a reconstrução das relações de ancestralidade sugerem que os lobos cinzentos norte-americanos (incluindo os lobos mexicanos) se originaram de um único evento de colonização da Ásia, consistente com os resultados de um estudo recente baseado em dados do genoma completo 28 . No entanto, não há evidências claras de um modelo fundador em série da expansão do lobo da Beríngia para a Eurásia. Os lobos do centro-leste da Ásia tiveram o menor desequilíbrio de ligação de todas as populações estudadas, e outras populações da Eurásia não mostraram um gradiente geográfico linear de desequilíbrio de ligação, implicando uma história evolutiva complexa.
Consistentemente, descobrimos que a antiga linhagem de lobos Taimyr não é claramente atribuída aos lobos siberianos modernos ou a qualquer outra população regional de lobos. O legado genético desta linhagem persistiu nas raças de cães árticos como resultado de uma mistura antiga, mas foi diluído pelo fluxo gênico subseqüente dos lobos modernos. A reconstrução das relações de ancestralidade entre as populações de lobos colocou o antigo lobo Taimyr como uma linhagem irmã dos modernos lobos eurasianos e cães domésticos, com uma posição ambígua em relação aos lobos norte-americanos. Essa ambigüidade sugere que a divergência de linhagens levando aos lobos modernos da Eurásia e da América do Norte, ao lobo Taimyr e aos cães domésticos ocorreu em um curto período e provavelmente foi desencadeada por mudanças ambientais associadas ao início do Último Máximo Glacial.
A história evolutiva de outro grande carnívoro holártico, o urso pardo, segue um padrão semelhante de substituição de linhagem global 4 e hibridização introgressiva generalizada de espécies intimamente relacionadas 11 , 50 . Os padrões observados podem, portanto, representar uma resposta evolutiva comum das espécies da megafauna às mudanças ambientais durante a transição Pleistoceno/Holoceno, o que justifica um estudo comparativo incluindo múltiplas espécies existentes e extintas.
Materiais e métodos
conjunto de dados
Nosso conjunto de dados consistia em lobos cinzentos Canis lupus representando as linhagens geograficamente espalhadas pela Eurásia e América do Norte, e excluía táxons geneticamente distintos, como o lobo dourado africano e o lobo do Himalaia, que são considerados subespécies de Canis lupus ou espécies distintas Canis anthus 44 e Canis himalayensis 51 , respectivamente.
Usando o CanineHD BeadChip (Illumina), genotipamos 84 lobos cinzentos de diferentes partes da Ásia: Geórgia, Cáucaso (11 indivíduos), Nagorno-Karabakh, Cáucaso (18 indivíduos), Mongólia (14 indivíduos), Rússia Yakutia-Sakha Republic (38 indivíduos) e Arábia Saudita (dois indivíduos). Dois indivíduos adicionais (Mongólia e Arábia Saudita) amostrados como lobos cinzentos foram geneticamente identificados como cães domésticos, e um indivíduo de Nagorno-Karabakh foi identificado como um chacal dourado. Esses indivíduos mal identificados foram incluídos nos conjuntos de dados de cães e chacais. Os lobos modernos foram comparados com o lobo cinzento de Taimyr na Sibéria datado de 34,9 Kya 10 . Usamos os genótipos de SNP que foram extraídos dos dados da sequência do genoma completo em Skoglund et al . 10para permitir a comparação com o conjunto de dados de cães de raça pura de Vaysse et al . 52 . Além disso, genotipamos amostras de 44 lobos cinzentos modernos da Bulgária, 7 chacais dourados da Eurásia ( Canis aureus; 3 da Bulgária e 4 do Cáucaso) e 3 chacais de dorso preto ( Canis mesomelas ) da Namíbia. Todas essas amostras foram genotipadas em 167.989 locos SNP autossômicos e 5.660 locos SNP do cromossomo X.
Também incluímos amostras de outros estudos publicados, todos genotipados usando o CanineHD BeadChip:
Lobos europeus e norte-americanos de Vaysse et al . 52 ;
Lobos europeus de Stronen et al . 53 ;
Lobos norte-americanos, cães mestiços e coiotes ( Canis latrans ) de Cronin et al . 54 ;
Lobos mexicanos de Fitak et al . 55 ;
cães domésticos de raça pura de Vaysse et ai . 52 ;
cães eurasianos de vida livre e cães de raça pura de Pilot et al . 30 ;
cães de vida livre, raças de cães asiáticos e um cão doméstico neolítico da Irlanda datado de 4,8 Kya de Frantz et al . 49 ;
Huskies do Alasca de Vernau et al . 56 , incluído no Fitak et al . 55 conjuntos de dados;
Como amostras pequenas das duas espécies de chacais influenciaram os resultados da análise da estrutura populacional, posteriormente produzimos dados adicionais para 94 chacais dourados de nove países europeus e do Cáucaso e 6 chacais de dorso negro da Namíbia e os incluímos em análises selecionadas. A distribuição geográfica dos canídeos selvagens e de vida livre estudados é apresentada na Fig. 1 , e a Tabela Complementar 1 contém a lista de todas as amostras.
Todos os conjuntos de dados incluídos nas análises foram gerados usando CanineHD BeadChip, o que facilitou sua fusão. A fusão de conjuntos de dados independentes pode ser potencialmente comprometida por incompatibilidades entre genótipos de diferentes conjuntos de dados (efeito de lote). No entanto, a maioria dos bancos de dados listados acima foram gerados usando o método de chamada SNP “TOP/BOT”, que foi projetado para garantir relatórios uniformes de designação de fita e orientação em diferentes conjuntos de dados (Illumina, Inc. 2006). Os conjuntos de dados que foram relatados usando o método de chamada SNP “forward” foram modificados para caber nos conjuntos de dados restantes, invertendo os loci incompatíveis com base na lista fornecida pelo Illumina. Observamos que indivíduos originários de diferentes conjuntos de dados, mas representando as mesmas raças de cães ou populações de lobos, foram agrupados em MISTURAe análises PCA, que demonstram a fusão correta dos conjuntos de dados (tabela complementar 8 ). Alguns dos conjuntos de dados publicados relataram um conjunto reduzido de loci SNP em comparação com o número total incluído no chip SNP usado e não relatou loci do cromossomo X. Portanto, o conjunto de dados mesclado incluiu os dados de 106.549 loci SNP autossômicos. Removemos deste conjunto de dados todos os indivíduos com mais de 20% de dados ausentes, exceto o lobo Taimyr.
Aprovação ética
O estudo obteve a aprovação do Comitê de Ética da Faculdade de Ciências da Universidade de Lincoln (CoSREC365). As amostras de tecido usadas neste estudo foram obtidas de indivíduos que foram mortos como resultado de caça legal ou em colisões de veículos. Nenhum animal foi sacrificado para o propósito desta pesquisa. Todos os métodos foram realizados de acordo com as diretrizes da American Society of Mammalogists sobre o uso de animais selvagens em pesquisa 57 e com todas as diretrizes e regulamentos sobre coleta de amostras de animais selvagens nos países onde as amostras foram coletadas e analisadas.
Estimativa de heterozigosidade da população e desequilíbrio de ligação
Usamos o conjunto de dados descrito acima para calcular a heterozigose observada e esperada em populações regionais de lobos, usando PLINK 58 . Usamos o mesmo software para avaliar o desequilíbrio de ligação em populações regionais de lobos calculando o coeficiente de associação genotípica par a par do genoma r 2 entre alelos de loci SNP autossômicos com MAF > 0,15 e contendo não mais que 10% de dados ausentes. Como as estimativas de desequilíbrio de ligação dependem do tamanho da amostra 59 , realizamos os cálculos para 10 indivíduos não aparentados por população, selecionados aleatoriamente se o tamanho total da amostra fosse maior. O parentesco entre os indivíduos foi avaliado com base nas distâncias IBS (identidade por estado) calculadas no PLINK. Excluímos a população da Arábia Saudita desses cálculos devido ao tamanho da amostra muito pequeno.
Análise da estrutura da população
No conjunto de dados mesclado, havia grandes diferenças nos tamanhos das amostras entre as populações, o que poderia enviesar a inferência da estrutura genética da população 60 . Conforme recomendado por Meirmans 61, reduzimos os tamanhos de amostra de populações maiores removendo indivíduos com mais de 20% de dados ausentes e, em seguida, removendo indivíduos em excesso aleatoriamente para obter tamanhos de amostra comparáveis de diferentes populações de lobos e cães, bem como número total semelhante de lobos e cães . O conjunto de dados final incluiu 697 indivíduos: 83 asiáticos, 108 lobos europeus e 119 norte-americanos, 160 cães de vida livre, 180 cães de raça pura, o cão irlandês neolítico, três cães-lobo da Checoslováquia, dois cães-lobo de Saarloos, 29 coiotes, oito chacais dourados e três chacais de dorso negro. O lobo Taimyr foi mantido no conjunto de dados, apesar de conter 57% dos dados ausentes (veja abaixo).
Antes da análise da estrutura da população, podamos o conjunto de dados de loci em alto desequilíbrio de ligação. Isso foi obtido removendo loci com alelos com coeficiente de associação genotípica par a par em todo o genoma r 2 ≥ 0,5, calculado em PLINK em 50 janelas deslizantes SNP, com um tamanho de passo de 10 SNPs. Também podamos loci invariantes e aqueles com frequência de alelos menores (MAF) muito baixa (<0,01), bem como loci com mais de 10% de dados ausentes. Isso resultou em um conjunto de dados de 90.554 loci. A poda foi realizada com base na variação em todo o conjunto de dados.
A sequência completa do genoma do antigo lobo Taimyr teve baixa cobertura (1,03x), mas a genotipagem SNP foi realizada apenas para loci que tiveram o Phred Quality Score mínimo de 30 10 , o que corresponde a 99,9% de precisão de chamada de base. Como resultado, genótipos confiáveis estavam disponíveis para 41,9% do número total de 8.686.809 loci SNP conhecidos para cães e para 45.735 (42,9%) de loci SNP de 106.549 loci totais no conjunto de dados mesclado.
Uma alta porcentagem de dados ausentes pode afetar as estimativas de diferenciação genética de um indivíduo de populações com as quais ele é comparado. Portanto, criamos um novo conjunto de dados incluindo apenas loci que continham dados para o lobo Taimyr, tinham MAF > 0,01 e estavam livres de LD forte (r 2 ≤ 0,5). Isso resultou em um conjunto de dados de 42.320 loci.
Ambos os conjuntos de dados foram analisados usando a abordagem de agrupamento de máxima verossimilhança implementada no ADMIXTURE 31 . A ANÁLISE DE MISTURA foi executada para valores K (o número de clusters) variando de 1 a 10, usando o critério de término de execução padrão, que interrompe as iterações quando o aumento na verossimilhança logarítmica entre as iterações é menor que 10 −4 . O K ótimo foi identificado como o valor associado ao menor erro de validação cruzada em comparação com outros valores de K. Este erro foi avaliado em um procedimento de validação cruzada 31, onde todos os genótipos observados (loci) foram divididos em dez partições de tamanho aproximadamente igual, então todos os genótipos dentro de cada partição foram convertidos em ausentes iterativamente e as execuções foram repetidas para os conjuntos de dados reduzidos.
Também realizamos uma análise de componentes principais (PCA) usando o pacote SMARTPCA do software EIGENSOFT 62 para visualizar os componentes dominantes da variabilidade no conjunto de dados. As relações inferidas entre as populações modernas foram consistentes entre o conjunto de dados de 90.554 loci e o conjunto de dados reduzido de 42.320 loci, mas os resultados para o lobo Taimyr diferiram consideravelmente (ver Resultados). Portanto, em todas as análises posteriores, usamos o conjunto de dados de 42.320 loci que não continha nenhum dado ausente para o lobo Taimyr.
Análise de relações de ancestralidade e padrões de miscigenação
Reconstruímos as relações de ancestralidade entre as populações de lobos e avaliamos a presença de fluxo gênico de cães usando o software TREEMIX 33 . O conjunto de dados usado nesta análise consistiu em lobos da Ásia, Europa e América do Norte, cães de vida livre dos mesmos continentes, cães de raça pura de origem européia e do leste asiático, coiotes e chacais dourados. Chacais de dorso preto foram usados como grupo externo.
As relações de ancestralidade foram reconstruídas assumindo ou nenhum fluxo gênico pós-divergência entre as populações, ou um número definido (5 ou 10) bordas de migração (eventos de fluxo gênico) inferidos de acordo com a ordem de sua intensidade. O número máximo inferido de 10 bordas de migração resultou de um trade-off entre a possibilidade de detectar eventos de fluxo gênico de intensidade moderada e manter a clareza do gráfico resultante. A análise foi baseada no conjunto de dados podado por LD, e as árvores foram construídas usando blocos de 100 loci SNP em vez de loci individuais, para explicar ainda mais o LD. O suporte do nó foi avaliado por meio de 100 réplicas de bootstrap, geradas por blocos de reamostragem de 100 SNPs. Realizamos uma análise jackknife para avaliar se a inclusão de cada borda de migração no modelo melhorou significativamente o ajuste desse modelo aos dados. Além da análise incluindo o conjunto completo de populações, realizamos uma análise incluindo apenas canídeos modernos da Ásia e o lobo Taimyr, para identificar os eventos de fluxo gênico entre suas respectivas linhagens.
Análise de blocos de ancestralidade usando o software LAMP
Utilizamos o software LAMP 34 para avaliar a presença de blocos cromossômicos originários da hibridação em canídeos individuais. Selecionamos este software porque ele permite estimar blocos de ancestralidade sem definir a priori populações ancestrais não miscigenadas. Como as populações de lobos da Eurásia mostram evidências de mistura anterior com cães 15 , 48 , e algumas populações de cães de vida livre e raças antigas de cães mostram assinaturas de mistura de lobos 12 , 30 , 46 , a identificação prévia de indivíduos não misturados não é possível. na LÂMPADA, a identificação de populações ancestrais é integrada à análise de miscigenação, e essa abordagem exclusiva tornou esse software ideal para a análise de nosso conjunto de dados.
A análise da mistura de cão-lobo foi baseada no conjunto de dados contendo 461 genótipos de lobo (todos os indivíduos com menos de 20% de dados ausentes) e 622 genótipos de cães, incluindo todos os cães de vida livre e um representante de cada raça moderna (para evitar super-representação de raças de cães). Este conjunto de dados foi filtrado para incluir apenas os loci SNP que foram genotipados para o lobo Taimyr, foram variáveis no conjunto de dados (MAF > 0,01) e com não mais de 10% de dados ausentes. Na análise de LAMP , este conjunto de dados foi subseqüentemente podado para loci com alelos em forte desequilíbrio de ligação (r 2 > 0,1).
A proporção de mistura de 0,43:0,57 foi determinada com base nas frequências dos genótipos de lobo e cão. Essa proporção acomodou um cenário conservador onde nenhum indivíduo é miscigenado. Usamos uma taxa de recombinação de 1e-10 e fração de sobreposição entre janelas adjacentes (offset) de 0,2. Assumimos 10 gerações desde a miscigenação, porque o poder de detectar a miscigenação recente diminuiu quando eventos de miscigenação mais distantes foram assumidos (isto é, a suposição de 100 gerações desde a miscigenação resultou em altas estimativas de miscigenação para todos os indivíduos).
Também realizamos uma análise da mistura lobo - chacal dourado. Para tanto, foram utilizados todos os genótipos de chacais dourados (8 indivíduos) e lobos eurasianos (122 indivíduos) com taxa de genotipagem acima de 80%. Assumimos uma proporção de mistura de 0,06:0,94, que era a proporção de chacais para lobos. Os demais parâmetros foram os mesmos da análise anterior.
Análise de blocos de ancestralidade usando o software ELAI
Realizamos uma análise adicional do bloco de ancestralidade usando o software ELAI 35 . Enquanto LAMP assume um episódio de mistura em um número especificado de gerações antes do presente, ELAI implementa um modelo assumindo mistura contínua ao longo de várias gerações, o que é mais realista para mistura de cão-lobo. O ELAI não requer um mapa de recombinação, mas estima as taxas locais de recombinação como parte da análise de ancestralidade, garantindo alta precisão. Ao contrário DE LAMP, ELAI não requer loci de filtragem para desequilíbrio de ligação. Além disso, ELAIexplica a presença de subestrutura populacional dentro dos grupos de mistura. Para esta análise, assumimos a mistura entre lobos e cães, representando dois grupos principais, ao longo de 100 gerações, e a presença de 10 grupos de camada inferior (5 vezes o número de grupos de nível superior, conforme recomendado em Guan 35 ). Parâmetros do modelo oculto de Markov foram usados para estimar 20 passos de maximização esperados. Indivíduos não miscigenados tiveram que ser identificados a priori , o que foi feito por meio do LAMPresultados descritos acima. Os indivíduos foram classificados como não miscigenados com base nas proporções de miscigenação estimadas <0,001 para cães e <0,01 para lobos; usamos limiares diferentes para obter números representativos semelhantes de cães (304) e lobos (334) classificados como não miscigenados. Os limites são muito baixos em ambos os casos e, portanto, a precisão da identificação de indivíduos não miscigenados não é comprometida, nem para cães nem para lobos.
Disponibilidade de dados
Os genótipos de canídeos produzidos neste estudo e a informação geográfica associada podem ser acedidos através do seguinte link: https://data.mendeley.com/datasets/4k3yrn3brm/draft?a=1de061e5-6af3-4093-aabe-d45ea8a2dec4 .
Referências
Lorenzen, ED et ai . Respostas espécie-específicas da megafauna do Quaternário Superior ao clima e aos humanos. Nature 479 , 359–364, https://www.nature.com/articles/nature10574 (2011).
Shapiro, B. et ai . Ascensão e queda do bisão da estepe beringiano. Science 306 , 1561–1565, https://doi.org/10.1126/science.1101074 (2004).
PUBLICIDADESCASPubMedGoogle ScholarCampos, PF et ai . Antigas análises de DNA excluem os humanos como a força motriz por trás da dinâmica populacional do boi-almiscarado ( Ovibos moschatus ) do Pleistoceno tardio. Proc. Natl Acad. Sci USA 107 , 5675–5680, https://doi.org/10.1073/pnas.0907189107 (2010).
PUBLICIDADESPubMedPubMed CentralGoogle ScholarAnijalg, P. et ai . Migrações em larga escala de ursos marrons na Eurásia e na América do Norte durante o Pleistoceno Superior. J. Biogeog. 45 , 394–405, https://doi.org/10.1111/jbi.13126 (2018).
Google ScholarStiller, M. et ai . Definhando – 25.000 anos de declínio genético precederam a extinção dos ursos das cavernas. Mol. Biol. Evolução 27 , 975–978, https://doi.org/10.1093/molbev/msq083 (2010).
CASPubMedGoogle ScholarPalkopoulou, E. et ai . Genomas completos revelam assinaturas de declínios demográficos e genéticos no mamute-lanoso. atual Biol. 25 , 1395–1400, https://doi.org/10.1016/j.cub.2015.04.007 (2015).
CASPubMedPubMed CentralGoogle ScholarErsmark, E. et ai . Demografia populacional e diversidade genética no leão da caverna do Pleistoceno. Quaterna Aberta. 1 , 1–14, https://doi.org/10.5334/oq.aa (2015).
Google ScholarCooper, A. et ai . Eventos abruptos de aquecimento levaram à renovação da megafauna holártica do Pleistoceno Superior. Science 349 , 602–606, https://doi.org/10.1126/science.aac4315 (2015).
PUBLICIDADESCASPubMedGoogle ScholarRacimo, F., Sankararaman, S., Nielsen, R. & Huerta-Sánchez, E. Evidência de introgressão adaptativa arcaica em humanos. Nat. Rev. Genet. 16 , 359, https://doi.org/10.1038/nrg3936 (2015).
CASPubMedPubMed CentralGoogle ScholarSkoglund, P., Ersmark, E., Palkopoulou, E. & Dalen, L. O genoma do lobo antigo revela uma divergência inicial de ancestrais de cães domésticos e mistura em raças de alta latitude. atual Biol. 25 , 1515–1519, https://doi.org/10.1016/j.cub.2015.04.019 (2015).
CASPubMedGoogle ScholarBarlow, A. et ai . Sobrevivência genômica parcial de ursos das cavernas em ursos marrons vivos. Nat. Eco. Evolução 2 , 1563–1570, https://doi.org/10.1038/s41559-018-0654-8 (2018).
PubMedPubMed CentralGoogle ScholarMiao, B., Wang, Z. & Li, Y. A análise genômica revela adaptação à hipóxia no mastim tibetano pela introgressão do lobo cinzento do planalto tibetano. Mol. Biol. Evolução 34 , 734–743, https://doi.org/10.1093/molbev/msw274 (2017).
CASPubMedGoogle ScholarSchweizer, RM et ai . Seleção natural e origem de um alelo melanístico em lobos cinzentos norte-americanos. Mol. Biol. Evolução 35 , 1190–1209 (2018).
CASPubMedPubMed CentralGoogle ScholarPilot, M. et ai . Assinaturas em todo o genoma de gargalos populacionais e seleção diversificada em lobos europeus. Hereditariedade 112 , 428–442 (2014).
CASPubMedGoogle ScholarFan, Z. et ai . Padrões mundiais de variação genômica e mistura em lobos cinzentos. Genoma Res. 26 , 163–173, https://doi.org/10.1101/gr.197517.115 (2016).
CASPubMedPubMed CentralGoogle ScholarLeonard, JA et ai . Extinções da megafauna e o desaparecimento de um lobo ecomorfo especializado. atual Biol. 17 , 1146–1150, https://doi.org/10.1016/j.cub.2007.05.072 (2007).
CASPubMedGoogle ScholarPilot, M. et ai . História filogeográfica de lobos cinzentos na Europa. BMC Evol. Biol. 10 , 104, https://doi.org/10.1186/1471-2148-10-104 (2010).
CASPubMedPubMed CentralGoogle ScholarKoblmüller, S. et ai . Genomas mitocondriais inteiros iluminam antigas dispersões intercontinentais de lobos cinzentos ( Canis lupus ). J. Biogeogr. 43 , 1728–1738, https://doi.org/10.1111/jbi.12765 (2016).
Google ScholarLoog, L. et ai . Os lobos modernos traçam sua origem a uma expansão do final do Pleistoceno da Beríngia. bioRxiv , 370122, https://doi.org/10.1101/370122 (2018).
Freedman, AH & Wayne, RK Decifrando a origem dos cães: de fósseis a genomas. Ana. Rev. Anim. Biosci. 5 , 281–307, https://doi.org/10.1146/annurev-animal-022114-110937 (2017).
Google ScholarLeonard, JA, Echegaray, J., Randi, E. & Vilà, C. Impacto da hibridização com cães domésticos na conservação de canídeos silvestres em cães de vida livre e na conservação da vida selvagem . (ed. Gompper, ME) 170–184. (Oxford University Press, 2014).
Lescureux, N. & Linnell, JD Irmãos guerreiros: As complexas interações entre lobos ( Canis lupus ) e cães ( Canis familiaris ) em um contexto de conservação. Biol. Conserva 171 , 232–45, https://doi.org/10.1016/j.biocon.2014.01.032 (2014).
Google ScholarFreedman, AH et ai . O sequenciamento do genoma destaca a história inicial dinâmica dos cães. PLoS Genet. 10 , e1004016, https://doi.org/10.1371/journal.pgen.1004016 (2014).
CASPubMedPubMed CentralGoogle ScholarMoura, AE et ai . Caça não regulamentada e recuperação genética de um grave declínio populacional: o caso preventivo dos lobos búlgaros. Conserva Genet. 15 , 407–415, https://doi.org/10.1007/s10592-013-0547-y (2014).
Google ScholarGopalakrishnan, S. et ai . O fluxo gênico interespecífico moldou a evolução do gênero. Canis. atual Biol. 28 , 3441–3449 (2018).
CASPubMedGoogle ScholarvonHoldt, BM et ai . Uma perspectiva de todo o genoma sobre a história evolutiva de enigmáticos canídeos parecidos com lobos. Genoma Res. 21 , 1294–1305, https://doi.org/10.1101/gr.116301.110 (2011).
CASPubMedPubMed CentralGoogle ScholarvonHoldt, BM et ai . A análise da sequência do genoma inteiro mostra que duas espécies endêmicas de lobo norte-americano são misturas do coiote e do lobo cinzento. ciência Adv. 2 , e1501714, https://doi.org/10.1126/sciadv.1501714 (2016).
PUBLICIDADESPubMedPubMed CentralGoogle ScholarSinding, MHS et ai . Genômica populacional de lobos cinzentos e canídeos semelhantes a lobos na América do Norte. PLoS Genetics 14 , e1007745 (2018).
PubMedPubMed CentralGoogle ScholarThalmann, O. et ai . Genomas mitocondriais completos de canídeos antigos sugerem uma origem européia de cães domésticos. Science 342 , 871–874, https://doi.org/10.1126/science.1243650 (2013).
PUBLICIDADESCASPubMedGoogle ScholarPilot, M. et ai . Sobre a origem dos mestiços: história evolutiva dos cães de criação livre na Eurásia. Proc. Roy. Sociedade B 282 , 2015–2189, https://doi.org/10.1098/rspb.2015.2189 (2015).
CASGoogle ScholarAlexander, DH, Novembre, J. & Lange, K. Estimativa baseada em modelo rápido de ancestralidade em indivíduos não relacionados. Genoma Res. 19 , 1655–1664, https://doi.org/10.1101/gr.094052.109 (2009).
CASPubMedPubMed CentralGoogle ScholarGarcia-Erill, G. & Albrechtsen, A. Avaliação do ajuste do modelo de proporções de mistura inferidas. bioRxiv , 708883, https://doi.org/10.1101/708883 (2019).
Pickrell, JK & Pritchard, JK Inferência de divisões e misturas populacionais a partir de dados de frequência de alelos em todo o genoma. PLoS Genetics 8 , e1002967, https://doi.org/10.1371/journal.pgen.1002967 (2012).
CASPubMedPubMed CentralGoogle ScholarSankararaman, S., Sridhar, S., Kimmel, G. & Halperin, E. Estimando ancestralidade local em populações misturadas. Sou. J. Hum. Genet. 82 , 290–303, https://doi.org/10.1016/j.ajhg.2007.09.022 (2008).
CASPubMedPubMed CentralGoogle ScholarGuan, Y. Detectando estrutura de haplótipos e ancestralidade local. Genética 196 , 625-642 (2014).
PubMedPubMed CentralGoogle ScholarFredrickson, RJ, Siminski, P., Woolf, M. & Hedrick, PW Resgate genético e depressão por endogamia em lobos mexicanos. Proc. Roy. Sociedade B 274 , 2365–2371 (2007).
Google ScholarSefc, KM & Koblmüller, S. Origem híbrida antiga do lobo oriental ainda não fora da mesa: um comentário sobre Rutledge et al . (2015). Biol. Deixe 12 , 20150834 (2016).
PubMedPubMed CentralGoogle ScholarRutledge, LY, Devillard, S., Boone, JQ, Hohenlohe, PA & White, BN RAD sequenciamento e simulações genômicas resolvem origens híbridas na América do Norte. Canis. Biol. Deixe 11 , 20150303, https://doi.org/10.1098/rsbl.2015.0303 (2015).
CASPubMedGoogle ScholarHohenlohe, PA et ai . Comentário sobre “A análise da sequência do genoma inteiro mostra que duas espécies endêmicas de lobo norte-americano são misturas do coiote e do lobo cinzento”. ciência Adv. 3 , e1602250, https://doi.org/10.1126/sciadv.1602250 (2017).
PUBLICIDADESPubMedPubMed CentralGoogle ScholarMcEvoy, BP, Powell, JE, Goddard, ME & Visscher, PM Dispersão da população humana 'fora da África' estimada a partir do desequilíbrio de ligação e frequências alélicas de SNPs. Genoma Res. 21 , 821–829, https://doi.org/10.1101/gr.119636.110 (2011).
CASPubMedPubMed CentralGoogle ScholarWang, GD et ai . Fora do sul da Ásia Oriental: a história natural dos cães domésticos em todo o mundo. Célula Res . 26 , 21–33, https://www.nature.com/articles/cr2015147 (2016).
Clark, PU et ai . O Último Máximo Glacial. Science 325 , 710–714, https://doi.org/10.1126/science.1172873 (2009).
PUBLICIDADESCASPubMedGoogle ScholarGermonpré, M. et ai . Cães e lobos fósseis de sítios paleolíticos na Bélgica, Ucrânia e Rússia: osteometria, DNA antigo e isótopos estáveis. J. Arqueol. Sci 36 , 473–490, https://doi.org/10.1016/j.jas.2008.09.033 (2009).
Google ScholarKoepfli, K.-P. et al . Evidências genômicas revelam que os chacais dourados africanos e eurasianos são espécies distintas. atual Biol. 25 , 2158–2165, https://doi.org/10.1016/j.cub.2015.06.060 (2015).
CASPubMedGoogle ScholarHindrikson, M., Männil, P., Ozolins, J., Krzywinski, A. & Saarma, U. Contrariando a tendência na hibridização cão-lobo: Primeira evidência da Europa de hibridação entre cadelas e lobos machos. PLoS ONE 7 , e46465, https://doi.org/10.1371/journal.pone.0046465 (2012).
PUBLICIDADESCASPubMedPubMed CentralGoogle ScholarKopaliani, N., Shakarashvili, M., Gurielidze, Z., Qurkhuli, T. & Tarkhnishvili, D. Fluxo gênico entre populações de lobos e cães pastores na Geórgia (Cáucaso). J. Hered. 105 , 345–353, https://doi.org/10.1093/jhered/esu014 (2014).
PubMedGoogle ScholarGalaverni, M. et ai . Desvendando o tempo de mistura, padrões de introgressão e indicadores fenotípicos em uma população de lobos hibridizada. Mol. Biol. Evolução 34 , 2324–2339, https://doi.org/10.1093/molbev/msx169 (2017).
CASPubMedPubMed CentralGoogle ScholarPilot, M. et ai . Mistura generalizada e de longo prazo entre lobos cinzentos e cães domésticos em toda a Eurásia e suas implicações para o estado de conservação dos híbridos. Evolução Appl. 11 , 662–680, https://doi.org/10.1111/eva.12595 (2018).
PubMedPubMed CentralGoogle ScholarFrantz, LA et ai . Evidências genômicas e arqueológicas sugerem uma origem dupla de cães domésticos. Science 352 , 1228–1231, https://doi.org/10.1126/science.aaf3161 (2016).
PUBLICIDADESCASPubMedGoogle ScholarCahill, JA et ai . Evidência genômica de mistura generalizada de ursos polares em ursos marrons durante a última era glacial. Mol. Biol. Evolução 35 , 1120–1129, https://doi.org/10.1093/molbev/msy018 (2018).
CASPubMedGoogle ScholarWerhahn, G. et ai . Evidência filogenética para o antigo lobo do Himalaia: rumo a um esclarecimento de seu status taxonômico com base na amostragem genética do oeste do Nepal. Roy. Sociedade Ciência Aberta. 4 , 170186 (2017).
PUBLICIDADESGoogle ScholarVaysse, A. et ai . Identificação de regiões genômicas associadas à variação fenotípica entre raças caninas usando mapeamento de seleção. PLoS Genet. 7 , e1002316, https://doi.org/10.1371/journal.pgen.1002316 (2011).
CASPubMedPubMed CentralGoogle ScholarStronen, AV et ai . Análises de todo o genoma sugerem que a seleção paralela para características universais pode eclipsar a seleção ambiental local em um carnívoro altamente móvel. Eco. Evolução 5 , 4410–4425, https://doi.org/10.1002/ece3.1695 (2015).
PubMedPubMed CentralGoogle ScholarCronin, MA, Cánovas, A., Bannasch, DL, Oberbauer, AM & Medrano, JF Variação de polimorfismo de nucleotídeo único (SNP) de lobos ( Canis lupus ) no sudeste do Alasca e comparação com lobos, cães e coiotes na América do Norte. J. Hered. 106 , 26–36, https://doi.org/10.1093/jhered/esu075 (2015).
CASPubMedGoogle ScholarFitak, RR, Rinkevich, SE & Culver, M. A análise ampla do genoma de SNPs é consistente com a ausência de ancestralidade de cães domésticos no ameaçado lobo mexicano ( Canis lupus baileyi ). J. Hered. 109 , 372–383, https://doi.org/10.1093/jhered/esy009 (2018).
CASPubMedPubMed CentralGoogle ScholarVernau, KM et ai . A análise de associação em todo o genoma identifica uma mutação no gene do transportador de tiamina 2 (SLC19A3) associado à encefalopatia do Husky do Alasca. PLoS ONE 8 , e57195, https://doi.org/10.1371/journal.pone.0057195 (2013).
PUBLICIDADESCASPubMedPubMed CentralGoogle ScholarSikes, RS Comitê de Cuidados e Uso de Animais da Sociedade Americana de Mammalogistas. 2016 Diretrizes da Sociedade Americana de Mammalogistas para o uso de mamíferos selvagens em pesquisa e educação. J. Mamífero. 97 , 663–688, https://doi.org/10.1093/jmammal/gyw078 (2016).
PubMedPubMed CentralGoogle ScholarPurcell, S. et ai . PLINK: um conjunto de ferramentas para análises de associação de todo o genoma e de ligação baseada na população. Sou. J. Hum. Genet. 81 , 559–575, https://doi.org/10.1086/519795 (2007).
CASPubMedPubMed CentralGoogle ScholarTeare, MD, Dunning, AM, Durocher, F., Rennart, G. & Easton, DF Distribuição de amostragem de medidas de desequilíbrio de ligação resumidas. Ana. Zumbir. Genet. 66 , 223–233 (2002).
CASPubMedGoogle ScholarPuechmaille, SJ A estrutura do programa não recupera de forma confiável a estrutura correta da população quando a amostragem é desigual: a subamostragem e novos estimadores aliviam o problema. Mol. Eco. Res. 16 , 608–627 (2016).
Google ScholarMeirmans, PG A subamostragem revela que a amostragem desequilibrada afeta os resultados da estrutura em um conjunto de dados de várias espécies. Hereditariedade 122 , 276–287 (2019).
CASPubMedGoogle ScholarPrice, AL et ai . A análise de componentes principais corrige a estratificação em estudos de associação em todo o genoma. Nature Genet . 38 , 904–909, https://www.nature.com/articles/ng1847 (2006).
CASPubMedGoogle ScholarJarvis, A., Reuter, HI, Nelson, A. & Guevara, E. Hole-filled SRTM for the globe Versão 4, disponível no CGIAR-CSI SRTM 90 m Database, http://srtm.csi.cgiar.org , https://cgiarcsi.community/data/srtm-90m-digital-elevation-database-v4-1/ (2018).
Boitani, L., Phillips, M., & Jhala, Y. Canis lupus . A Lista Vermelha de Espécies Ameaçadas da IUCN 2018: e.T3746A119623865, https://doi.org/10.2305/IUCN.UK.2018-2.RLTS.T3746A119623865.en , baixado em 15 de setembro de 2018. (2018).
Reconhecimentos
Este artigo é dedicado à memória do Prof. Eduard Yavruyan, co-autor deste estudo que faleceu recentemente. Agradecemos a dois revisores anônimos por seus comentários úteis sobre o manuscrito. Agradecemos a Matthew Cronin e Rob Roy Ramey por compartilhar informações sobre o Cronin et al. (2015) conjunto de dados. Este trabalho foi financiado por doações do Leverhulme Trust (Research Fellowship RF-2017-185 to MP), a Agência Nacional Polonesa de Intercâmbio Acadêmico (NAWA; Bolsa de Retorno Polonês PPN/PPO/2018/1/00037 para MP), a National Science Center (concessão nº 2011/01/B/NZ8/02978 para WB e MP), a Fundação Russa para Pesquisa Básica (concessão nº 18-316-20009/18 para IMO e NM) e o Decanato de Ciências Científicas Pesquisa na King Saud University através do Vice-Reino de Cátedras de Pesquisa (para ANA e OBM).
Informação sobre o autor
Autores e Afiliações
Contribuições
MP desenhou o estudo; MP e AEM realizaram a análise dos dados; WB coordenou o projeto; IMO, NM, ANA, OBM, EGY, NHM, VH, NK, ET, MK, PS e WB contribuíram com amostras ou dados; MP escreveu o manuscrito; AEM, PS e WB forneceram contribuições importantes para a revisão crítica do manuscrito. Todos os autores revisaram o manuscrito.
Nenhum comentário:
Postar um comentário
Observação: somente um membro deste blog pode postar um comentário.