Subsistência denisovana do Pleistoceno Médio e Superior na caverna Baishiya Karst
Nature volume 632 , páginas 108–113 ( 2024 )
Resumo
Dados genéticos e paleoantropológicos fragmentados sugerem que os denisovanos já foram amplamente distribuídos pelo leste da Eurásia 1 , 2 , 3 . Apesar das evidências arqueológicas limitadas, isso indica que os denisovanos foram capazes de se adaptar a uma gama altamente diversificada de ambientes. Aqui integramos análises zooarqueológicas e proteômicas da assembléia faunística do final do Pleistoceno Médio ao Final da caverna Baishiya Karst no planalto tibetano, onde uma mandíbula denisovana e DNA mitocondrial sedimentar denisovano foram encontrados 3 , 4 . Usando zooarqueologia por espectrometria de massa, identificamos um novo espécime de costela de hominídeo que data de aproximadamente 48-32 mil anos atrás (camada 3). A análise proteômica da espingarda atribui taxonomicamente este espécime à linhagem denisovana, estendendo sua presença na caverna Baishiya Karst até o Pleistoceno Superior. Ao longo da sequência estratigráfica, a assembleia faunística é dominada por Caprinae, juntamente com megaherbívoros, carnívoros, pequenos mamíferos e aves. A alta proporção de modificações antrópicas nas superfícies ósseas sugere que os denisovanos foram o principal agente de acumulação faunística. A cadeia operatória de processamento de carcaças indica que os táxons animais eram explorados pela sua carne, medula e peles, enquanto o osso também era utilizado como matéria-prima para a produção de ferramentas. Nossos resultados lançam luz sobre o comportamento dos denisovanos e suas adaptações aos ambientes diversos e flutuantes do final do Pleistoceno Médio e Superior da Eurásia oriental.
Conteúdo semelhante sendo visualizado por outras pessoas
Principal
Análises antigas de DNA de vários fósseis de hominídeos da Caverna Denisova, na Rússia, revelaram a existência de uma linhagem irmã de Neandertais no leste da Eurásia, os chamados Denisovanos. 1 . Com base na ancestralidade genética denisovana presente em diversas populações do Leste, Sul e Sudeste Asiático 5 , 6 , infere-se que os denisovanos eram comuns no leste da Eurásia durante o Pleistoceno Superior 1 . A mandíbula de Xiahe (denominada Xiahe 1) e o DNA mitocondrial sedimentar Denisovan (mtDNA) da caverna Baishiya Karst (doravante, BKC; 3.280 metros acima do nível do mar; dados estendidos Fig. 1 ) na Bacia de Ganjia, no nordeste do planalto tibetano, apóiam esta afirmação 3 , 4 . Juntos, eles mostram que os denisovanos ocuparam BKC desde pelo menos 160 mil anos atrás (ka) até cerca de 60 ka, e possivelmente até cerca de 45 ka. 3 , 4 .
Escavações arqueológicas no BKC revelaram uma estratigrafia bem preservada contendo um rico conjunto lítico e faunístico, que fornece evidências da ocupação de hominídeos de pelo menos cerca de 190 ka a cerca de 30 ka (ref. 4 ). No entanto, a maioria dos sítios do Pleistoceno no Planalto Tibetano, como o sítio Jiangjunfu 01 7 , produziram apenas alguns espécimes ósseos fragmentados. Além do pequeno conjunto faunístico do sítio 151 na Bacia do Lago Qinghai, que foi ocupado durante o último período de degelo 8 , nenhum outro dado zooarqueológico ou paleontológico 9 estão disponíveis para o Pleistoceno Médio e Superior no Planalto Tibetano (seções de Informações Suplementares 1 e 2 ). Portanto, o BKC oferece uma oportunidade única para estudar as estratégias arcaicas de subsistência dos hominídeos e a ecologia da fauna em que estavam inseridos, no planalto tibetano de alta altitude.
Construímos um banco de dados de referência proteômica estendido de espécies de mamíferos presentes dentro ou ao redor do Planalto Tibetano através de cromatografia líquida com análise de espectrometria de massa em tandem (LC-MS/MS) (seção de Informações Suplementares 3 e Dados Suplementares 3 e 4 ), para permitir alta subsequente - zooarqueologia de rendimento por análise de espectrometria de massa (ZooMS) de 1.857 espécimes ósseos e dentários de BKC (Tabela Suplementar 2.1 ). Em seguida, integramos a identificação taxonômica através do ZooMS com dados zooarqueológicos de um maior número de restos faunísticos ( n = 2.567; Tabela Suplementar 2.1 e Dados Estendidos Fig. 2 ), alguns dos quais já foram identificados taxonomicamente através de observações morfológicas. Juntos, este conjunto de dados fornece uma nova imagem da paleoecologia e das estratégias de subsistência dos denisovanos no planalto tibetano.
Composição da comunidade faunística do BKC
Combinando identificações morfológicas e ZooMS, identificamos taxonomicamente 2.005 (78,1%) dos 2.567 espécimes faunísticos analisados (Extended Data Fig. 2 , Supplementary Data 5 e Supplementary Information seção 4 ). Nossos resultados mostram que os caprinos (Caprinae), principalmente bharal ( Pseudois nayaur ), dominam a assembleia faunística (Dados Estendidos Fig. 3 e Informações Suplementares seção 4.2 ). A alta proporção de bovídeos - por exemplo, Caprinae, iaque selvagem ( Bos cf. mutus ) e gazela tibetana ( Procapra cf. picticaudata ) - e equídeos ( Equus sp.) ao longo da estratigrafia revela uma paisagem dominada por gramíneas na Bacia de Ganjia durante o final do Pleistoceno Médio e Superior (Dados Suplementares 5A e Informações Suplementares seção 4.1 ). A presença de espécies florestais arbustivas, como o veado-vermelho ( Cervus elaphus ), o cervo-almiscarado ( Moschus sp.), o esquilo voador de dentes estriados ( Aeretes melanopterus ) e o porco-espinho ( Hystrix cf. subcristata ) (Dados Suplementares 5A ), reflete a presença de habitats florestais-arbustivos em mosaico de pequena escala (seção de informações complementares 4.1 ), semelhante aos atuais contrafortes e vales fluviais da bacia 4 . Além disso, vários carnívoros (por exemplo, a hiena-malhada ( Crocuta crocuta ultima ), o lobo ( Canis lupus ), a raposa tibetana ( Vulpes ferrilata ) e o leopardo-das-neves ( Panthera cf. uncia )) e aves (por exemplo, a águia-real ( Aquila chrysaetos) ) e faisão comum ( Phasianus colchicus )) também estavam presentes (Dados Suplementares 5A ).
Atualmente, pouco se sabe sobre as mudanças na comunidade faunística no Planalto Tibetano durante o Pleistoceno Médio e Superior (seção 1 de Informações Complementares ). Notavelmente, identificamos apenas grandes carnívoros extintos ( Crocuta sp.) e megaherbívoros (rinoceronte lanoso ( Coelodonta sp.)) abaixo da camada 6 (Dados Estendidos Fig. 3 e Figura Complementar 4.2 ). Embora os tamanhos de amostra para camadas mais jovens sejam menores que aqueles para a camada 10 (a faixa etária máxima modelada é de cerca de 109 ka a mais de 225 ka; Tabela Suplementar 2.2 ) e camada 11 (Tabela de Dados Estendidos 1 ), os dados atuais sugerem que há houve uma mudança notável na composição da comunidade faunística em torno do BKC durante a formação das camadas 6 e 5 (a faixa etária máxima modelada é em torno de 60-104 ka, Tabela Suplementar 2.2 ). Além disso, nossos dados também documentam um aumento na proporção de Caprinae ao longo do tempo, juntamente com uma diminuição na proporção de Bos sp. Não podemos dizer se essas mudanças são o resultado de mudanças nas estratégias de forrageamento dos hominídeos ou, mais especificamente, relacionadas a mudanças no ambiente circundante (seção de Informações Suplementares 4.3 ). No entanto, a presença persistente de bovídeos e equídeos (Figura 4.2 Complementar ) combinada com pequenas variações na diversidade taxonômica (Figura 4.3 Complementar ) ao longo da estratigrafia sugere um ambiente aberto geralmente estável na Bacia de Ganjia.
Os hominídeos eram os principais acumuladores
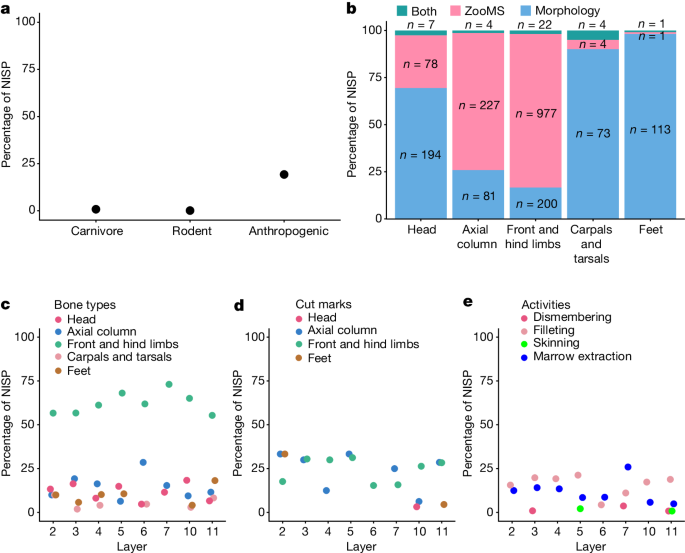
As superfícies das amostras ósseas do BKC estão muito bem preservadas, e a maioria das amostras ( n = 1.616, 88,7% de n = 1.821) estão nos estágios de intemperismo 1 ou 2, fornecendo evidências limitadas de intemperismo subaéreo. Foram identificados vestígios de atividades roedoras, carnívoras e antrópicas (seção de informações complementares 5.2 e dados estendidos Fig. 4a ). A roedura de roedores é limitada ( n = 3, 0,1%; Fig. 1a ), com uma proporção ligeiramente maior de ossos modificados de carnívoros ( n = 16, 0,8%; Fig. 1a ), embora os coprólitos carnívoros estejam ausentes. Por outro lado, uma proporção maior da assembleia faunística exibe evidências de modificações antrópicas ( n = 386, 19,3%; Fig. 1a ). A maioria desses espécimes foi identificada através do ZooMS (Extended Data Fig. 4b ). Marcas de corte e entalhes de percussão foram identificados em ossos de herbívoros e alguns grandes carnívoros (por exemplo, Crocuta sp.) (Tabela Suplementar 5.2 ). A maior proporção de ossos modificados antropogenicamente e a presença de artefatos de pedra em cada camada 4 sugere que a assembleia faunística do BKC se acumulou principalmente através de atividades de hominídeos (seção 5.2 de Informações Complementares ).
a , Porcentagens de espécimes identificados taxonomicamente ( n = 2.005) com modificações carnívoras, roedoras ou antropogênicas. NISP, número de espécimes identificados. b , Porcentagens de espécimes identificados taxonomicamente dentro de diferentes tipos de ossos usando morfologia, ZooMS ou ambos simultaneamente (Dados Suplementares 5 ). Os tipos ósseos são baseados em observações morfológicas. O número mostrado em cada barra é o NISP correspondente envolvido no cálculo. c , Porcentagens de espécimes de Caprinae de diferentes tipos de ossos em cada camada. d , Porcentagens de espécimes de Caprinae com marcas de corte em diferentes tipos de osso em cada camada. e , Porcentagens de espécimes de Caprinae com modificações antrópicas indicando diferentes atividades de processamento de carcaça em cada camada. Para a e c – e , os valores representam porcentagens do NISP que se enquadram nas respectivas categorias.
Extensas atividades antropogênicas
Espécimes morfologicamente identificáveis são derivados em grande parte de fragmentos de cabeça, carpos, tarsos e ossos do pé associados (Fig. 1b ). O ZooMS aumentou nossas identificações taxonômicas e, subsequentemente, nos permitiu identificar com sucesso um número maior de fragmentos axiais e da diáfise dos membros anteriores ou posteriores (Fig. 1b ), que geralmente são sub-representados nas representações do perfil esquelético 10 , 11 . Como resultado, a integração de conjuntos de dados identificados morfologicamente e pelo ZooMS revela uma composição mais completa da estrutura do elemento esquelético para todo o conjunto 12 , 13 , 14 , 15 .
No conjunto de dados integrado (Tabela de Dados Estendida 1 ), Caprinae não está apenas presente em cada camada, mas também representada por todas as porções do esqueleto (cranial, axial, membros anteriores e posteriores e ossos do pé) na maioria das camadas (Fig. 1c ). A extensão da atividade antrópica é onipresente entre os herbívoros ao longo do tempo e consistentemente alta para os Caprinae (cerca de 20-40% para todos os espécimes identificados taxonomicamente; Figura 5.3 suplementar ). Marcas de corte relacionadas às práticas de filetagem em espécimes de Caprinae são as mais frequentes em toda a estratigrafia, exceto nas camadas 6 e 7, nas quais dominam os entalhes de percussão associados à extração de medula óssea nos ossos dos membros anteriores e posteriores (Fig. 1d, e , Dados Estendidos Fig. 5 ) e Figura Complementar 5.5 . Além de Caprinae, a alta frequência de marcas de corte e entalhes de percussão em vários tipos de ossos de outros herbívoros, incluindo Bos sp., Cervus sp., Equus sp. e Coelodonta sp. (Fig. Complementar 5.3 ), sugere que a aquisição de recursos animais não estava restrita a um determinado táxon.
Modificações antropogênicas também estão presentes em carnívoros, pequenos mamíferos e aves (Fig. 2 e Dados Estendidos Fig. 5 ). Entre os espécimes carnívoros ( n = 102; Dados Suplementares 5C ), marcas de corte e entalhes de percussão estão presentes em vários espécimes ( n = 8) de Crocuta sp., Pantherinae e Canidae (não Vulpes vulpes ou Vulpes ferrilata ) (Tabela Suplementar 5.2 ) . Entre os pequenos mamíferos, foram observados entalhes de percussão em um exemplar da diáfise do rádio de Marmota sp. (camada 9; Fig. 2c ), indicando extração de medula. Além disso, sete Lepus sp. (camadas 3, 4 e 11) e quatro Marmota sp. (camadas 9–11) fragmentos da diáfise dos membros anteriores e posteriores mostram fraturas ósseas recentes, que geralmente são tratadas como vestígios de atividades humanas. Finalmente, entre os restos de aves ( n = 45), marcas de corte ( n = 1; Fig. 2a ) e quebras ósseas frescas ( n = 3) estão presentes em espécimes de águias, mas não em espécimes de outras espécies de aves, incluindo faisões. , codorna ou aquele espécime de coruja. No geral, os dados zooarqueológicos sugerem que os hominídeos BKC utilizavam uma vasta gama de espécies, incluindo grandes herbívoros e, em menor grau, carnívoros, pequenos mamíferos e aves.
a , úmero direito de Aquila chrysaetos (camada 4) com aglomerados de marcas de corte superficial e reto, associados à remoção de penas. b , Crocuta crocuta ultima atlas (camada 10a), com marca de corte oblíqua gerada durante a desarticulação. c , Marmota sp. (ZooMS táxon ID) diáfise do rádio (camada 9), com cicatriz em escamas medulares concoidal negativa (triângulo preto) produzida por ruptura antrópica. d , Um possível retocador (camada 11). Equus sp. P2 inferior direito com um conjunto de marcas de arranhões em sua superfície vestibular. e , Ferramenta óssea expedita (ID do táxon ZoomMS: Caprinae; camada 10b). Esta diáfise do úmero é deliberadamente moldada por percussão direta contínua (indicada por triângulos pretos na imagem ampliada à direita) em sua superfície cortical. Para todos os painéis, as imagens ampliadas (à direita em a , b , d , e e inferior em c ) são ampliações das regiões indicadas com linhas pontilhadas nas imagens principais. Exceto onde indicado, as identificações taxonômicas são provenientes de análise morfológica. Barras de escala, 2 cm ( a – d , imagens principais), 1 cm ( e , imagem principal) e 1 mm (todas as imagens ampliadas).
Identificamos um possível retocador (Fig. 2d ) da camada 11 e três ferramentas ósseas expeditas (Fig. 2e ) das camadas 4, 9 e 10 (Tabela de Dados Estendidos 2 ). O possível retocador é produzido a partir de um dente, identificado como Equus sp. Embora as ferramentas ósseas expeditas sejam derivadas de diáfises ósseas de membros que foram lascadas usando percussão direta, o ZooMS nos permitiu identificar esses ossos como pertencentes a Caprinae e Cervinae ou Gazella sp. (Tabela de dados estendida 2 ). Os artefactos ósseos parecem, portanto, ter sido derivados dos táxons que são dominantes na assembleia faunística do BKC, e não de um foco deliberado numa única espécie. 16 .
Um novo indivíduo denisovano
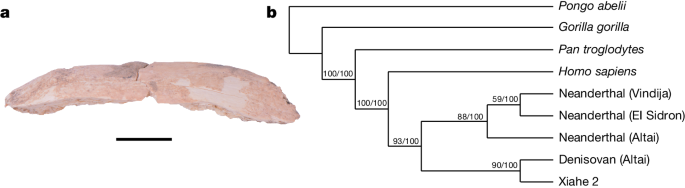
Durante a triagem ZooMS dos fragmentos não identificáveis, uma amostra de costela foi identificada como Homininae (Fig. 3a e Dados Estendidos Fig. 6a ). A amostra contém 14 marcadores peptídicos de colágeno tipo I (COL1) correspondentes a Homininae, bem como um marcador peptídico exclusivo de Hominoidea (Dados Suplementares 4 e Dados Estendidos Fig. 6a ). Considerando a distribuição geográfica atual e passada de outros grandes símios 17 , em particular o gênero Pan , este espécime poderia ser confirmado como Homo sp. Portanto, nomeamos este espécime de hominídeo Xiahe 2 (número de campo, BSY-19-B896-1; número ZooMS, BSY-941). Xiahe 2 foi quebrado em dois pedaços durante a escavação e pertence à parte distal de uma costela (51,5 mm de comprimento). O espécime Xiahe 2 vem da camada 3 do T3, que foi datado 4 a 48–32 ka (Tabela Suplementar 2.2 ). Os valores de desamidação da glutamina de Xiahe 2 (ácido COL1α1 508–519 = 0,52 e ácido COL1α1 435–453 = 0,46) são semelhantes aos de outras amostras da camada 3 e amostras diretamente datadas por radiocarbono em cerca de 50–30 ka (Extended Data Fig ) 6b . No entanto, os valores de desamidação de Xiahe 2 são diferentes daqueles das amostras modernas (Extended Data Fig. 6b ), sugerindo que a idade de Xiahe 2 é consistente com a idade da camada 3 (48-32 ka).
Outras análises proteômicas shotgun fornecem informações mais específicas sobre a atribuição taxonômica de Xiahe 2. Reconstruímos 4.597 posições de aminoácidos para o espécime Xiahe 2 nas 21 sequências de proteínas usadas para análise filogenética (14,5% do alinhamento total da sequência de proteínas concatenadas; Tabela Suplementar 4.1 e Dados Suplementares 6 ). Este é um proteoma consideravelmente maior do que as seis proteínas endógenas que foram utilizadas para a análise filogenética da mandíbula Xiahe 1 3 . Os nós na árvore filogenética têm altos valores de suporte através de métodos de máxima verossimilhança e bayesianos, com Xiahe 2 caindo consistentemente junto com o genoma Denisovan de alta cobertura publicado (Fig. 3b ). Como tal, pode-se determinar que o indivíduo Xiahe 2 é, entre os indivíduos de referência disponíveis, o mais estreitamente relacionado com o indivíduo D3 Denisovan de alta cobertura. A topologia e o posicionamento da amostra Xiahe 2 são semelhantes aos resultados obtidos na análise anterior da mandíbula Xiahe 1 3 . A descoberta do Xiahe 2 Denisovan estende a evidência fóssil da presença de Denisovans desde o final do Pleistoceno Médio até o Pleistoceno Superior em BKC, de acordo com o mtDNA sedimentar Denisovan recuperado da camada 3 em BKC 4 .
Preservação e desamidação de proteínas
A desamidação do colágeno ósseo aumenta gradualmente de cima para baixo através das camadas no BKC, mas com considerável sobreposição entre as camadas (seção 6 de Informações Complementares e Figura 6.2 Complementar ). Os níveis médios de desamidação para as camadas 3, 4 e 5 são altamente semelhantes, assim como os das camadas 6 e 7. A camada 10 e, em particular, a camada 11 apresentam níveis avançados de desamidação de glutamina (Figura 6.2 Complementar ). Nossos dados sugerem que esses resultados não são motivados pela identidade taxonômica, comprimento ósseo, tipo ósseo ou método de extração de proteína (Dados Estendidos Fig. 7 e Figuras Complementares 6.3 – 6.5 ). No entanto, a extensão da desamidação aumenta gradualmente dentro da estratigrafia, de acordo com estimativas de idade geocronológica, bem como com evidências estratigráficas 4 (Dados Estendidos Fig. 8 ). Por outro lado, os valores de desamidação dos espécimes das covas históricas, que contêm uma mistura de vestígios do Pleistoceno e do Holoceno, variam muito. Estas observações estão de acordo com outros locais de cavernas do Pleistoceno, onde a desamidação da glutamina é geralmente mais avançada para espécimes ósseos cronologicamente mais antigos e/ou termicamente mais antigos, em casos em que a estratigrafia abrange quantidades consideráveis de tempo (térmico). 18 , 19 , 20 .
Discussão e conclusão
Estudos anteriores mostram que BKC é atualmente a única caverna bem preservada no planalto tibetano que abrange o final do Pleistoceno Médio ao Final. 3 , 4 (Seção de informações complementares 1 ). As análises da mandíbula e do mtDNA sedimentar de Xiahe 1 revelam que os denisovanos ocuparam a caverna pelo menos por volta de 160 ka, 100 ka (camada 7) e 60 ka (camada 4), e possivelmente até 45 ka (camada final 4) 3 , 4 . A costela Xiahe 2 identificada aqui, e o mtDNA sedimentar denisovano descoberto na camada 3 (ref. 4 ), mostram que a ocupação denisovana ocorreu no local até pelo menos 48–32 ka. As camadas 10 e 11 do BKC fornecem os restos arqueológicos mais ricos da caverna, incluindo mais de 60% dos espécimes ósseos aqui analisados (Tabela Suplementar 2.1 ), mas infelizmente sem quaisquer restos de hominídeos ou mtDNA sedimentar para determinar a identidade biológica dos ocupantes, então distante. No entanto, os valores de desamidação obtidos para Xiahe 1, COL1α1 508–519 (0,04) e COL1α1 435–453 (0,00) estão, exclusivamente, dentro da faixa daqueles observados para amostras ósseas das camadas 10 e 11 (Fig. 4f ). Além disso, Xiahe 1 tem uma idade mínima da série U de cerca de 160 ka (ref. 3 ), que também corresponde à idade cronológica da camada 10 ou inferior (Fig. 4g ). Embora isto não mostre que Xiahe 1 deriva definitivamente das camadas 10, 11 ou de uma camada mais antiga em BKC, mostra que os Xiahe Denisovans foram os ocupantes mais prováveis durante a formação destas camadas.
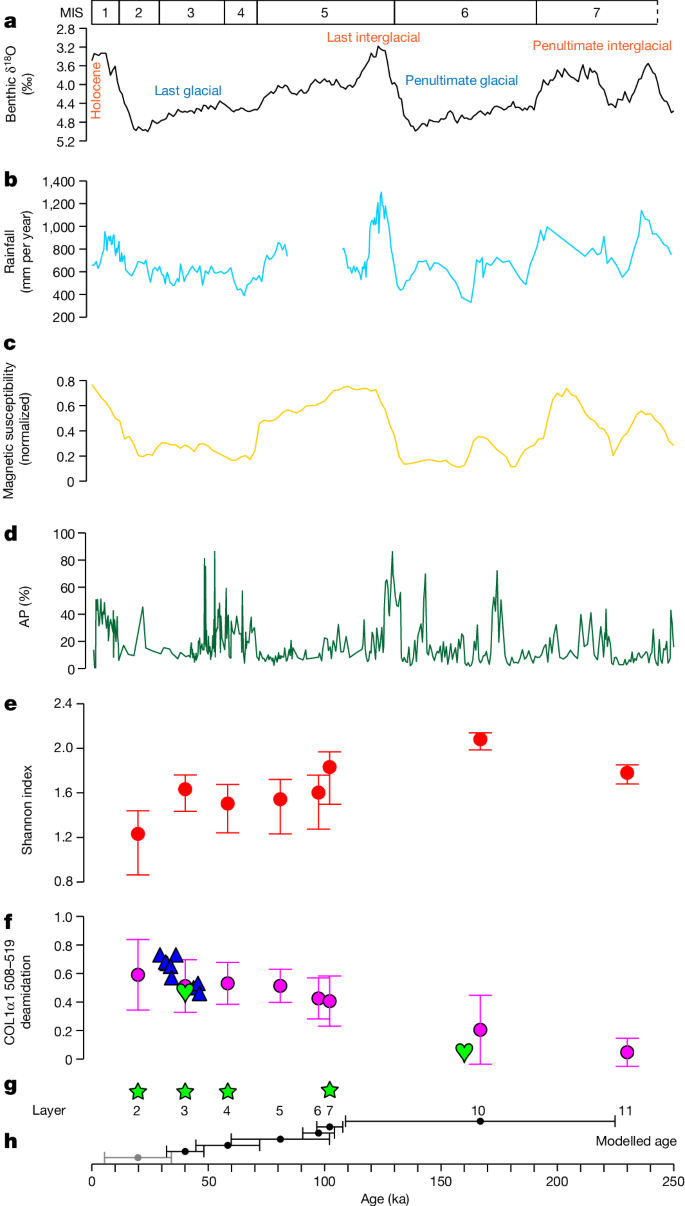
a , pilha bentônica LR04 δ 18 O records 27 . b , 10 Precipitação baseada em amostras de loess de Baoji, norte da China 28 . c , registro de suscetibilidade magnética (normalizado) do planalto chinês de Loess 29 . d , Percentagens de pólen arbóreo (AP) (resolução de cerca de 600 anos) da Bacia Zoige, margem oriental do Planalto Tibetano 30 . e , índice de Shannon entre unidades estratigráficas no BKC. f , Valores de desamidação para o peptídeo COL1α1 508–519 de amostras ósseas. Os pontos roxos e as barras de erro representam os valores médios de desamidação dos ossos de cada camada com intervalos de probabilidade de 68,2%. Os triângulos azuis representam os valores de desamidação de amostras ósseas individuais das camadas 2–3 com datas de radiocarbono ( n = 9, Dados Suplementares 2 ). Os corações verdes representam os valores de desamidação dos dois espécimes Denisovan (Xiahe 1 e 2). g , Camadas estratigráficas e mtDNA sedimentar denisovano. O mtDNA Denisovan extraído de sedimentos nas camadas 2, 3, 4 e 7 é indicado por estrelas verdes. h , Faixa etária modelada de cada camada. Os pontos pretos e os intervalos das barras indicam a idade média modelada e a faixa etária para cada camada (Tabela Suplementar 2.2 ). A faixa etária da camada 2 ainda está em avaliação e atualmente não há datas para as camadas 5 e 11. A estimativa da faixa etária indicada para a camada 5 é baseada no intervalo de idade entre as camadas 4 e 6. Informações cronológicas detalhadas estão disponíveis em Suplemento Seção de informações 2 .
Até o momento, não há evidências da presença de outros hominídeos no BKC para as camadas 3 a 11, nem há evidências de outra ocupação arcaica de hominídeos em outras partes do planalto tibetano do nordeste durante o mesmo período (seção 1 de informações complementares ). Portanto, é razoável supor que os denisovanos ocuparam o BKC pelo menos de cerca de 167 ka (idade média modelada para a camada 10) a cerca de 40 ka (idade média modelada para a camada 3), e possivelmente de mais de 224 ka (idade máxima modelada para a camada 3). 10) a 32 ka (idade mínima modelada para a camada 3) (Tabela Suplementar 2.2 ). Assim, a assembleia faunística BKC documenta o comportamento e a subsistência denisovanos na Bacia de Ganjia durante o último ciclo glacial-interglacial-glacial: o penúltimo período glacial, estágio de isótopo marinho (MIS) 6, representado pelas subcamadas inferiores da camada 10 e possivelmente da camada 11 ; o último período interglacial, MIS 5e, representado pela camada de transição 10/9 (149,2–109 ka) e possivelmente pela camada 10a; e o último período glacial, MIS 4 e 3, representado pelas camadas 4 e 3 (Fig. 4 ).
A análise abrangente da assembleia faunística do BKC mostra que os denisovanos exploraram uma ampla gama de táxons animais que estavam presentes na paisagem dominada por gramíneas ao redor da Bacia de Ganjia. O papel dos Caprinae torna-se cada vez mais proeminente durante a ocupação dos hominídeos, especialmente nas camadas 5 a 2, após o MIS 5, em que os espécimes de Caprinae compõem mais da metade da assembleia faunística (Extended Data Fig. 3 ). Análises de modificações antrópicas em espécimes de Caprinae indicam que a cadeia completa de processamento de carcaça, incluindo o abate sistemático e o uso de material ósseo para ferramentas, está presente no BKC (seção de informações complementares 5.3 ). Além de Caprinae, os restos mortais de megaherbívoros, carnívoros, pequenos mamíferos e aves foram usados de forma semelhante de diversas maneiras. Isto revela que os denisovanos fizeram pleno uso dos recursos animais disponíveis para sobreviver no planalto tibetano de alta altitude durante o último ciclo glacial-interglacial-glacial. Durante os períodos glacial e interglacial, a Bacia de Ganjia pode ter fornecido um refúgio adequado com disponibilidade de recursos relativamente estável, apesar de sua altitude, especialmente em comparação com regiões de maior altitude do planalto tibetano ou com as condições ambientais flutuantes no vizinho planalto chinês de Loess. 21 , 22 , 23 .
Ao comparar o BKC com outros locais denisovanos ou possíveis locais denisovanos, nomeadamente a Caverna Denisova na Rússia e a Caverna Tam Ngu Hao 2 (Cobra) no Laos, descobrimos que as suas assembleias faunísticas são compatíveis com os seus respectivos ambientes geográficos, correspondendo a altitudes elevadas, elevadas. latitude e ambientes tropicais (ou subtropicais) (seção de informações complementares 4.1 ). Nossos resultados, portanto, fornecem evidências de plasticidade paleoecológica e comportamental em denisovanos. Além disso, estes conhecimentos levantam questões sobre a causa e o momento da extinção dos Denisovanos no Planalto Tibetano, bem como sobre as origens das assinaturas genéticas dos Denisovanos nos humanos modernos. 6 , 24 , 25 , 26 .
Métodos
Caverna Cársica de Baishiya
A caverna Baishiya Karst (BKC; 35,45° N, 102,57° E, 3.280 metros acima do nível do mar) está localizada na Bacia de Ganjia, nordeste do planalto tibetano. 4 (Dados estendidos Fig. 1 e seção de informações complementares 2 ). É uma caverna cárstica e fica cerca de 20 m acima do leito do rio Jianglagou, em frente à caverna. A mandíbula Xiahe (Xiahe 1) foi encontrada nesta caverna em 1980 e foi datada em pelo menos 160 ka pela datação da série U da crosta carbonática na mandíbula 3 . O indivíduo Xiahe 1 foi identificado como Denisovan por análise paleoproteômica 3 . Duas unidades conectadas (T2 e T3, 1 m × 2 m, respectivamente) foram escavadas nesta caverna em 2018 e 2019, revelando 11 camadas. A estrutura cronológica para as camadas 2 a 10 construída por luminescência opticamente estimulada (OSL) e métodos de datação por radiocarbono indica que os hominídeos pré-históricos ocuparam a caverna de cerca de 190 ka a cerca de 30 ka (ref. 4 ). O mtDNA denisovano extraído de sedimentos das camadas 4 e 7 indica que o local foi ocupado por denisovanos por volta de 100 ka, cerca de 60 ka e possivelmente até 45 ka (ref. 4 ), fornecendo mais evidências inequívocas da ocupação denisovana no BKC. Além disso, o mtDNA Denisovan também foi recuperado das camadas 2 e 3, e a idade deste ainda está sob cálculo e avaliação detalhados.
Quadro cronológico
Um estudo anterior 4 usou OSL e datação por radiocarbono para estabelecer uma estrutura cronológica para as camadas 2 a 10 de T2. Neste estudo, aplicamos esta estrutura tanto para T2 quanto para T3, mas usamos a faixa etária máxima para representar a idade de cada camada (Tabela Suplementar 2.2 ). de Informações Suplementares Consulte a seção 2 para obter detalhes.
Seleção de amostra
Um total de 3.642 amostras ósseas foram coletadas sistematicamente de T2 e T3 durante escavações em 2018 e 2019, 3.582 das quais foram registradas com coordenadas tridimensionais, todas armazenadas no Laboratório Chave de Sistemas Ambientais da China Ocidental (Ministério da Educação) na Universidade de Lanzhou . Dentre estes, selecionamos quase todos os ossos maiores que 20 mm ( n = 2.407), bem como alguns fragmentos ósseos menores (menores que 20 mm) com características morfológicas ( n = 160) adequadas para análise taxonômica morfológica, totalizando 2.567 espécimes (Tabela Suplementar 2.1 ). Entre esses espécimes, 2.407 espécimes foram descobertos nas camadas 1 a 11, 138 espécimes derivados de duas cavas da era histórica (H1 e H3) e 22 espécimes não possuíam informações específicas da camada. Como as camadas 1 a 10 foram seriamente destruídas pelos dois poços históricos (H1 e H3), apenas partes dessas camadas foram preservadas. 4 , resultando no pequeno número de ossos ( n = 764) coletados das camadas 1–9. Portanto, cerca de 64% da assembleia óssea analisada deriva das camadas 10 e 11 ( n = 1.643). Nesta assembleia, selecionamos 1.857 espécimes para análise do ZooMS, incluindo 53 espécimes com identificações taxonômicas baseadas em análise morfológica para confirmação proteômica de suas atribuições taxonômicas (Tabela Suplementar 2.1 ).
Amostras de referência do ZooMS
Os atuais bancos de dados de referência do ZooMS são dominados pela fauna da Eurásia ocidental, complementados com conjuntos de dados de referência específicos de famílias. Além disso, existe uma variedade de séries de marcadores de massa peptídica prevista a partir de sequências COL1 com base nos recursos genômicos disponíveis. No entanto, um número substancial de táxons de mamíferos que estão potencialmente presentes no BKC não estão representados em nenhum destes recursos. Portanto, coletamos 39 espécimes com informações taxonômicas conhecidas, representando 39 espécies e 29 gêneros, que atualmente habitam a região mais ampla do Himalaia, ou que estiveram presentes nesta região no Pleistoceno ou Holoceno (Dados Suplementares 3 ). Entre esses exemplares de referência, dez são do Pleistoceno ou Holoceno, enquanto outros são exemplares modernos. As 39 amostras de referência também incluíram 6 amostras de espécies para as quais sequências genômicas estão disponíveis, mas para as quais nenhuma impressão digital de massa de peptídeo de colágeno tipo I de espectrometria de massa de ionização por dessorção a laser assistida por matriz de referência (MALDI-TOF MS) foi relatada antes , como o panda gigante ( Ailuropoda melanoleuca ) e o antílope tibetano ( Pantholops hodgsonii ). Espécimes de referência foram coletados na Universidade de Lanzhou, no Instituto de Zoologia, na Academia Chinesa de Ciências (CAS) e no Instituto Noroeste de Biologia do Planalto, CAS (Dados Suplementares 3 ).
Depois de obter impressões digitais de massa peptídica para cada amostra de referência (ver método de extração abaixo), 14 espécies, representando 14 gêneros, foram selecionadas para análise LC-MS/MS para validar (novas) massas de marcadores peptídicos.
Análise zooarqueológica
Elementos esqueléticos de diagnóstico foram identificados usando espécimes comparativos de vertebrados modernos e antigos do Laboratório Chave de Sistemas Ambientais da China Ocidental (Ministério da Educação) da Universidade de Lanzhou e atlas osteológicos comparativos 31 , 32 , 33 , 34 , 35 , 36 , 37 . Os táxons identificados foram classificados em seis classes de tamanho corporal (classe 0 – classe V) com base no peso vivo, seguindo critérios previamente estabelecidos. 38 : classe I ( Marmota himalayana , Lepus oiostolus , Hystrix sp., Mustelidae, Vulpes ferrilata ); classe II ( Procapra sp., Moschus sp., Panthera sp., Canis lupus ); classe III ( Pseudois nayaur , Ovis ammon , Cervus elaphus , Crocuta crocuta ); classe IV ( Equus sp., Bos mutus ); e classe V ( Coelodonta sp.). Para os carnívoros, os animais grandes correspondem à classe II ou classe III, e os animais pequenos correspondem à classe I. Para os herbívoros, os animais mega, grandes, médios e pequenos correspondem às classes V, IV, III ou II e I, respectivamente. Além disso, as aves são classificadas na classe 0.
Cada espécime foi observado sob uma lente portátil de baixa ampliação (20×) usando uma fonte de luz oblíqua, e foi observado e fotografado usando um microscópio digital Keyence VHX-7000N em diferentes ampliações quando necessário. Modificações na superfície óssea, incluindo modificações antrópicas (marcas de corte e corte, marcas de percussão e entalhes e flocos ósseos de impacto), vestígios de roedor de animais (covas, perfurações, pontuações, sulcos e assim por diante), pisoteio e modificações mecânicas modernas produzidas por ferramentas de escavação, foram registrado de acordo com a literatura 39 , 40 , 41 , 42 , 43 . Os estágios de intemperismo da superfície óssea foram classificados em quatro níveis (I-IV) de acordo com os critérios clássicos 39 . Também registramos amostras de ossos queimados com base em mudanças macroscópicas de cor durante a exposição ao fogo. 44 . Os tipos de fratura dos fragmentos ósseos maiores que 20 mm foram registrados e classificados de acordo com critérios previamente descritos 45 . As cinco regiões anatômicas neste artigo são cabeça (incluindo chifre e galhada); coluna axial (incluindo vértebras, costelas e pelve); membros anteriores e posteriores (incluindo escápulas, úmeros, rádios e ulnas, metacarpos, fêmures, tíbias e metatarsos); carpais e tarsos; e pés, com base na estrutura definida em relatório anterior 46 .
Os retocadores ósseos foram identificados de acordo com critérios previamente descritos 47 . Ferramentas ósseas convenientes foram identificadas com base em critérios quantitativos descritos anteriormente 48 , 49 . Especificamente, amostras de fragmentos ósseos com mais de quatro a seis cicatrizes de lascas resultantes de lascas e/ou retoques, dispostas com alta frequência de continuidade e/ou intercalação, podem ser interpretadas como tendo sido formadas por percussão proposital e são identificadas como ferramentas ósseas expeditas. .
Para o nosso estudo, as medidas de abundância taxonômica são baseadas no número de espécimes identificados (NISP) e não no número mínimo de indivíduos (MNI). Por um lado, o NISP pode ser comparado quantitativamente com os dados do ZooMS, que são essencialmente uma contagem NISP. Por outro lado, o MNI da maioria dos táxons em cada camada é 1 ou 2 (Dados Suplementares 5A ), o que não conduz a estimativas de abundância taxonômica. O NISP em nosso estudo é calculado como o número de espécimes identificados para espécies (por exemplo, Aquila chrysaetos ) e gênero (por exemplo, Coelodonta sp.), e ocasionalmente para (sub)família (por exemplo, Caprinae) 50 .
Extração e digestão de colágeno ZooMS
Amostramos aproximadamente 10–30 mg de cada amostra. Dois protocolos foram aplicados às amostras ósseas BKC, bem como às amostras de referência: o método de extração não destrutivo com tampão de bicarbonato de amônio (AmBic) 51 e o método de extração insolúvel em ácido (ácido) 19 . Detalhes para ambos os protocolos foram fornecidos anteriormente 12 , 19 . O protocolo ácido foi aplicado a todas as amostras de referência e a todas as amostras BKC. Além disso, o protocolo AmBic foi aplicado a todas as amostras de referência e 192 amostras BKC. Resumidamente, antes da extração, todas as amostras ósseas foram armazenadas em 100 µl de solução AmBic durante a noite, para remover qualquer contaminação solúvel. Após a remoção desta solução, para o protocolo AmBic, 100 µl de bicarbonato de amônio (50 mM) foram adicionados às amostras ósseas e seguido de incubação por uma hora a 65 °C. Em seguida, 50 µl do sobrenadante contendo a proteína solúvel foram transferidos para uma nova placa de 96 poços ou tubo Eppendorf e digeridos com tripsina (Promega, V115A) a 37 °C durante a noite. A digestão foi terminada com 1 µl de ácido trifluoroacético (TFA) a 5%. A limpeza e purificação dos peptídeos foram feitas em placas C18 ZipTips (Thermo Fisher Scientific) ou C18 (Thermo Fisher Scientific), com eluição em 50 µl ou 100 µl, respectivamente, com solução de lavagem TFA a 0,1% e TFA a 0,1% em acetonitrila a 50%. como solução de condicionamento e eluição. Para o protocolo ácido, as amostras ósseas foram desmineralizadas em solução de ácido clorídrico (HCl) 0,6 M por um a dois dias. O HCl foi descartado e o sedimento foi lavado três vezes com AmBic 50 mM até o pH ficar em torno de 8. As etapas subsequentes foram idênticas ao protocolo AmBic. Todas as amostras de referência foram processadas em tubos Eppendorf individuais e as amostras ósseas BKC foram processadas utilizando placas de 96 poços.
Extração do proteoma ósseo Xiahe 2
Quatro digestões proteômicas foram geradas e utilizadas para análise por LC-MS/MS da amostra de costela Xiahe 2, derivadas de um total de duas amostras ósseas retiradas desta amostra. Ambas as amostras foram primeiro armazenadas durante a noite em AmBic 50 mM para remover qualquer contaminação de proteína solúvel potencialmente presente nas superfícies ósseas. Posteriormente, a primeira extração de proteína da primeira amostra envolveu uma extração AmBic ZooMS na qual a amostra foi incubada em AmBic 50 mM por uma hora a 65 °C. A digestão das proteínas solubilizadas no sobrenadante resultante foi subsequentemente realizada em solução e durante a noite, a 37 °C (tripsina, Promega, V115A). O sedimento restante, bem como a segunda amostra de osso, foram subsequentemente desmineralizados em HCl 0,6 M durante um ou dois dias, até ser observada desmineralização. Os extratos foram centrifugados e a solução ácida contendo proteínas solúveis em ácido foi removida, seca em speedvac e ressuspensa em AmBic 50 mM. Posteriormente, a digestão das proteínas foi realizada durante a noite utilizando tripsina a 37°C. Finalmente, o quarto extrato referia-se à ressuspensão do sedimento proteico desmineralizado restante da primeira amostra em AmBic 50 mM. Novamente, a digestão das proteínas foi realizada utilizando tripsina a 37 °C. Em cada caso, os péptidos foram acidificados para terminar o processo de digestão utilizando 1% de TFA, brevemente centrifugados para sedimentar quaisquer resíduos minerais ou proteicos restantes e purificados utilizando C18 ZipTips como descrito acima para ZooMS. A análise LC-MS/MS foi realizada em 10 µl de cada um dos quatro eluatos peptídicos.
MALDI-TOF MS
Para análises MALDI-TOF MS, 1 µl dos peptídeos eluídos foi misturado com 1 µl de solução de matriz (ácido α-ciano-4-hidroxicinâmico a 1% na solução de condicionamento) e colocado em uma placa alvo MALDI de 384 poços. Cada amostra foi manchada em triplicado. Todos os dados dos espectros MS foram obtidos no Fraunhofer IZI, Leipzig, utilizando o Autoflex Speed LRF MALDI-TOF (Bruker). A fusão de réplicas espectrais foi realizada em R usando os pacotes MALDIquant v.1.22.1 e MALDIquantForeign v.0.14 52 , 53 , 54 . As massas dos marcadores peptídicos foram observadas em mMass 55 e identificado em comparação com o banco de dados ZooMS atualizado (Dados Suplementares 4 ).
Análise LC-MS/MS
Dez microlitros dos peptídeos eluídos de 14 espécies (Dados Suplementares 3 ) foram processados usando análise LC-MS/MS na Universidade de Copenhague.
um NanoLC fácil da Thermo Fisher Scientific, com o gradiente especificado na Tabela Suplementar 3.1 Para cromatografia líquida, utilizou-se , a 250 nl por min. O carregamento foi feito a 500 nl por min. As fases móveis são A: 5% de acetonitrila, 0,1% de ácido fórmico; B: 95% de acetonitrila, 0,1% de ácido fórmico. O emissor consistia em um tubo capilar flexível de sílica fundida Polymicro de 75 µm de diâmetro interno e 20 cm de comprimento puxado e embalado com partículas de sílica ligadas C18 de 1,9 µm de diâmetro (ReproSil-Pur, C18-AQ, Dr. Maisch). A coluna foi montada em uma fonte de eletropulverização com um forno de coluna regulado para 40°C.
Para espectrometria de massa, a tensão da fonte foi de +2.000 V com um tubo de transferência de íons ajustado a 275 °C. Um Exploris 480 da Thermo Fisher Scientific estava operando em modo dependente de dados, consistindo em uma primeira varredura MS1 com uma resolução de 120.000 entre valores m / z de 350 e 1.400. Os dez principais precursores monoisotópicos foram selecionados se estivessem acima de uma intensidade de 2 × 10 4 with a charge state between 2 and 6, and were then dynamically excluded after one appearance with their isotopes (±20 ppm) for 20 s. The selected peptides were acquired for MS2 at an Orbitrap resolving power of 60,000, with a normalized collision energy (HCD) set at 30%, a quadrupole isolation width of 1.2 m/z and a first m/z of 100.
Validação do marcador peptídico COL1
As sequências peptídicas foram adquiridas a partir dos dados brutos de LC-MS/MS usando PEAKS v.7.0 (ref. 56 ), com espécies intimamente relacionadas com sequências de colágeno disponíveis formando bancos de dados de referência. Desamidação (NQ), hidroxilação (P) e oxidação (M) foram definidas como modificações variáveis, e nenhuma modificação fixa foi incluída. A tolerância ao erro de massa parental foi ajustada para 10 ppm, a tolerância ao íon fragmento para 0,07 Da e tripsina como protease. Os peptídeos foram filtrados para uma taxa de descoberta falsa (FDR) de 0,5%, com base em recomendações conservadoras anteriores feitas para análise PEAKS de paleoproteomas esqueléticos 57 , e a reconstrução da sequência foi realizada em R 52 usando os pacotes Tidyverse v.2.0.0 (ref. 58 ), Zelador v.2.2.0 (ref. 59 ), Biostrings v.2.68.1 (ref. 60 ) e msa 61 . Polimorfismos entre as referências e a sequência reconstruída dentro do marcador peptídico foram validados manualmente no PEAKS.
Análise LC-MS/MS de Xiahe 2
As sequências peptídicas foram identificadas a partir de dados de LC-MS/MS usando PEAKS v.7.0 (ref. 56 ) com um banco de dados que consiste no proteoma de referência humano (UP000005640 com uma sequência de proteína por gene, baixado em 22/02/2022) com variação arcaica adicional de Neandertais 62 e um denisovano 63 . A tolerância ao erro de massa parental foi definida como 10,0 ppm e a tolerância ao erro de massa do fragmento como 0,07 Da. Desamidação (NQ), hidroxilação (P), oxidação (M) e piro-Glu (E e Q) foram definidas como modificações variáveis. Os péptidos foram filtrados para 0,5% de FDR e exportados para processamento adicional.
Construção das sequências da proteína Xiahe 2 e análise filogenética
Para dados proteômicos do indivíduo Xiahe 2, as sequências proteicas foram reconstruídas para todas as proteínas com cinco ou mais peptídeos (Tabela Suplementar 4.1 e Dados Suplementares 6 ) em R 52 usando os pacotes Janitor v.2.2.0 (ref. 59 ), Biostrings v.2.68.1 (ref. 60 ) e Tidyverse v.2.0.0 (ref. 58 ). O arquivo protein.csv exportado do PEAKS foi usado para obter uma visão geral das proteínas presentes no conjunto de dados e sua abundância, enquanto o arquivo protein-peptídeos.csv foi usado para reconstrução de sequência. Para cada posição de aminoácido, foi obtido um consenso majoritário com base nas contagens de peptídeos. As sequências reconstruídas foram posteriormente alinhadas usando Geneious Prime v.2023.2.1 com uma lista de proteomas de referência, e todos os polimorfismos foram validados manualmente em PEAKS. Os proteomas de referência utilizados foram o proteoma de referência humano UP000005640 (baixado do Uniprot 17-01-2022), traduções de três genomas de Neandertal 62 and a translation of a Denisovan genome63.
Uma filogenia foi construída usando os proteomas de referência descritos acima, bem como proteínas correspondentes de Gorilla gorilla , Pongo abelii e Pan troglodytes . As sequências foram alinhadas no Geneious Prime para identificar e corrigir variações de isoformas entre as referências. Em resumo, as posições 1264–1270 (baseadas em P29400) do COL4A5 foram removidas para os Neandertais e P. troglodytes ; as posições 261–313 (com base em P12107) do COL11A1 foram removidas para todos os indivíduos; Foram adicionadas 235 incógnitas após a posição 219 (com base em P39060) de COL18A1 para G. gorilla , devido à falta de porções da sequência de referência; e as posições 3–21 de COL27A1 (baseado em Q8IZC6) para G. gorilla foram substituídas por incógnitas devido a grandes diferenças de sequência. As sequências de proteínas foram concatenadas pelo indivíduo, com um comprimento de proteína concatenada de 31.781 posições de aminoácidos.
As análises filogenéticas foram realizadas utilizando o modelo de substituição de Dayhoff e particionamento por proteína. A sequência de P. abelii foi usada como grupo externo. Uma árvore Bayesiana foi gerada usando MrBayes v.3.2.7 (ref. 64 ), com configurações seguindo um relatório anterior 3 , exceto que a análise foi executada apenas para 100.000 gerações, porque o desvio padrão das frequências divididas já era zero neste ponto. RAxML v.4.0 (ref. 65 ) a análise foi executada por meio do Geneious Prime, com 1.000 bootstraps (bootstrapping rápido com busca pela árvore de ML com melhor pontuação). A filogenia foi plotada em R usando Ape v.5.7.1 (ref. 66 ).
Análise da comunidade faunística
Para melhor comparar os conjuntos de dados obtidos por observações morfológicas e pelo ZooMS, unimos todos os resultados nos níveis de grupo taxonômico do ZooMS, porque estes são geralmente menos específicos do que as designações de espécies obtidas através da análise morfológica. Por exemplo, atribuímos os táxons Bos cf. mutus e Bos grunniens dos grupos taxonômicos morfológicos para Bos sp., porque essas espécies não podem ser separadas usando ZooMS. Porém, para melhor visualização dos dados morfológicos, mantivemos as informações das espécies de alguns exemplares utilizando os nomes das espécies, como Bos grunniens ou Bos cf. mutus , em alguns locais quando necessário. Os nomes dos táxons usados no texto estão de acordo com os conjuntos de dados específicos (morfologia, ZooMS e ambos combinados) e as informações do conjunto de dados de origem são indicadas sempre que necessário. O conjunto de dados combinado, um híbrido de identificações morfológicas e taxonômicas do ZooMS, foi utilizado para analisar a composição da comunidade faunística no BKC e calcular índices de diversidade. Dentro deste conjunto de dados, excluímos grupos como pássaros, roedores e atribuições taxonômicas para níveis interordenados ou subordenados.
Os índices de Shannon-Weaver e Simpson, incluindo os intervalos de confiança (97,5–2,5%), foram calculados de acordo com método descrito anteriormente 67 avaliar a ecologia comunitária de cada unidade estratigráfica. Estes foram calculados em R 52 usando o pacote vegan v.2.6-4 68 . Os valores dos índices de Shannon-Weaver e Simpson são positivamente proporcionais à diversidade, sugerindo que valores de índice mais elevados indicam maior diversidade.
Desamidação
A desamidação da glutamina (Gln; Q) em ácido glutâmico (Glu; E) em amostras ósseas é uma das modificações pós-traducionais comuns em contextos arqueológicos. De acordo com estudos anteriores 69 , 70 , foi proposto um método eficaz para calcular a desamidação da glutamina para peptídeos de colágeno tipo I selecionados. Aqui calculamos a desamidação da glutamina para dois peptídeos, COL1α1 508–519 (GVQGPPGPAGPR; P1105) e COL1α1 435–453 (DGEAGAQGPPGPAGPAGER; P1706), que possuem taxas de desamidação relativamente lentas 69 . Eles também estão comumente presentes nos espectros BKC MALDI-TOF MS, e suas sequências peptídicas são idênticas para a maioria dos mamíferos terrestres 19 . A desamidação é expressa numa escala de 0 a 1, com 1 indicando nenhuma desamidação e 0 indicando desamidação completa.
Resumo do relatório
Mais informações sobre o desenho da pesquisa estão disponíveis no Nature Portfolio Reporting Summary vinculado a este artigo.
Disponibilidade de código
All R code used for protein sequence reconstruction is available through Zenodo at https://doi.org/10.5281/zenodo.11281297 (ref. 72).
References
Reich, D. et al. Genetic history of an archaic hominin group from Denisova Cave in Siberia. Nature 468, 1053–1060 (2010).
Demeter, F. et al. A Middle Pleistocene Denisovan molar from the Annamite Chain of northern Laos. Nat. Commun. 13, 2557 (2022).
Chen, F. et al. Uma mandíbula denisovana do final do Pleistoceno Médio do Planalto Tibetano. Natureza 569 , 409–412 (2019).
Zhang, D. et al. DNA denisovano em sedimentos do Pleistoceno Superior da caverna Baishiya Karst no planalto tibetano. Ciência 370 , 584–587 (2020).
Prüfer, K. et al. A sequência completa do genoma de um Neandertal das montanhas Altai. Natureza 505 , 43–49 (2014).
Larena, M. et al. Ayta filipina possui o mais alto nível de ascendência denisovana do mundo. Curr. Biol. 31 , 4219–4230 (2021).
Cheng, T. et al. Ocupação hominínea do planalto tibetano durante o Último Complexo Interglacial. Quat. Ciência. Rev. 265 , 107047 (2021).
Wang, J. et al. Estratégias de subsistência de caçadores-coletores pré-históricos no planalto tibetano durante a última deglaciação. Ciência. Ciência da Terra da China. 63 , 395–404 (2020).
Wang, Y. et al. Estratigrafia integrativa quaternária, biotas e evolução paleogeográfica do planalto Qinghai-Tibetano e áreas adjacentes. Ciência. Ciência da Terra da China. 67 , 1360–1394 (2024).
Morin, E., Ready, E., Boileau, A., Beauval, C. & Coumont, M.-P. Problemas de identificação e quantificação em análise arqueozoológica, parte I: insights de um teste cego. J. Arqueol. Teoria do Método 24 , 886–937 (2017).
Morin, E., Ready, E., Boileau, A., Beauval, C. & Coumont, M.-P. Problemas de identificação e quantificação em análises arqueozoológicas, parte II: apresentação de um método alternativo de contagem. J. Arqueol. Teoria do Método 24 , 938–973 (2017).
Sinet-Mathiot, V. et al. Combinando ZooMS e zooarqueologia para estudar o comportamento dos hominídeos do Pleistoceno Superior em Fumane (Itália). Ciência. Rep. 9 , 12350 (2019).
Ruebens, K. et al. A ocupação do Paleolítico Médio Superior de Abri du Maras (Camada 1, Neroniano, Sudeste da França): integrando análises líticas, ZooMS e datação por radiocarbono para reconstruir o comportamento de caça dos Neandertais. J. Paleolit. Arqueol. 5 , 4 (2022).
Sinet-Mathiot, V. et al. A identificação da fauna não identificada melhora a compreensão das estratégias de subsistência dos hominídeos durante a transição do Paleolítico Médio para o Superior. Arqueol. Antropol. Ciência. 15 , 139 (2023).
Smith, GM et al. de aproximadamente 45.000 anos de idade A ecologia, subsistência e dieta do Homo sapiens em Ilsenhöhle em Ranis, Alemanha. Nat. Eco. Evol. 8 , 564–577 (2024).
Martisius, NL et al. A identificação não destrutiva do ZooMS revela a seleção estratégica de matéria-prima para ferramentas ósseas pelos Neandertais. Ciência. Rep. 10 , 7746 (2020).
Almécija, S. et al. Macacos fósseis e evolução humana. Ciência 372 , eabb4363 (2021).
Brown, S. et al. Examinando a preservação do colágeno através da desamidação da glutamina na Caverna Denisova. J. Arqueol. Ciência. 133 , 105454 (2021).
Welker, F. et al. Variações na desamidação da glutamina para uma montagem óssea Châtelperroniana, medida pela impressão digital da massa peptídica do colágeno. Ciência. Tecnologia. Arqueol. Res. 3 , 15–27 (2016).
Wang, N. et al. Aplicação em larga escala de paleoproteômica (Zooarqueologia por espectrometria de massa; ZooMS) em duas assembleias de fauna paleolítica da China. Processo. R.Soc. B 290 , 20231129 (2023).
Xiao, J. et al. Tamanho do grão de quartzo como indicador da força das monções de inverno no planalto de Loess, na China central, durante os últimos 130.000 anos. Quat. Res. 43 , 22–29 (1995).
Ding, ZL et al. Expansão gradual do ambiente desértico em todo o norte da China nos últimos 3,5 milhões de anos e implicações para a evolução das monções. Planeta Terra. Ciência. Vamos. 237 , 45–55 (2005).
Owen, LA e Dortch, JM Natureza e tempo da glaciação quaternária no orógeno Himalaia-Tibetano. Quat. Ciência. Rev. 88 , 14–54 (2014).
Huerta-Sánchez, E. et al. Adaptação à altitude em tibetanos causada pela introgressão de DNA semelhante ao Denisovan. Natureza 512 , 194–197 (2014).
Jacobs, GS et al. Múltiplas ascendências denisovanas profundamente divergentes em papuas. Célula 177 , 1010–1021 (2019).
Zhang, X. et al. A história e evolução do haplótipo Denisovan- EPAS1 em tibetanos. Processo. Acad. Nacional. Ciência. EUA 118 , e2020803118 (2021).
Lisiecki, LE & Raymo, ME Uma pilha Plioceno-Pleistoceno de 57 δ bentônicos distribuídos globalmente 18 Ó registros. Paleoceanografia 20 , PA1003 (2005).
Beck, JW et al. Um recorde de 550.000 anos de chuvas de monções no Leste Asiático de 10 Estar em loess. Ciência 360 , 877–881 (2018).
Sun, Y., Clemens, SC, An, Z. & Yu, Z. Escala de tempo astronômica e implicação paleoclimática de registros empilhados de monções de 3,6 Myr do planalto chinês de Loess. Quat. Ciência. Rev. 25 , 33–48 (2006).
Zhao, Y. et al. Evolução da vegetação e da variabilidade climática no planalto tibetano ao longo dos últimos 1,74 milhões de anos. Ciência. Av. 6 , eaay6193 (2020).
Schmid, E. Atlas de ossos de animais: para pré-historiadores, arqueólogos e geólogos quaternários (Elsevier, 1972).
O Grupo de Compilação de Fósseis de Vertebrados Chineses do Instituto de Paleontologia e Paleoantropologia de Vertebrados da Academia Chinesa de Ciências. Manual de Fósseis de Vertebrados Chineses (Science Press, 1979).
Pales, L. & Lamert, P. Atlas Osteológico: Para Servir na Identificação de Mamíferos Quaternários (Centro Nacional de Pesquisa Científica, 1981).
Chen, DX Atlas Anatômico Comparativo de Tigre, Leopardo e Ossos de Animais Semelhantes (China Medical Science Press, 1994).
França, DL Identificação de Ossos Humanos e Não Humanos: um Atlas Colorido (CRC Press, 2009).
Hillson, S. Ossos e dentes de mamíferos: um guia introdutório aos métodos de identificação (University College London, 1992).
Hillson, S. Teeth 2ª ed. (Cambridge Univ. Press, 2012).
Cérebro, CK Os caçadores ou os caçados? (Univ. Chicago Press, 1981)
Behrensmeyer, AK Informações tafonômicas e ecológicas do intemperismo ósseo. Paleobiologia 4 , 150–162 (1978).
Binford, LR Bones: Homens Antigos e Mitos Modernos (Academic Press, 1981).
Lyman, LR Vertebrate Tafonomia (Cambridge Univ. Press, 1994).
Fisher, WJ Modificações da superfície óssea em zooarqueologia. J. Arqueol. Teoria do Método 2 , 7–68 (1995).
Fernández-Jalvo, Y. & Andrews, P. Atlas de identificações tafonômicas: mais de 1001 imagens de fósseis e modificações recentes de ossos de mamíferos (Springer, 2016).
Stiner, MC et al. Queima diferencial, recristalização e fragmentação de osso arqueológico. J. Arqueol. Ciência. 22 , 223–237 (1995).
Villa, P. & Mahieu, E. Padrões de ruptura de ossos longos humanos. J. Hum. Evol. 21 , 27–48 (1991).
Stiner, MC As Faunas da Caverna Hayonim, Israel: Um Registro de 200.000 anos de Dieta Paleolítica, Demografia e Sociedade (Peabody Museum Press, 2005).
Mallye, J.-B. e outros. Os retocadores ósseos Mousterianos da Caverna Noisetier: experimentação e identificação de marcas. J. Arqueol. Ciência. 39 , 1131–1142 (2012).
Backwell, LR & d'Errico, F. O primeiro uso de ferramentas de osso: uma reavaliação das evidências do desfiladeiro de Olduvai, Tanzânia. Paleontol. Africana 40 , 95–158 (2004).
Doyon, L. et al. Uma tecnologia óssea expedita de 115.000 anos em Lingjing, Henan, China. PLoS One 16 , e0250156 (2021).
Grayson, DK Zooarqueologia Quantitativa: Tópicos na Análise de Faunas Arqueológicas (Academic Press, 1984).
van Doorn, NL, Hollund, H. & Collins, MJ Uma abordagem nova e não destrutiva para análise ZooMS: extração com tampão de bicarbonato de amônio. Arqueol. Antropol. Ciência. 3 , 281–289 (2011).
Equipe principal R. R: Uma linguagem e ambiente para computação estatística https://www.R-project.org/ (R Foundation for Statistical Computing, 2022).
Gibb, S. & Strimmer, K. MALDIquant: a versatile R package for the analysis of mass spectrometry data. Bioinformatics 28, 2270–2271 (2012).
Gibb, S. MALDIquantForeign: rotinas de importação/exportação para 'MALDIquant'. R versão 3.3.3 https://CRAN.R-project.org/package=MALDIquantForeign (2022).
Strohalm, M., Hassman, M., Košata, B. & Kodíček, M. mMass data miner: uma alternativa de código aberto para análise de dados espectrométricos de massa. Comunhão rápida. Espectro de Massa. 22 , 905–908 (2008).
Zhang, J. et al. PEAKS DB: pesquisa de banco de dados assistida por sequenciamento de novo para identificação de peptídeos sensível e precisa. Mol. Célula. Proteômica 11 , M111.010587 (2012).
Welker, F. Elucidação dos efeitos proteômicos entre espécies na identificação do proteoma ósseo humano e hominídeo por meio de um experimento de bioinformática. BMC Evol. Biol. 18 , 23 (2018).
Wickham, H. et al. Bem-vindo ao Tidyverse. J. Software de código aberto. 4 , 1686 (2019).
Firke, S. Janitor: ferramentas simples para examinar e limpar dados sujos. R versão 3.1.2 https://CRAN.R-project.org/package=janitor (2023).
Pagès, H., Aboyoun, P., Gentleman, R. & DebRoy, S. Biostrings: efficient manipulation of biological strings. https://bioconductor.org/packages/Biostrings (2022).
Bodenhofer, U., Bonatesta, E., Horejs-Kainrath, C. & Hochreiter, S. msa: an R package for multiple sequence alignment. Bioinformatics 31, 3997–3999 (2015).
Castellano, S. et al. Patterns of coding variation in the complete exomes of three Neandertals. Proc. Natl Acad. Sci. USA 111, 6666–6671 (2014).
Meyer, M. et al. A high-coverage genome sequence from an archaic Denisovan individual. Science 338, 222–226 (2012).
Ronquist, F. et al. MrBayes 3.2: inferência filogenética bayesiana eficiente e escolha de modelo em um grande espaço de modelo. Sist. Biol. 61 , 539–542 (2012).
Stamatakis, A. RAxML-VI-HPC: análises filogenéticas baseadas em máxima verossimilhança com milhares de táxons e modelos mistos. Bioinformática 22 , 2688–2690 (2006).
Paradis, E. & Schliep, K. Ape 5.0: um ambiente para filogenética moderna e análises evolutivas em R. Bioinformatics 35 , 526–528 (2019).
Gardener, M. Ecologia Comunitária: Métodos Analíticos usando R e Excel (Pelagic Publishing, 2014).
Oksanen, J. et al. vegano: pacote ecológico comunitário. R versão 2.6-4 https://CRAN.R-project.org/package=vegan (2022).
van Doorn, NL et al. Desamidação específica do local da glutamina: um novo marcador de deterioração do colágeno ósseo. Comunhão rápida. Espectro de Massa. 26 , 2319–2327 (2012).
Wilson, J., van Doorn, NL e Collins, MJ Avaliando a extensão da degradação óssea usando desamidação de glutamina no colágeno. Anal. Química. 84 , 9041–9048 (2012).
Perez-Riverol, Y. et al. Os recursos do banco de dados PRIDE em 2022: um centro para evidências proteômicas baseadas em espectrometria de massa. Ácidos Nucleicos Res. 50 , D543–D552 (2022).
Fagernäs, Z. Reconstrução da sequência da caverna Baishiya Karst. Zenodo 10.5281/zenodo.11281297 (2024).
Agradecimentos
Agradecemos à Administração Nacional do Patrimônio Cultural da China, à Administração Provincial do Patrimônio Cultural de Gansu, ao Instituto Provincial de Relíquias Culturais e Pesquisa Arqueológica de Gansu, ao Departamento de Cultura, Rádio, Televisão e Turismo da Prefeitura Autônoma Tibetana de Gannan, ao Condado de Xiahe e ao Templo Baishiya pelo apoio nossas escavações arqueológicas em BKC. Agradecemos a T. Zhang do Instituto Noroeste de Biologia do Planalto, CAS, e L. Zhang da Universidade de Lanzhou, Centro Nacional de Recursos de Coleção de Animais, Instituto de Zoologia, CAS, Museu da Universidade de Lanzhou por fornecer amostras modernas de ossos de animais para análise proteômica. Este trabalho foi apoiado pela Fundação Nacional de Ciências Naturais da China (41988101, 42325103, 42130502, 42001086 e 42301171) e pela Fundação de Pós-Doutorado em Ciências da China (2020M683605). FW recebeu financiamento do Conselho Europeu de Pesquisa no âmbito do programa de pesquisa e inovação Horizonte 2020 da União Europeia (948365). O GMS é financiado pelo programa de investigação e inovação Horizonte 2020 da União Europeia no âmbito do esquema Marie Sklodowska-Curie (101027850). O trabalho no Centro de Pesquisa de Proteínas da Fundação Novo Nordisk é financiado em parte por uma doação da Fundação Novo Nordisk (NNF14CC0001).
Informações do autor
Autores e Afiliações
Contribuições
FC, DZ e FW conceberam o estudo. DZ, FC, JW, TL, XS, JY, TC, ZP, XC, YH, YL, DL e HX realizaram investigações de campo e escavações. HX realizou amostragem proteômica. FW, HX, ZF e GT realizaram análises proteômicas. JW, TL e GMS realizaram a análise zooarqueológica. FC e ZS analisaram dados ambientais. J.-JH e JVO facilitaram o estudo. DZ, FW, HX, JW, ZF e FC escreveram o artigo com contribuições de todos os autores.
Autores correspondentes
Declarações éticas
Interesses conflitantes
Os autores declaram não haver interesses conflitantes.
Revisão por pares
Informações de revisão por pares
A Nature agradece a Timothy Cleland e ao(s) outro(s) revisor(es) anônimo(s) por sua contribuição para a revisão por pares deste trabalho.
Informações adicionais
Nota do editor A Springer Nature permanece neutra em relação a reivindicações jurisdicionais em mapas publicados e afiliações institucionais.
Figuras e tabelas de dados estendidas
Dados estendidos Fig. 1 Locais do final do Pleistoceno Médio e Superior no Leste Asiático.
Os números representam sítios arqueológicos e/ou fósseis de hominídeos como segue: 1. Crânio de Harbin; 2. Jinniushan; 3. Xujiayao; 4. Caverna Tianyuan; 5. Localidade 1 e 2 de Shuidonggou; 6.Jianjunfu 01; 7. Yangshang; 8. Dalí; 9. Lingjing; 10. Caverna Huanglong; 11. Nwya Devu; 12. Quesang; 13. Piluo; 14. Caverna Xinglong; 15.Hualongdong; 16. Tongzi; 17. Guanyindong; 18. Caverna Fuyan; 19. Mabá; 20. Penghu 1; 21. Lunadong; 22. Zhirendong (Caverna Zhiren). Informações detalhadas são fornecidas em Dados Suplementares 1 . Mapas base gerados pelo ArcGIS 10.7 usando dados raster de https://www.naturalearthdata.com/ .
Dados estendidos Fig. 2 Fotografias de espécimes faunísticos recuperados do BKC durante as escavações de 2018 e 2019.
a , Coelodonta sp. inferior esquerdo M1 (camada 10b). b , Equus sp. P2 superior esquerdo (camada 10b). c , Bos cf. mutus superior esquerdo M3 (camada 10). d , Ovis ammon deixou o úmero (camada 10). e , Procapra cf. picticaudata metacarpo esquerdo (camada 2). f , Pseudois nayaur deixou a tíbia (camada 11). g , Moschu sp. canino superior direito (camada 5). h , Cervus elaphus deixou o metatarso (camada 10). eu , Panthera cf. uncia vértebra lombar (camada 10d). j , Canis lupus úmero esquerdo (camada 11). k , Crocuta crocuta ultima ulna esquerda (camada 10a). l , crânio Vulpes ferrilata (camada 2). m , Martes cf. foina mandíbula esquerda (camada 11). n , Mustela sp. mandíbula esquerda (camada 7). o , Aeretes melanopterus mandíbula esquerda (camada 6). p , Hystrix cf. mandíbula esquerda subcristata (camada 10a). q , Marmota Himalayana mandíbula esquerda (camada 10). r , Lepus oiostolus calcâneo esquerdo (camada 10c1). s , Myospalax cf. crânio cansus (camada 4). t , Aquila chrysaetos deixou o tarsometatarso (camada 11). você , Fasiano cf. colchicus tibiotarso esquerdo (camada 10d). As identificações são provenientes de análise taxonômica morfológica.
Dados estendidos Fig. 3 Composição relativa de espécies de BKC, para táxons selecionados.
Proporções de táxons selecionados identificados usando morfologia, ZooMS e ambos combinados, por camada. O NISP total para cada camada é fornecido na parte superior. Os dados combinados são baseados na Tabela de Dados Estendidos 1 ( n = 1.558).
Dados estendidos Fig. 4 Distribuição de diferentes modificações na superfície óssea entre as amostras ósseas.
a , Porcentagens de amostras ósseas com diferentes modificações na superfície óssea entre diferentes tipos de osso (indet. representa amostras indeterminadas). b , Porcentagens de espécimes ósseos com modificações antropogênicas identificadas por morfologia (Dados Suplementares 5A ), ZooMS (Dados Suplementares 5B ) e ambos combinados (Dados Suplementares 5C ). Em a , b , o número exibido em cada barra é o NISP total correspondente usado no cálculo.
Dados Estendidos Fig. 5 Exemplos de modificações antrópicas em espécimes faunísticos de BKC.
a , b , Caprinae (camada 2) ( a ) e mamífero classe II (cf. Caprinae, camada 10c) ( b ) espinhas neurais vertebrais, com marcas de corte geradas durante a descarnação. c , d , colo femoral Pseudois nayaur (camada 3) ( c ) e maléolo medial da tíbia (camada 11) ( d ), com marcas de corte criadas durante a desarticulação. e , f , Procapra cf. picticaudata (camada 10b) ( e ) e Caprinae (camada 11) ( f ) falange 1, com marca(s) de corte oblíqua gerada(s) durante atividades de esfola. g , h , diáfise do úmero de Caprinae (camada 3) ( g ) e diáfise do fêmur (camada 11; ID do táxon ZooMS) ( h ), com duas marcas de corte paralelas relacionadas a atividades de açougue. eu , Bos sp. diáfise do úmero (camada 9), com marcas de corte associadas às atividades de descarnação. Descamação concoidal da borda medular (triângulo preto) e danos na superfície da percussão cortical (seta branca) indicam consumo de medula óssea. j – l , diáfise do rádio de Caprinae (camada 7) ( j ) com escama óssea aderente (triângulo preto) e Bos sp . (ID do táxon ZoomMS) flocos ósseos (camada 10a ( k ) e camada 11 ( l )), são indicações de atividades de extração de medula óssea. m , diáfise de úmero/fêmur de mamífero Classe III/IV (camada 8), com evidência de queimadura após o fragmento da diáfise ter sido recentemente quebrado e mostrando uma sequência de gradientes de cor da cavidade medular até o córtex (seta branca). Exceto onde indicado, as identificações taxonômicas são provenientes de análise morfológica.
Dados estendidos Fig. 6 O Homo sp. espécime de costela (Xiahe 2) identificado pelo ZooMS.
a , espectro MALDI-TOF MS de Xiahe 2. b , Comparação dos valores de desamidação de Xiahe 2 com valores de espécimes faunísticos da camada 3 (n = 139, incluindo Xiahe 2, indicado pelo ponto vermelho), os espécimes ósseos datados por radiocarbono (~50–30 ka, n = 10, Dados Suplementares 2 ) e amostras de referência modernas (n = 20). Aqui, o valor de 1 indica nenhuma desamidação, enquanto o valor de 0 indica desamidação completa da glutamina única no peptídeo marcador COL1α1 508–519. Os gráficos de caixa contêm o intervalo dos dados com bigodes que se estendem até 1,5 vezes o intervalo interquartil. As caixas indicam os quartis superior e inferior, enquanto a linha central indica a mediana. Todos os pontos de dados individuais são representados por gráficos de pontos sobrepostos e quaisquer pontos de dados fora do intervalo do gráfico de caixa podem ser considerados discrepantes.
Dados estendidos Fig. 7 Comparação de desamidação entre métodos de extração proteômica.
a , b , gráficos de dispersão de COL1α1 508–519 (P1105; a ) e COL1α1 435–453 (P1706; b ) valores de desamidação para extrações à base de AmBic e à base de ácido. A linha azul e a área cinza indicam a média da suavidade de loess e seu intervalo de confiança de 95% associado. A linha vermelha indica uma linha 1:1, onde os valores de desamidação obtidos através de ambos os métodos seriam os mesmos. Em ambos os casos, observa-se uma alta correlação entre os valores observados para os dois métodos de extração (um R 2 de 0,93 e 0,9, respectivamente).
Dados Estendidos Fig. 8 Comparação da desamidação e idade cronológica dos níveis estratigráficos correspondentes.
A distribuição dos valores de desamidação COL1α1 508–519 (P1105 faixa de probabilidade de 68,2% de cada camada, indicada pela linha vermelha) em relação à idade cronológica (camadas 2–10; linha preta: faixas etárias de espécimes com datas diretas de radiocarbono; azul linha: faixas etárias de cada camada, ver Tabela Suplementar 2.2 ). Uma correlação linear (linha laranja) é estimada ao longo da estratigrafia (a sombra cinza indica o intervalo de confiança de 95%). A camada 11 não está incluída aqui porque nenhuma faixa etária direta está disponível atualmente. Além disso, a camada 10 é tratada como uma unidade estratigráfica única e homogênea (mas consulte as seções 2 e 7 de Informações Suplementares ).


Nenhum comentário:
Postar um comentário
Observação: somente um membro deste blog pode postar um comentário.