DNA de sedimentos do Pleistoceno revela alterações de hominíneos e faunísticos na Caverna Denisova
Natureza volume 595 , Páginas 399–403 ( 2021 )
Abstrato
A caverna de Denisova, no sul da Sibéria, é a localidade-tipo dos denisovanos, um grupo de hominídeos arcaico relacionado aos neandertais 1 , 2 , 3 , 4 . A dúzia de restos de hominíneos recuperados dos depósitos também incluem os neandertais 5 , 6 e o filho de um neandertal e um denisovano 7 , o que sugere que a caverna de Denisova era uma zona de contato entre esses hominíneos arcaicos. No entanto, as incertezas persistem sobre a ordem em que esses grupos apareceram no local, o momento e o contexto ambiental da ocupação dos hominídeos, e a associação de grupos específicos de hominídeos com as assembleias arqueológicas 5 , 8 , 9, 10 , 11. Aqui, relatamos a análise de DNA de 728 amostras de sedimentos que foram coletadas em forma de grade de camadas que datam da época do Pleistoceno. Nós recuperamos o DNA mitocondrial (mt) da fauna e dos hominíneos antigos de 685 e 175 amostras, respectivamente. A evidência mais antiga de mtDNA hominíneo é de Denisovans e está associada às primeiras ferramentas de pedra do Paleolítico Médio que foram depositadas aproximadamente 250.000 a 170.000 anos atrás; O mtDNA de Neandertal aparece pela primeira vez no final desse período. Detectamos um turnover no mtDNA de Denisovans que coincide com as mudanças na composição do mtDNA faunístico, e evidências de que Denisovans e Neanderthals ocuparam o local repetidamente - possivelmente até, ou depois, o início do Paleolítico Superior Inicial, pelo menos 45.000 anos atrás, quando o mtDNA humano moderno é registrado pela primeira vez nos sedimentos.
Principal
A caverna Denisova consiste em três câmaras (designadas Câmaras Principal, Leste e Sul) que contêm depósitos com sequências estratigráficas que se estendem do Pleistoceno Médio ao Holoceno. Os depósitos do Pleistoceno têm cronologias que foram construídas a partir da datação por radiocarbono de osso, dente e carvão 5 (a cerca de 50 mil anos atrás (ka)) e datação óptica de sedimentos 8 (a mais de 300 ka). As idades ópticas para as Câmaras Principal e Leste (Fig. 1a-c ) podem ser alinhadas em uma escala de tempo comum (Dados Estendidos Fig. 1), mas as escavações estão em andamento na Câmara Sul, onde as camadas são apenas provisoriamente reconhecidas. O DNA mitocondrial e o DNA nuclear foram recuperados de oito fósseis de hominíneos, permitindo que quatro fossem atribuídos a Denisovans (Denisova 2, Denisova 3, Denisova 4 e Denisova 8) 1 , 2 , 3 , 4 , três a Neandertais (Denisova 5, Denisova 9 e Denisova 15) 5 , 6 , 12 e um para o filho de um Neandertal e de um Denisovano (Denisova 11) 7. No entanto, há muito poucos fósseis para permitir a reconstrução detalhada do tempo e sequência da ocupação hominínea, e a associação das assembléias do Paleolítico Médio, Paleolítico Médio médio e Paleolítico Superior Inicial identificadas no local com grupos específicos de hominíneos. Além disso, dois fósseis denisovanos (Denisova 3 e Denisova 4) - mas nenhum vestígio humano moderno - foram recuperados das camadas iniciais do Paleolítico Superior, por isso é debatido se os homininos arcaicos ou os humanos modernos criaram os ornamentos associados e ferramentas de osso 9 , 10 , 11 .
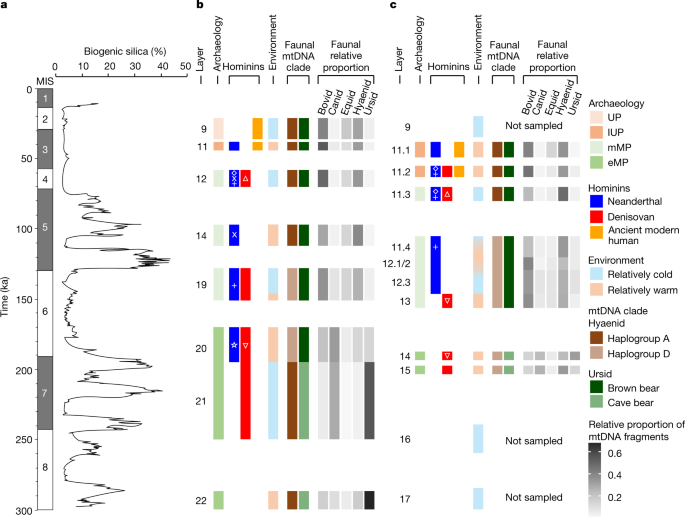
a , Câmara Leste, perfil sudeste. b , Câmara Leste, perfil noroeste. c , Câmara Principal, perfil sudeste. d , Árvore filogenética de genomas de mtDNA usada como referência para distinguir linhagens específicas de hominínicos e a colocação estimada da linhagem de mtDNA de Neandertal identificada na amostra M76 da camada da Câmara Principal 20. Círculos preenchidos em a - c indicam as localizações de amostras de sedimentos individuais e cores correspondem ao mtDNA do hominídeo detectado: vermelho (Denisovan), azul (Neandertal), amarelo (humano moderno antigo), cinza (hominídeo antigo não identificado) e branco (nenhum hominídeo antigo detectado). Outros símbolos denotam amostras para as quais o mtDNA pode ser atribuído a uma das linhagens específicas de hominina emd (Denisovan, triângulos abertos vermelhos; Neandertal, diamantes abertos azuis, cruzes e estrela). Seções estratigráficas compostas (modificadas de uma publicação anterior 8 ) à esquerda de cada perfil mostram idades inicial e final modeladas (em ka) para camadas sedimentares (incertezas a 95,4% de probabilidade). Os números dos espécimes hominíneos são mostrados circulados na camada da qual cada fóssil foi recuperado: Denisovan (vermelho), Neandertal (azul) e descendência Neandertal-Denisovan (ambas as cores) 5 . As linhas tracejadas em perfis em um e b indicam as áreas em que a atribuição é incerto camada 8. O sombreado de fundo denota a assembléia arqueológica associada: Paleolítico médio inicial (eMP) (verde escuro), Paleolítico médio médio (mMP) (verde claro), Paleolítico superior inicial (IUP) (laranja escuro) e Paleolítico superior (UP) (laranja claro) .
Um estudo piloto de preservação de DNA em sedimentos da caverna de Denisova identificou o mtDNA de hominídeo antigo em 12 das 52 amostras 13 , o que sugeriu um caminho para reconstruir a história ocupacional do local em resolução mais alta do que é possível a partir do escasso registro fóssil de hominídeo. Aqui relatamos a análise de 728 amostras de sedimento, coletadas em um padrão de grade de 10-15 cm dos depósitos expostos do Pleistoceno em todas as três câmaras (Dados estendidos Figs. 2 , 3a, b , seções de informações suplementares 1 , 2 ). Usando protocolos de laboratório automatizados, o DNA foi extraído de cada amostra, convertido em bibliotecas de fita simples e enriquecido para mtDNA de mamíferos e hominíneos 13 , 14, que identificamos no nível da família biológica usando um pipeline de análise estabelecido 13 .
Padrões de preservação de DNA
Identificamos o mtDNA de mamíferos antigos em 685 amostras (94%) de todas as camadas amostradas, incluindo aquelas com mais de 290 ka (dados estendidos Figs. 4 , 5a , Dados suplementares 1 ). O DNA recuperado dos depósitos em todas as três câmaras mostra substituições induzidas por desaminação que são características do DNA antigo 15 , 16 . Essas substituições aumentam significativamente com a idade (Dados estendidos Figs. 3 d, 4a ), o que argumenta contra a extensa lixiviação pós-deposição de DNA através das camadas 13 , 17. Também observamos uma redução significativa no comprimento médio dos fragmentos de DNA e no número de fragmentos de mtDNA recuperados com o aumento da idade (dados estendidos Figs. 3 e, f, 4b, c ), embora a variabilidade tenha sido maior entre as camadas do que para a desaminação, provavelmente como resultado das diferenças locais no ambiente geoquímico. Por exemplo, o menor número de fragmentos de DNA antigo (nenhum em algumas amostras) foi recuperado da unidade amostrada mais jovem (designada pdd-9) na Câmara Sul (Dados Estendidos Fig. 3f ), que é amplamente fosfatizada 8 e tem valores de pH ligeiramente ácidos ( entre 6 e 6,5) (Seção 3 de Informações Suplementares, Dados Suplementares 2 ).
MtDNA de hominídeo antigo
Detectamos mtDNA de hominídeo antigo em 175 amostras (24%), cobrindo quase todas as camadas em todas as três câmaras (Fig. 1a-c , Dados estendidos Figs. 3 a, b, 5b ). Quatro amostras mostraram evidências da presença de predominantemente um haplótipo e produziram fragmentos de mtDNA suficientes para reconstruir sequências de consenso de mtDNA que estão mais de 80% completas (informações suplementares seções 5 , 6 ). Três das sequências (amostras E202 e E213 da Câmara Leste (camadas 11.4 e 11.4 / 12.1) e amostra M65 da Câmara Principal (camada 19)) agrupam-se com Neandertais em árvores filogenéticas construídas com mtDNAs hominíneos previamente publicados (Informações suplementares seção 7), especificamente com Denisova 5, Denisova 15, Mezmaiskaya 1 e Scladina I-4A (Fig. 1d , Dados Estendidos Fig. 6a ). A quarta sequência (amostra M71 da Câmara Principal (camada 20)) é do tipo Denisovan e cai basal para Denisova 2 e Denisova 8, embora com baixo suporte de bootstrap (Dados Estendidos Fig. 6b ). A sequência de mtDNA mais completa (mais de 99% do genoma reconstruído) para um Neandertal da Câmara Principal (M65) tem uma estimativa de idade genética de 140 ka (intervalo de densidade posterior mais alto de 95,4% de 181–98 ka) (Informações suplementares seção 7 ) , consistente com o tempo de deposição da camada 19 (151 ± 17 a 128 ± 13 ka; aqui e abaixo, as incertezas sobre as idades ópticas são dadas a 95,4% de probabilidade) 8 .
Para as 171 amostras restantes, atribuímos fragmentos de mtDNA a grupos específicos de hominínicos, contando o número de fragmentos que suportam estados específicos de linhagem em locais de diagnóstico que distinguem entre os genomas de mtDNA humano moderno, Neandertal e Denisovano. Distinguimos três linhagens de Neandertal: a linhagem Sima de los Huesos (representando os Neandertais que viveram aproximadamente 430 ka na Espanha e cujo mtDNA está mais intimamente relacionado ao dos Denisovanos) 18 , 19 ; a linhagem Hohlenstein-Stadel (HST), que cai basal para todos os outros mtDNAs de Neandertal 20; e o 'típico' mtDNA do Neandertal, conhecido de todos os outros Neandertais. A presença de mtDNA humano antigo e moderno foi avaliada restringindo a análise a fragmentos desaminados para mitigar o efeito da contaminação de DNA humano atual. Identificamos Denisovan e mtDNA Neandertal típico em 79 e 47 amostras, respectivamente (com base em 54-9.093 fragmentos de mtDNA de hominina únicos) e mtDNA humano moderno em 35 amostras (com base em 55-2.200 fragmentos desaminados) (Fig. 1a-c , Extended Dados Fig. 3a, b ). Detectamos DNA de dois grupos de hominíneos em dez amostras, seja em uma única biblioteca ou em bibliotecas que foram preparadas a partir de subamostras independentes em alguns casos (Dados estendidos Fig. 6c , Seção 4 de informações suplementares ) Além disso, identificamos uma amostra (M76 da Câmara Principal (camada 20)) contendo fragmentos de mtDNA de hominina que suportam a ramificação compartilhada por HST e o mtDNA Neandertal típico, mas nenhuma das ramificações definindo essas linhagens. Este sinal não pode ser criado pela mistura de fragmentos de mtDNA de Neandertais, Denisovanos e humanos antigos ou modernos. Com base em simulações com mtDNA Neandertal ancestralizado, o mtDNA nesta amostra é compatível com a presença de uma linhagem de mtDNA de Neandertal previamente desconhecida que divergiu do mtDNA de Neandertal típico entre 255 e 230 ka, 20 a 45 mil anos após a divisão do HST e linhagens típicas de mtDNA de Neandertal (Seção 9 de Informações Suplementares ).
O mtDNA hominíneo mais antigo recuperado - identificado como Denisovan - se origina de uma amostra na camada 21 da Câmara Principal, que começou a acumular 250 ± 44 ka. Isso fornece a evidência genética mais antiga para a ocupação hominínea na caverna Denisova; Denisova 2 foi encontrado na camada 22.1, mas provavelmente é intrusivo de uma camada sobrejacente e tem uma idade estimada de 194–123 ka 5 . Entre todas as 223 amostras das primeiras camadas do Paleolítico Médio nas Câmaras Principal e Leste, 50 continham evidências para o mtDNA de Denisovan e apenas três (todas da camada 20 na Câmara Principal) para o mtDNA de Neandertal. Dois deles (M174 e M235) contêm mtDNA Neandertal típico e são de áreas nas quais a mistura em pequena escala com sedimentos sobrejacentes pode ter ocorrido 8; o terceiro (M76) é do meio da camada e carrega a linhagem de mtDNA de Neandertal até então desconhecida. Esses resultados apontam para os Denisovanos como os primeiros e principais criadores das primeiras assembléias do Paleolítico Médio, que têm mais de 170 ± 19 ka. Consistente com esta interpretação, a detecção do mtDNA de Neandertal em uma amostra de sedimento da camada 14 do Paleolítico Médio inicial na Câmara Leste no estudo piloto 13 foi devido a uma atribuição incorreta, que foi posteriormente corrigida para a camada Paleolítica Média 11.4 nesta câmara 8 ( Seção 2 de informações suplementares ) Nossos resultados também sugerem que os neandertais ocuparam a caverna Denisova pela primeira vez no final do início do Paleolítico Médio e podem, portanto, ter contribuído para a produção dessas assembléias em seus estágios posteriores.
Quarenta das 173 amostras das camadas médias do Paleolítico Médio nas Câmaras Principal e Leste (depositadas aproximadamente 160-60 ka) produziram mtDNA de Neandertal e / ou Denisovano, com ambos presentes em seis amostras (Fig. 1a-c ). O DNA de ambos os grupos também ocorre nas camadas deformadas do Paleolítico Médio na Câmara Sul (dados estendidos Fig. 3b ). Notavelmente, os sedimentos depositados entre 120 ± 11 e 97 ± 11 ka nas câmaras principal e leste não produziram vestígios de mtDNA de Denisovan, enquanto 12 amostras continham mtDNA de Neandertal. Isso sugere que apenas os Neandertais podem ter ocupado a caverna durante esse período, e possivelmente durante a maior parte do Estágio de Isótopo Marinho (MIS) 5 (Fig. 2 ).
a , Baikal Drilling Project 1996, registro de sílica biogênica composta de produtividade de diatomáceas no Lago Baikal, um proxy para a temperatura anual regional 28 . b , c , Dados resumidos para a Câmara Principal ( b ) e a Câmara Leste ( c ). Idades iniciais e finais para camadas e dados correspondentes são baseadas na escala de tempo comum em Dados Estendidos Fig. 1 . Os intervalos de tempo em branco representam lacunas na sequência estratigráfica ou não foram amostrados. As fases arqueológicas (início do Paleolítico Médio, Médio Paleolítico Médio, Paleolítico Superior inicial e Paleolítico Superior) seguem o esquema de cores da Fig. 1 e Dados Estendidos da Fig. 1. Os dados genéticos para hominídeos são da Fig. 1 (excluindo as amostras M174 e M235 da Câmara Principal, que são consideradas fora de contexto), e para populações de hienídeos e ursídeos dominantes da Fig. 10 de Dados Estendidos . Os símbolos para linhagens específicas de hominíneos mtDNA (Fig. 1d ) são inseridos em branco. As condições ambientais são inferidas de registros de pólen e restos de esqueletos da fauna de vertebrados 8 . As proporções relativas de mtDNA de bovídeo, canídeo, equídeo, hiaenídeo e ursídeo são de Extended Data Figs. 8 , 9 .
Apenas o antigo mtDNA humano moderno foi detectado nas camadas iniciais do Paleolítico Superior e do Paleolítico Superior na Câmara Principal (camadas 11.4 e acima, depositadas de 44 ± 5 a 21 ± 8 ka), exceto para uma amostra da camada Paleolítica Superior Inicial 11.2 que rendeu mtDNA Neandertal (Fig. 1c ). A associação entre as assembléias do Paleolítico Superior Inicial e o aparecimento de humanos modernos é ainda suportada pela recuperação do mtDNA humano moderno de uma amostra da camada Paleolítica Superior Inicial 11 na Câmara Sul, que foi depositada após 47 ± 8 ka (Dados Estendidos Fig. 3a) A situação na Câmara Leste é mais complexa: Denisovan, Neandertal e mtDNAs humanos modernos antigos foram recuperados da camada Paleolítica Superior inicial 11.2, e Neandertal e mtDNA humano moderno antigo da camada Paleolítica Superior Inicial 11.1 (Fig. 1a ). Dados esses resultados e a recuperação de dois fósseis de Denisova (Denisova 3 e Denisova 4) de camadas associadas às assembléias do Paleolítico Superior inicial, não podemos descartar a possibilidade de que, além dos humanos modernos, Denisovanos e Neandertais possam ter estado presentes durante o período de Produção inicial do Paleolítico Superior 9 , 10 , 11 .
Para 34 das 37 amostras que renderam 100 ou mais fragmentos de mtDNA de hominina desaminados, identificamos semelhanças com genomas de mtDNA de Neandertal e Denisovan específicos usando uma abordagem baseada em k -meros 21 , 22 (denotados por símbolos diferentes de círculos na Fig. 1a-c , Dados Estendidos Fig. 3b , Seção 8 de Informações Suplementares ) Esta análise revelou que as primeiras camadas do Paleolítico Médio nas Câmaras Principal e Leste e as primeiras camadas do Paleolítico Médio Médio na Câmara Leste, que abrangem o período entre 250 ± 44 e 146 ± 11 ka, contêm Denisova-2- e Denisova-8-like fragmentos de mtDNA. Isso contrasta com o mtDNA Denisovan recuperado das camadas médias do Paleolítico Médio depositadas após 80 ± 9 ka, que rendeu atribuições a sequências semelhantes a Denisova-3- e Denisova-4, assim como uma amostra da Câmara Sul. Esses resultados sugerem uma transição nas sequências de mtDNA em algum momento no intervalo 146-80 ka, possivelmente refletindo diferentes populações Denisovan. Nossos resultados também se alinham bem com as idades modeladas de Denisova 2, Denisova 3 e Denisova 4, e com a idade relativa de Denisova 8 inferida da datação molecular 5. Os sedimentos depositados entre cerca de 130 e 100 ka (e possivelmente mais, dado o intervalo de tempo subsequente de 20 milênios) - durante MIS 5 - contêm mtDNA e evidências fósseis apenas de Neandertais, com sequências de mtDNA semelhantes a Denisova-11 aparecendo apenas em sedimentos depositados após 80 ka.
MtDNA da fauna antiga
Todos os grandes mamíferos presentes no registro paleontológico da caverna de Denisova (atualmente disponível para as câmaras principal e leste apenas 8 ) também foram identificados no DNA do sedimento (Dados estendidos Fig. 7a , Informações suplementares seção 10 ). Além disso, o mtDNA antigo de camelídeos foi encontrado em uma amostra da Câmara Leste (camada 12), consistente com a presença de Camelus knoblochi no Pleistoceno na região 23 . Em contraste com os grandes mamíferos, os pequenos mamíferos (como Spalacidae, Leporidae e Sciuridae) estão amplamente ausentes dos dados genéticos (Dados Estendidos Fig. 7b), o que pode ser devido à sua biomassa inferior ou sub-representação entre as sondas de captura. Os limites nítidos entre a composição do mtDNA faunístico em algumas das camadas adjacentes (por exemplo, entre as camadas 22.1 e 21/20 na Câmara Principal) (Dados Estendidos Fig. 8 ) fornecem evidências adicionais de que a lixiviação pós-deposição de DNA é limitada, se ocorre.
Apesar da natureza altamente fragmentária dos restos fósseis na Gruta 8 de Denisova e das quantidades variáveis de DNA que podem ser depositadas por diferentes espécies, as mudanças na abundância relativa do DNA dos mamíferos ao longo do tempo são amplamente consistentes com as mudanças nos registros do esqueleto de algumas famílias, como como bovídeos, hienídeos, ursídeos e canídeos (dados estendidos Fig. 7c, d ). Os dados genéticos também fornecem a oportunidade de estudar a diversidade faunística em nível de espécie ou população, nos casos em que dados de referência abrangentes estão disponíveis (como é o caso de elefantídeos, ursídeos e hiaenídeos) (Informações Suplementares, seção 11) O mtDNA do elefante foi atribuído predominantemente a mamutes lanosos em todas as camadas, enquanto a abundância relativa de espécies de ursídeos mudou de mtDNA predominantemente de urso de caverna em camadas depositadas antes de 187 ± 14 ka para mtDNA de urso marrom exclusivamente após 112 ± 12 ka (Dados estendidos Fig. 10 ) . Também detectamos a presença de três haplogrupos de mtDNA do gênero Crocuta (hienas pintadas e hienas das cavernas) previamente descritos 24 . As camadas depositadas antes de 200 ka e depois de 120-80 ka contêm principalmente mtDNAs vistos em hienas pintadas africanas e hienas das cavernas europeias (haplogrupo A), enquanto as camadas de idade intermediária contêm mtDNA predominantemente de hienas das cavernas do leste asiático (haplogrupo D) e algumas da Europa hienas (haplogrupo B) (dados estendidos Fig. 10) As montanhas de Altai podem, portanto, ter sido uma zona de contato tanto para hominídeos quanto para linhagens distintas de hiena e outras faunas, como sugerido anteriormente por estudos de restos esqueléticos de mamíferos 25 .
Pelo menos dois turnovers principais de grandes mamíferos são evidentes a partir do DNA do sedimento (Fig. 2 ). Em primeiro lugar, mudanças marcantes nas proporções relativas de fragmentos de mtDNA de bovídeos, canídeos, equídeos, hienídeos e ursídeos, uma renovação nos haplogrupos de mtDNA de hiena e uma mudança de ursos das cavernas para ursos pardos ocorreram por volta de 190 ka, contemporaneamente com a transição climática de uma região interglacial período (MIS 7) a um período glacial (MIS 6). Os primeiros vestígios do mtDNA de Neandertal também aparecem nessa época. Um segundo turnover ocorreu entre cerca de 130 e 100 (ou 80) ka, durante e após a transição climática de MIS 6 para MIS 5: as proporções de mtDNA de bovídeos, canídeos, felinos e ursídeos diminuíram, enquanto as de cervídeos e equídeos aumentaram, e ursos das cavernas e dois haplogrupos de hiena desapareceram (Figura de dados estendidos10 ). Este período é notável também pela ausência de mtDNA Denisovan nos sedimentos da caverna. Essas mudanças sugerem que a renovação nas populações de hominíneos e faunísticas pode ter sido associada e relacionada a fatores ecológicos 25 .
Discussão
A identificação de mtDNA de hominídeo arcaico em 175 amostras de sedimento excede em uma ordem de magnitude o número de fósseis de hominídeo recuperados dos depósitos na Caverna de Denisova e fornece um perfil genético da presença de hominídeo em quase todas as camadas do Pleistoceno (Fig. 2) Esses dados são complementados por sequências de mtDNA da fauna de 685 amostras, que fornecem informações sobre a diversidade de outros grandes mamíferos e mudanças em sua abundância relativa. No entanto, advertimos que a sequência inferida de hominíneos e ocupação faunística é restringida por vários fatores: a existência de duas lacunas principais no registro estratigráfico (170-156 e 97-80 ka), a média temporal inerente ao acúmulo de cada camada de sedimento, a perturbação pós-deposição de algumas camadas devido à escavação de animais ou mistura em pequena escala 8 , 26 , e a precisão das idades ópticas usadas para construir a cronologia do local.
Além de reconstruir a história ocupacional da caverna de Denisova, nossos resultados têm implicações mais amplas para a compreensão do passado humano. Primeiro, fragmentos de mtDNA de Denisovan recuperados das camadas médias do Paleolítico Médio depositados na Caverna de Denisova após 80 ka mostram consistentemente a maior similaridade com o mtDNA de Denisova 3 e Denisova 4, assim como fragmentos de mtDNA recuperados de sedimentos na Caverna de Baishiya Karst no flanco nordeste do Platô Tibetano que são amplamente contemporâneos em idade (70-45 ka) 27 . Este padrão sugere que esta linhagem foi o tipo de mtDNA mais abundante transportado por Denisovans após 80 ka. Estudos paleontológicos 25sugeriram que os mamíferos do Pleistoceno migraram do sudeste da Ásia, ao longo do sopé oriental do Himalaia, para o noroeste de Altai. Essas migrações faunísticas podem ter estimulado a dispersão dos denisovanos na região em que seus restos mortais foram descobertos pela primeira vez. Em segundo lugar, a presença de mtDNA de Neandertal antes de 170 ka restringe ainda mais o momento de um evento inicial na história do Neandertal - a substituição da linhagem de mtDNA encontrada em neandertais que viveram 430 ka na Espanha por mtDNA que introgrediu de ancestrais primitivos de humanos modernos 18 , 19 —Entre 430 e 170 ka. O perfil de alta resolução do DNA do sedimento pode, portanto, fornecer um meio eficaz de preencher lacunas em nosso conhecimento da história evolutiva humana e da paleoecologia, independentemente da descoberta de restos de esqueletos.
Métodos
Nenhum método estatístico foi usado para predeterminar o tamanho da amostra. Os experimentos não foram randomizados e os investigadores não foram cegados quanto à alocação durante os experimentos e avaliação dos resultados.
Coleta de amostras de sedimentos
Coletamos um total de 728 amostras de sedimentos em um padrão de grade de 10-15 cm dos depósitos do Pleistoceno expostos em Main ( n = 274), East ( n = 252) e South ( n = 202) Chambers (Extended Data Figs) . 2 b – d, 3a, b) Desviávamos deste padrão apenas se as rochas estivessem no caminho ou se uma amostra estivesse claramente localizada no limite entre as camadas. Luvas, máscaras faciais e redes de cabelo foram usadas para minimizar a contaminação pelo DNA moderno. No momento da coleta da amostra, uma pequena área do perfil exposto em cada local da amostra foi primeiro limpa de volta a uma profundidade de aproximadamente 1 cm usando uma lâmina de bisturi esterilizada. Cada amostra foi então coletada com uma nova lâmina de bisturi, que foi inserida no depósito, e o sedimento extraído foi cuidadosamente colocado em um saco plástico zip-lock separado para cada amostra. Este processo foi repetido em cada local até que sedimento suficiente (vários gramas) tivesse sido coletado. Cada saco zip-lock foi selado imediatamente com fita adesiva e rotulado com seu número de amostra e prefixo M, E ou S para denotar Câmara Principal, Leste ou Sul,respectivamente. As posições das amostras e as camadas correspondentes foram registradas em um caderno de campo, e as atribuições das camadas foram verificadas quanto à precisão por MBK no campo e também contra fotografias de alta resolução. Os números e locais das amostras são exibidos nas Figs suplementares.1 - 3 . Todos os materiais foram adquiridos como parte de um acordo de cooperação científica entre o Instituto de Arqueologia e Etnografia, Ramo Siberiano da Academia Russa de Ciências e o Instituto Max Planck de Antropologia Evolutiva para projetos na área de paleogenética no Norte da Ásia, assinado em 25 Dezembro de 2018 e válido por um período de cinco anos. O Instituto de Arqueologia e Etnografia, filial siberiana da Academia Russa de Ciências supervisiona as escavações na caverna Denisova e obteve todas as autorizações necessárias para a realização de pesquisas arqueológicas de campo associadas a este projeto do Ministério da Cultura da Federação Russa.
Escala de tempo comum para as Câmaras Principal e Leste
We constructed a common time scale for the Pleistocene stratigraphic sequences in Main and East Chambers, but excluded South Chamber because of various stratigraphic complications that preclude definitive layer assignments to most of the Pleistocene deposit exposed at the time of sample collection (Supplementary Information section 2).
The stratified sequences in each chamber are numbered by layer, but layers with the same number cannot be traced stratigraphically between the chambers and are not necessarily equivalent in age. To correlate stratigraphic layers between chambers, a previous study8 constructed a separate Bayesian statistical age model for each chamber using their large dataset of optical ages, and then established isochrons (lines of equal age) between the chambers, using the modelled start and end ages for each depositional phase (figure 3 and extended data table 1 in ref. 8). Bayesian age models were constructed separately for each chamber because there is no a priori reason to assume that sediments have accumulated continuously, or at the same rate, in each of the chambers. Time gaps in the stratigraphic sequence may therefore differ between the chambers owing to erosional events or periods of little or no sediment deposition.
Here we created a common time scale for the Pleistocene stratigraphic sequences in Main and East Chambers, using the modelled start and end ages for each depositional phase and the modelled time gaps (Extended Data Fig. 1). To construct this time scale, only point estimates of modelled age and not their associated uncertainties were taken into consideration; for example, if the start and end ages for a specific phase are 100 ± 10 and 50 ± 5 ka, respectively, then the corresponding time depth (size of coloured box in Extended Data Fig. 1a, b) is estimated to extend from 100 to 50 ka. The modelled ages for Main and East Chambers were projected horizontally onto a linear scale to derive a common time scale for the depositional phases in these two chambers (Extended Data Fig. 1c). We use the common timeline to display the mtDNA data and to show (Extended Data Fig. 1d) the Bayesian modelled ages (95.4% highest posterior density interval) for individual hominin fossils5.
Sampling, DNA extraction, library preparation and shotgun sequencing
Sampling, DNA extraction and library preparation were performed in a dedicated clean room at the Max Planck Institute for Evolutionary Anthropology (Leipzig). Sediment subsamples of between 29 and 191 mg were transferred into 2-ml tubes using antistatic spatulas. Before use, each spatula was soaked in 12% bleach for approximately 15 min with occasional mixing, washed thoroughly with water, dried and UV-treated with a dose of 7 J cm−2 in a UV-C crosslinker from both sides. One ml of extraction buffer29 was added to subsamples with less than 100 mg of sediment, and 2 ml to samples with 100 mg or more. The resulting lysates were incubated overnight at 37 °C on rotation. Aliquots of 150 μl of lysate were purified on a BRAVO NGS workstation B (Agilent Technologies) following a previously described bead-based protocol29 using binding buffer ‘D’. The remaining lysate was stored at –20 °C. Negative controls containing no sample material were included for both the lysis and purification steps. Supplementary Data 1 provides sample information.
The entire volume of DNA extract (30 μl) from each sample was converted into a single-stranded DNA library using the BRAVO NGS workstation B as previously described30. Approximately six million molecules of a control oligonucleotide were added into each sample during the library preparation to evaluate the potential presence of inhibitors, which may reduce the efficiency of the process31. The number of sample and control library molecules obtained in each reaction was determined using two real-time PCR assays30. All libraries were tagged with two indices, amplified to PCR plateau, and purified as described in the aforementioned protocol30.
Aliquots of 1 μl from each library were pooled into sets of up to 92 samples (including controls) and sequenced on an Illumina MiSeq v3 platform in 76 cycle paired-end reads using micro or nano flowcells. Base calling was completed using Bustard (Illumina). The resulting shotgun data were evaluated only for the presence of the expected index pairs, which was confirmed in all sequencing runs.
Hybridization capture for mammalian and hominin mtDNA
Each library was enriched separately with two mtDNA probe sets, one targeting 242 mammalian mtDNA genomes13 and the other using the revised Cambridge Reference Sequences (rCRS32) for targeting hominin mtDNA, in two successive rounds of on-bead hybridization capture following a previously published protocol13 with minor adjustments. All capture reactions were performed in sets of 384 samples (including extraction and library negative controls), using the BRAVO NGS workstation B. For some samples, 30 PCR cycles were performed in post-capture amplification of the enriched libraries (thus reaching PCR plateau for all libraries). For others, PCR cycles were reduced to between 12 and 16.
Bibliotecas capturadas foram agrupadas combinando alíquotas de 5 μl de cada biblioteca em conjuntos de aproximadamente 92 bibliotecas (incluindo controles) para bibliotecas enriquecidas de mtDNA de mamíferos e conjuntos de aproximadamente 180 bibliotecas (incluindo controles) para bibliotecas enriquecidas de mtDNA humano. Cada pool foi sequenciado na plataforma Illumina HiSeq 2500. A chamada de base foi concluída usando Bustard (Illumina). Os dados de captura resultantes foram avaliados conforme descrito nas seções de Informações Suplementares 3 , 4 quanto à presença de DNA de mamífero antigo e / ou hominídeo.
Identificação de taxa de mamíferos
O processamento inicial dos dados de captura de mamíferos foi realizado usando um pipeline 13 descrito anteriormente . Em resumo, as leituras de extremidades pareadas sobrepostas foram fundidas em sequências de moléculas de comprimento total usando leeHom 33 ( https://bioinf.eva.mpg.de/ ) e mapeadas para os 242 genomas mitocondriais de mamíferos incluídos no projeto da sonda de captura 13 . As leituras que não podiam ser mescladas com sobreposição, sequências não mapeadas e sequências menores que 35 pares de bases (bp) foram removidas. Apenas uma única sequência foi retida de duplicatas mostrando identidade de sequência perfeita, e sequências com menos de duas duplicatas foram removidas. Todas as sequências únicas foram então atribuídas a táxons de mamíferos em nível de família usando BLAST 34e o algoritmo ancestral comum mais baixo implementado no MEGAN 35 . Sequências atribuídas a cada família foram mapeadas para os genomas mitocondriais de todas as espécies disponíveis dentro de cada família (Dados Suplementares 3 ), exigindo uma pontuação de qualidade de mapeamento de pelo menos 25, e sequências duplicadas residuais foram removidas usando bam-rmdup ( https: // github .com / mpieva / biohazard-tools ) com base no compartilhamento de coordenadas de início e fim de alinhamento idênticas. O genoma do mtDNA que produziu o maior número de sequências alinhadas após a remoção de duplicatas de PCR foi então usado para determinar o número de fragmentos de DNA atribuídos à respectiva família, bem como a frequência de substituições de citosina (C) por timina (T) no fragmento termina.
A identificação de 'táxons antigos' (isto é, táxons para os quais as sequências de DNA antigas foram recuperadas) foi realizada de acordo com os seguintes critérios: (1) o número de fragmentos atribuídos a um determinado táxon deve compreender pelo menos 1% do número total de fragmentos identificados taxonomicamente; (2) pelo menos 10 fragmentos supostamente desaminados (isto é, fragmentos mostrando substituições de C para T nas bases 5 'e / ou 3' terminais) tinham que estar presentes; (3) a frequência das substituições de C para T deve ser significativamente maior do que 10% com base em intervalos de confiança binomiais de 95% em um ou ambos os terminais; e (4) os fragmentos tinham que produzir uma cobertura de pelo menos 500 pb do genoma de referência do mtDNA. Os Dados Suplementares 1 fornecem informações sobre cada amostra e controles. O teste estatístico foi realizado usando R versão 3.5.1.
Identificação de fragmentos de DNA de hominídeos antigos
O processamento dos dados de captura de mtDNA de hominíneo foi realizado conforme descrito anteriormente 13 , usando um pipeline de análise que diferia do processamento dos dados de captura de mtDNA de mamífero (Seção 3 de Informações Suplementares ) apenas em que as sequências de moléculas de comprimento total foram mapeadas para o rCRS de o genoma mitocondrial humano, e duplicatas foram removidas com base nas coordenadas de início e fim de alinhamento usando bam-rmdup ( https://github.com/mpieva/biohazard-tools ) antes que as sequências fossem atribuídas a táxons de mamíferos.
Para garantir que todas as bibliotecas foram sequenciadas com uma profundidade suficiente, as bibliotecas que produziram menos de três duplicatas de sequência, em média, foram sequenciadas mais profundamente e os dados finais foram mesclados para análise. A presença de DNA de hominídeo antigo foi determinada de acordo com os seguintes critérios: (1) o número de fragmentos atribuídos a hominídeos deveria compreender pelo menos 1% do número total de fragmentos identificados taxonomicamente; (2) pelo menos 10 fragmentos supostamente desaminados (isto é, fragmentos que mostram uma substituição C-para-T nas três bases 5 'e / ou 3' terminais) tinham que estar presentes; e (3) a frequência das substituições de C para T deve ser significativamente maior do que 10% com base em intervalos de confiança binomiais de 95% em ambos os terminais. Para algumas amostras, várias subamostras foram retiradas e de alguns lisados, várias bibliotecas foram preparadas.Os dados resultantes foram analisados em uma base por biblioteca, bem como mesclados por lisado (subamostra). Dados suplementares 1 fornece resultados de dados individuais e mesclados e informações sobre cada amostra e controle. O teste estatístico foi realizado usando R versão 3.5.1.
Resumo do relatório
Mais informações sobre o desenho da pesquisa estão disponíveis no Nature Research Reporting Summary, vinculado a este artigo.
Disponibilidade de dados
The mitochondrial consensus sequences reported from Main Chamber layers 19 (M65) and 20 (M71), and from East Chamber layers 11.4 (E202) and 11.4/12.1 (E213), are available in the Dryad digital repository (https://doi.org/10.5061/dryad.k3j9kd567), and the raw data for each mammalian mtDNA and human mtDNA enriched library are available in the European Nucleotide Archive under accession number PRJEB44036. Any other relevant data are available from the corresponding authors upon reasonable request.
References
- 1.
Krause, J. et al. The complete mitochondrial DNA genome of an unknown hominin from southern Siberia. Nature 464, 894–897 (2010).
- 2.
Meyer, M. et al. A high-coverage genome sequence from an archaic Denisovan individual. Science 338, 222–226 (2012).
- 3.
Sawyer, S. et al. Nuclear and mitochondrial DNA sequences from two Denisovan individuals. Proc. Natl Acad. Sci. USA 112, 15696–15700 (2015).
- 4.
Slon, V. et al. A fourth Denisovan individual. Sci. Adv. 3, e1700186 (2017).
- 5.
Douka, K. et al. Age estimates for hominin fossils and the onset of the Upper Palaeolithic at Denisova Cave. Nature 565, 640–644 (2019).
- 6.
Prüfer, K. et al. The complete genome sequence of a Neanderthal from the Altai Mountains. Nature 505, 43–49 (2014).
- 7.
Slon, V. et al. The genome of the offspring of a Neanderthal mother and a Denisovan father. Nature 561, 113–116 (2018).
- 8.
Jacobs, Z. et al. Timing of archaic hominin occupation of Denisova Cave in southern Siberia. Nature 565, 594–599 (2019).
- 9.
Shunkov, M. V., Kozlikin, M. B. & Derevianko, A. P. Dynamics of the Altai Paleolithic industries in the archaeological record of Denisova Cave. Quat. Int. 559, 34–46 (2020).
- 10.
Shunkov, M. V., Fedorchenko, A. Y., Kozlikin, M. B. & Derevianko, A. P. Initial Upper Palaeolithic ornaments and formal bone tools from the East Chamber of Denisova Cave in the Russian Altai. Quat. Int. 559, 47–67 (2020).
- 11.
Derevianko, A. P., Shunkov, M. V. & Kozlikin, M. B. Who were the Denisovans? Archaeol. Ethnol. Anthropol. Eurasia 48, 3–32 (2020).
- 12.
Sawyer, S. Insights into Neandertals and Denisovans from Denisova Cave. PhD thesis, Univ. Leipzig (2016).
- 13.
Slon, V. et al. Neandertal and Denisovan DNA from Pleistocene sediments. Science 356, 605–608 (2017).
- 14.
Slon, V. et al. Captura mitocondrial de mamíferos, uma ferramenta para rastreamento rápido de preservação de DNA em restos faunísticos e não diagnósticos, e sua aplicação a espécimes do Pleistoceno Médio da Caverna Qesem (Israel). Quat. Int . 398 , 210–218 (2016).
- 15
Briggs, AW et al. Padrões de dano em sequências de DNA genômico de um Neandertal. Proc. Natl Acad. Sci. USA 104 , 14616–14621 (2007).
- 16
Sawyer, S., Krause, J., Guschanski, K., Savolainen, V. & Pääbo, S. Temporal patterns of nucleotide misincorporations and DNA fragmentation in Ancient DNA. PLoS ONE 7 , e34131 (2012).
- 17
Haile, J. et al. Cronologia do DNA antigo dentro dos depósitos de sedimentos: as reconstruções paleobiológicas são possíveis e a lixiviação do DNA é um fator? Mol. Biol. Evol . 24 , 982–989 (2007).
- 18
Meyer, M. et al. Sequência do genoma mitocondrial de um hominíneo de Sima de los Huesos. Nature 505 , 403-406 (2014).
- 19
Meyer, M. et al. Sequências de DNA nuclear dos hominíneos Sima de los Huesos do Pleistoceno Médio. Nature 531 , 504–507 (2016).
- 20
Peyrégne, S. et al. O DNA nuclear de dois primeiros Neandertais revela 80.000 anos de continuidade genética na Europa. Sci. Adv . 5 , eaaw5873 (2019).
- 21
Bray, N. L., Pimentel, H., Melsted, P. & Pachter, L. Near-optimal probabilistic RNA-seq quantification. Nat. Biotechnol. 34, 525–527 (2016).
- 22.
Vernot, B. et al. Unearthing Neanderthal population history using nuclear and mitochondrial DNA from cave sediments. Science 372, eabf1667 (2021).
- 23.
Titov, V. V. Habitat conditions for Camelus knoblochi and factors in its extinction. Quat. Int. 179, 120–125 (2008).
- 24.
Westbury, M. V. et al. Hyena paleogenomes reveal a complex evolutionary history of cross-continental gene flow between spotted and cave hyena. Sci. Adv. 6, eaay0456 (2020).
- 25.
Agadjanian, A. K. & Shunkov, M. V. Paleolithic man of Denisova Cave and zoogeography of Pleistocene mammals of northwestern Altai. Paleontol. J. 52, 66–89 (2018).
- 26.
Morley, M. W. et al. Hominin and animal activities in the microstratigraphic record from Denisova Cave (Altai Mountains, Russia). Sci. Rep. 9, 13785 (2019).
- 27.
Zhang, D. et al. Denisovan DNA in Late Pleistocene sediments from Baishiya Karst Cave on the Tibetan Plateau. Science 370, 584–587 (2020).
- 28.
Prokopenko, A. A., Hinnov, L. A., Williams, D. F. & Kuzmin, M. I. Orbital forcing of continental climate during the Pleistocene: a complete astronomically tuned climatic record from Lake Baikal, SE Siberia. Quat. Sci. Rev. 25, 3431–3457 (2006).
- 29.
Rohland, N., Glocke, I., Aximu-Petri, A. & Meyer, M. Extração de DNA altamente degradado de ossos, dentes e sedimentos antigos para sequenciamento de alto rendimento. Nat. Protocols 13, 2447–2461 (2018).
- 30.
Gansauge, MT, Aximu-Petri, A., Nagel, S. & Meyer, M. Preparação manual e automática de bibliotecas de DNA de fita simples para o sequenciamento de DNA de vestígios biológicos antigos e outras fontes de DNA altamente degradado. Nat. Protocolos 15, 2279–2300 (2020).
- 31
Glocke, I. & Meyer, M. Estendendo o espectro de sequências de DNA recuperadas de ossos e dentes antigos. Genome Res . 27 , 1230–1237 (2017).
- 32
Andrews, R. M. et al. Reanalysis and revision of the Cambridge reference sequence for human mitochondrial DNA. Nat. Genet. 23, 147 (1999).
- 33.
Renaud, G., Stenzel, U. & Kelso, J. leeHom: adaptor trimming and merging for Illumina sequencing reads. Nucleic Acids Res. 42, e141 (2014).
- 34.
Altschul, S. F., Gish, W., Miller, W., Myers, E. W. & Lipman, D. J. Basic local alignment search tool. J. Mol. Biol. 215, 403–410 (1990).
- 35.
Huson, D. H., Auch, A. F., Qi, J. & Schuster, S. C. MEGAN analysis of metagenomic data. Genome Res. 17, 377–386 (2007).
Reconhecimentos
Este projeto foi financiado pela Max Planck Society, o European Research Council (contrato de concessão nº 694707 para SP) e o Australian Research Council (bolsas FT150100138 para ZJ, FT140100384 para BL e FL130100116 para RGR). KO foi apoiado por um Prêmio do Programa de Treinamento em Pesquisa do Governo Australiano. MVS, MBK e APD foram apoiados por RFBR, projeto no. 20-29-01011. VS reconhece o financiamento da Alon Fellowship. Agradecemos a S. Grote, L. Jauregui, S. Lin, F. Mafessoni e L. Skov pela ajuda na organização do trabalho de laboratório e visualização de dados; J. Lihanova, F. Müller, B. Schellbach e A. Weihmann pela ajuda com a preparação e sequenciamento de amostras; e V. Vaneev pela assistência de campo.
Financiamento
Financiamento de acesso aberto fornecido pela Max Planck Society.
Informação sobre o autor
Afiliações
Contribuições
EIZ, ZJ, SP, RGR e MM desenharam o estudo. ZJ, BL, KO e RGR coletaram amostras em campo. MVS, MBK, APD, ZJ e RGR forneceram contexto e interpretação arqueológica, estratigráfica e geocronológica. EIZ, EE, SN, JR e AS realizaram experimentos de laboratório. EIZ, ZJ, BV, CdF, FR, VS e JK realizaram, auxiliaram ou supervisionaram a análise e visualização dos dados. EIZ, ZJ, RGR e MM escreveram o manuscrito com contribuições de todos os autores.
Autores correspondentes
Declarações de ética
Interesses competitivos
Os autores declaram não haver interesses conflitantes.
Informação adicional
Informações da revisão por pares A Nature agradece Katerina Douka, Peter Heintzman e David Reich por sua contribuição para a revisão por pares deste trabalho. Relatórios de revisores pares estão disponíveis.
Nota do editor A Springer Nature permanece neutra em relação a reivindicações jurisdicionais em mapas publicados e afiliações institucionais.
Figuras e tabelas de dados estendidos
Dados estendidos Fig. 1 Cronologias para camadas sedimentares e fósseis de hominídeos.
a , b , Sequências estratigráficas esquemáticas para a Câmara Leste ( a ) e Câmara Principal ( b ), com idades e atribuições de camada de uma publicação anterior 8. As associações arqueológicas para cada camada são codificadas por cores para diferenciar entre as quatro fases principais do artefato (Paleolítico médio inicial, verde escuro; Paleolítico médio médio, verde claro; Paleolítico superior inicial, laranja escuro; Paleolítico superior, laranja claro). As áreas brancas indicam camadas sem arqueologia, áreas cinza escuro denotam intervalos de tempo modelados e áreas cinza claro denotam intervalos de tempo entre estimativas pontuais de idade para camadas sucessivas que têm incertezas de idade sobrepostas. Os símbolos de espátula indicam as camadas das quais os mtDNAs derivados de sedimentos Denisovan (vermelho) e Neandertal (azul) foram recuperados em um estudo piloto 13 (corrigido para a atribuição incorreta de uma amostra na Câmara Leste para a camada 14 em vez da camada 11.4). c, Escala de tempo comum construída para as câmaras principal e oriental (métodos) para facilitar a comparação entre as camadas e exibir dados de mtDNA. d , Faixas etárias (95,4% de densidade posterior mais alta (HPD)) para fósseis de Neandertais (azul), Denisovanos (vermelho) e descendentes Neandertal-Denisovanos (Denisova 11, ambas as cores), plotados na escala de tempo comum. As idades dos fósseis foram estimadas usando um modelo bayesiano que incorpora radiocarbono, idades das séries óticas e do urânio, informações estratigráficas e dados genéticos gerados a partir de sequências de DNA de hominíneos 5 .
Dados estendidos Fig. 2 Localizações de amostras de DNA de sedimentos.
a, Plan of cave interior, showing location of each chamber. Red lines denote stratigraphic profiles sampled in 2017. Grid coordinates for the excavation squares are shown along the top and left side of the plan, and the corresponding squares (each consisting of a Cyrillic letter and a number) are shown at the top of the stratigraphic profiles in Fig. 1a–c, Extended Data Figs. 3a, b, 5, 8, 9, Supplementary Figs. 1, 2, 3b, d. b, Southeast profile of Main Chamber after excavations in 2016. c, Southeast profile of East Chamber after excavation in 2015. d, Northwest profile of East Chamber after excavation in 2016. White circles represent samples collected for sediment DNA analysis. Individual sample numbers are provided in Supplementary Figs. 1 (Main) and 2 (East); sample numbers referred to in the text are indicated in b, c. Sample locations in South Chamber are indicated in Extended Data Fig. 3a, b, Supplementary Fig. 3.
Extended Data Fig. 3 Ancient hominin and mammalian mtDNA data for sediment samples in South Chamber.
a, b, Sample locations in upper section (a) and lower section (b) of southeast profile, using the same symbols for hominins as in Fig. 1. Layer 11 (associated with the Initial Upper Palaeolithic) and layer 22 are numbered, but other layers are denoted as either deformed Middle Palaeolithic (dMP) or as phosphate deformation deposits (pdd-9 and -12). The dMP and pdd deposits have been substantially affected by post-depositional deformation and phosphatization, respectively (Supplementary Information section 2). c, Proportion of mtDNA fragments assigned to different families of large mammal in each layer, arranged in relative stratigraphic order. Values were obtained by averaging across the percentages of fragments assigned to each family in all samples from a specified layer. d, The 5′ C-to-T substitution frequencies (putative deamination rates, in per cent) of mtDNA fragments assigned to bovid, canid, hyaenid and ursid in each layer. Individual values are shown as vertical bars, and the mean and s.d. values by black symbols. e, Average size (base pairs (bp)) of mammalian mtDNA fragments in each layer; individual values are shown as vertical bars and the mean and s.d. values by black symbols. f, Number of unique mtDNA fragments in each layer assigned to mammalian taxa per milligram of sediment from each library; individual values are shown as vertical bars and the mean and s.d. values by black symbols. Spearman’s correlation test (one-sided) was used to test for correlation between stratigraphic depth (layer) and 5′ C-to-T substitution frequency (minimum n = 112, positive correlation, maximum P = 1.8 × 10−5), number of unique ancient mtDNA fragments (minimum n = 79, negative correlation, maximum P = 1.7 × 10−5), and average fragment size (minimum n = 79, negative correlation, maximum P = 1.8 × 10−8).
Extended Data Fig. 4 Ancient mammalian mtDNA preservation in Main and East Chambers.
a, The 5′ C-to-T substitution frequencies (putative deamination rates, in per cent) of mtDNA fragments assigned to bovid, canid, hyaenid and ursid, plotted as a function of time. b, c, Average size of mammalian mtDNA fragments (b) and number of unique mtDNA fragments assigned to taxa per milligram of sediment from each library (c), plotted as a function of time. Individual values for Main and East Chambers are shown as brown and green circles, respectively, and the mean and s.d. values by black symbols. The latter symbols are positioned on the time axis at the mean age for the relevant layer, and the time intervals for the corresponding layers in Main and East Chambers are denoted by brown and green shading, respectively (Extended Data Fig. 1). Spearman’s correlation test (one-sided) was used to test for correlation between stratigraphic depth (layer) and 5′ C-to-T substitution frequency (minimum n = 133, positive correlation, maximum P = 2.7 × 10−8), number of unique ancient mtDNA fragments (minimum n = 222, negative correlation, maximum P = 0.02), and average fragment size (minimum n = 222, negative correlation, maximum P = 2.0 × 10−25).
Extended Data Fig. 5 Abundance of ancient mammalian and hominin mtDNA in Main and East Chambers.
a , b , Número de fragmentos de mtDNA exclusivos atribuídos a táxons de mamíferos ( a ) e número de fragmentos desaminados atribuídos a hominíneos ( b ) por miligrama de sedimento de cada biblioteca, para amostras na Câmara Principal (perfil sudeste) e Câmara Leste (sudeste e perfis noroeste). Fragmentos de mamíferos antigos abrangem todos os fragmentos de mtDNA atribuídos a famílias consideradas como contendo DNA antigo com base em sinais de desaminação de citosina. A intensidade do sombreamento em círculos preenchidos reflete a abundância relativa; as amostras que não produziram mtDNA antigo são mostradas como círculos brancos. O sombreamento de fundo em a denota camadas que foram depositadas sob condições relativamente frias (azul) ou relativamente quentes (laranja) 8 , e emb denota camadas associadas ao início do Paleolítico Médio (verde escuro), médio Paleolítico Médio (verde claro), Paleolítico Superior inicial (laranja escuro) e Paleolítico Superior (laranja claro) assembléias arqueológicas 8 .
Dados estendidos Fig. 6 Árvores filogenéticas para sequências de mtDNA atribuídas a linhagens específicas de hominina e consistência da recuperação de mtDNA de hominina a partir de amostras de sedimentos individuais.
a , b , Árvore de união de vizinhos construída com sequências de consenso de mtDNA de neandertal recém-construídas (em negrito) e genomas de mtDNA de neandertal publicados (apenas genes codificadores de proteína) ( a ) e genomas de mtDNA de Denisovan e Sima de los Huesos publicados e um Denisovan recém-reconstruído Consenso de mtDNA (apenas genes codificadores de proteínas) ( b ). Os taxa foram agrupados com base em diferenças de pares, com suporte para cada nó baseado em um teste de bootstrap com 500 repetições. Posições com falta de dados ou de lacunas foram removidos, resultando em 9,219 e 7,495 posições usadas para análise em uma e b , respectivamente. c, Recuperação de fragmentos de mtDNA de hominíneos antigos e identificação de linhagens de hominíneos nos casos em que várias bibliotecas foram preparadas a partir da mesma amostra de sedimento. Cada retângulo representa uma biblioteca e mostra o número correspondente de fragmentos desaminados. Bibliotecas produzidas a partir da mesma subamostra de sedimentos são mostradas por retângulos adjacentes; bibliotecas de diferentes subamostras são mantidas separadas.
Dados estendidos Fig. 7 Proporções de fragmentos de mtDNA de mamíferos e restos de esqueletos nas câmaras principal e leste.
a, b, Proportion (in per cent) of ancient DNA fragments (red) and skeletal remains (blue)8 assigned to various families of large (a) and small (b) mammals, combined for all samples in Main and East Chambers and ranked in descending order of mtDNA percentage. c, d, Proportion (in per cent) of ancient mtDNA fragments and skeletal remains assigned to families of large mammals for individual layers in Main Chamber (c) and East Chamber (d), arranged in relative stratigraphic order. The mtDNA values were obtained by averaging across the percentages of fragments assigned to each family in all samples from a specified layer.
Dados estendidos Fig. 8 Proporções de fragmentos de mtDNA antigos de grandes mamíferos na Câmara Principal.
Gráficos de pizza mostrando proporções de fragmentos de mtDNA atribuídos a famílias de mamíferos específicas para cada amostra; círculos vazios denotam amostras que não produziram mtDNA antigo.
Dados estendidos Fig. 9 Proporções de fragmentos de mtDNA antigos de grandes mamíferos na Câmara Leste.
a , b , Gráficos de pizza mostrando proporções de fragmentos de mtDNA atribuídos a famílias específicas de mamíferos para cada amostra nos perfis sudeste ( a ) e noroeste ( b ).
Dados estendidos Fig. 10 Números e proporções de fragmentos de mtDNA antigos atribuídos a diferentes grupos de mtDNA de ursídeos, hienídeos e elefantídeos nas câmaras principal e leste.
Círculos em preto e branco denotam o número total de amostras atribuídas a um clado de mtDNA específico, plotado em função do tempo (idade média para a camada relevante). As barras correspondentes são codificadas por cores com base na proporção (em por cento) das amostras atribuídas a cada grupo e se estendem ao longo dos intervalos de tempo para as respectivas camadas nas Câmaras Principal e Leste.
Informação suplementar
Informação suplementar
Este arquivo contém uma Discussão Suplementar, Referências Suplementares, Tabelas Suplementares 1-10 e Figuras Suplementares 1-44.
Dados Suplementares 1
Resumo dos lisados, extratos e bibliotecas de DNA e resultados da captura de mtDNA em mamíferos e humanos.
Dados Suplementares 2
Resumo dos resultados dos testes de pH.
Dados Suplementares 3
Genomas de mtDNA usados como referências para diferentes análises.
Direitos e permissões
Acesso aberto Este artigo está licenciado sob uma Licença Internacional Creative Commons Atribuição 4.0, que permite o uso, compartilhamento, adaptação, distribuição e reprodução em qualquer meio ou formato, desde que você dê os devidos créditos ao (s) autor (es) original (is) e à fonte, fornecer um link para a licença Creative Commons e indicar se foram feitas alterações. As imagens ou outro material de terceiros neste artigo estão incluídos na licença Creative Commons do artigo, a menos que indicado de outra forma em uma linha de crédito para o material. Se o material não estiver incluído na licença Creative Commons do artigo e seu uso pretendido não for permitido por regulamentação legal ou exceder o uso permitido, você precisará obter permissão diretamente do detentor dos direitos autorais. Para ver uma cópia desta licença, visitehttp://creativecommons.org/licenses/by/4.0/ .
Sobre este artigo
Cite este artigo
Zavala, EI, Jacobs, Z., Vernot, B. et al. O DNA do sedimento do pleistoceno revela alterações de hominídeos e faunísticas na caverna Denisova. Nature 595, 399–403 (2021). https://doi.org/10.1038/s41586-021-03675-0
Recebido
Aceitaram
Publicados
Data de emissão
Comentários
Ao enviar um comentário, você concorda em cumprir nossos Termos e Diretrizes da comunidade . Se você encontrar algo abusivo ou que não esteja de acordo com nossos termos ou diretrizes, sinalize como impróprio.

Nenhum comentário:
Postar um comentário
Observação: somente um membro deste blog pode postar um comentário.