A história genômica do lobo cinzento revela uma dupla ascendência de cães
Abstrato
O lobo cinzento ( Canis lupus ) foi a primeira espécie a dar origem a uma população doméstica, e manteve-se difundido durante a última Idade do Gelo, quando muitas outras espécies de grandes mamíferos foram extintas. Pouco se sabe, no entanto, sobre a história e possível extinção de populações de lobos do passado ou quando e onde os progenitores de lobos da atual linhagem canina ( Canis familiaris ) viveram 1 , 2 , 3 , 4 , 5 , 6 , 7 , 8.
Aqui analisamos 72 genomas de lobos antigos nos últimos 100.000 anos da Europa, Sibéria e América do Norte. Descobrimos que as populações de lobos estavam altamente conectadas ao longo do Pleistoceno Superior, com níveis de diferenciação uma ordem de magnitude menor do que são hoje. Essa conectividade populacional nos permitiu detectar a seleção natural ao longo das séries temporais, incluindo a rápida fixação de mutações no gene IFT8840.000-30.000 anos atrás.
Mostramos que os cães estão mais intimamente relacionados aos lobos antigos da Eurásia oriental do que aos da Eurásia ocidental, sugerindo um processo de domesticação no leste. No entanto, também descobrimos que os cães no Oriente Próximo e na África derivam até metade de sua ancestralidade de uma população distinta relacionada aos lobos modernos do sudoeste da Eurásia, refletindo um processo de domesticação independente ou mistura de lobos locais. Nenhum dos genomas de lobos antigos analisados é uma correspondência direta para qualquer uma dessas ascendências de cães, o que significa que as populações progenitoras exatas ainda precisam ser localizadas.
Principal
O lobo cinzento ( Canis lupus ) esteve presente na maior parte do hemisfério norte nas últimas centenas de milhares de anos e, ao contrário de muitos outros grandes mamíferos, não foi extinto no final do Pleistoceno. Estudos de genomas atuais descobriram que a estrutura populacional atual se formou principalmente nos últimos ~ 30.000-20.000 anos 9 , 10 , 11 , ou aproximadamente desde o Último Máximo Glacial (LGM; ~ 28-23 mil anos atrás (ka) 12 ) . Os lobos siberianos anteriores ao LGM têm ancestrais que são em grande parte basais à diversidade atual, o que levou a sugestões de que muitas linhagens de lobos pré-LGM foram extintas 13 , 14. Entre as questões centrais está, portanto, em que medida a população global de lobos esteve sujeita a processos de extinção ou respondeu às mudanças climáticas com novas adaptações.
Embora seja claro que os lobos cinzentos deram origem aos cães, não há consenso sobre quando, onde e como isso aconteceu 1 , 2 , 3 , 4 , 5 , 6 , 7 , 8 . Restos esqueléticos atribuíveis à atual linhagem canina aparecem arqueologicamente em 14 ka 15 , e as estimativas genéticas de quando os ancestrais dos cães e lobos modernos divergiram variam de 40 a 14 ka 9 , 13 , 16.
No entanto, dados genéticos de cães modernos e antigos acoplados a lobos modernos, aos quais estudos anteriores foram amplamente restritos, podem não ser capazes de resolver a origem dos cães. A diversidade genética dentro dos cães é afetada por sua história dinâmica e é incapaz de identificar com segurança uma origem. As relações com os lobos modernos também podem ser afetadas pela extinção local e fluxo gênico desde a domesticação 6 , 9 . As regiões onde os primeiros cães foram encontrados também não implicam necessariamente locais de origem, pois a existência de cães anteriores em outros lugares não pode ser excluída. Em vez disso, a origem dos cães poderia ser resolvida se a diversidade genética do lobo no espaço e no tempo fosse exaustivamente caracterizada e pudesse ser determinado quais populações estavam mais próximas dos ancestrais dos cães.
Genomas de lobo abrangendo 100.000 anos
Sequenciamos 66 novos genomas de lobos antigos da Europa, Sibéria e noroeste da América do Norte para uma mediana de cobertura de 1 × (intervalo, 0,02-13 ×) (Fig. 1a,b ), incorporamos cinco genomas de lobos antigos previamente sequenciados 14 , 17 e aumento da cobertura para um 13 .
Também sequenciamos um genoma de dhole antigo do Cáucaso, contextualmente datado de >70 ka, para servir como um grupo externo. Frações de DNA do cromossomo X mostraram que 69% dos lobos eram do sexo masculino (intervalo de confiança de 95% (IC), 57-80%; P = 0,0013, teste binomial), espelhando a sobre-representação masculina entre genomas antigos de mamutes lanosos 18 , bisonte 19 , ursos marrons 19 e cães domésticos 8. Para lobos sem datas ou com datas além do limite de radiocarbono de ~50 ka, estimamos as idades através da datação da ponta mitocondrial 20 e obtivemos um IC médio de 95% de 21.573 anos e um erro de previsão médio de 5.133 anos (Figs. 1 e 2 complementares ) . Nós mesclamos genótipos de polimorfismo de nucleotídeo único (SNP) chamados desses genomas com os de lobos modernos em todo o mundo ( n = 68), cães modernos ( n = 369) e antigos ( n = 33) e outras espécies de canídeos ( Métodos ). O conjunto de dados total abrange os últimos 100.000 anos (Fig. 1b ).
a , Locais de amostragem de lobos antigos e um dhole antigo analisado aqui, em um mapa base da Terra Natural ( naturalearthdata.com ). b , Idades e cobertura de sequenciamento de lobos antigos. c , PC1 de um PCA no grupo externo f 3 -estatísticas plotadas em relação à idade da amostra. Os PCs foram calculados apenas a partir de lobos antigos, com lobos e cães atuais projetados no enredo. d , estimativas de heterozigosidade a partir de amostragem de duas leituras em locais verificados como heterozigotos em um coiote. Barras denotam 95% CIs de jackknifing de bloco.
Em uma análise de componentes principais (PCA) em uma matriz de deriva genética compartilhada, os lobos antigos se agruparam fortemente por idade e não por geografia ( r PC1 de Pearson, idade da amostra = 0,85, P = 5 × 10 −21 ) (Fig. 1c ) . Da mesma forma, os lobos antigos compartilham mais deriva com os lobos modernos quanto mais jovens eles são (Dados Estendidos Fig. 1a e Fig. 3 Complementar ).
Estudos anteriores sugeriram uma rotatividade de ascendência LGM 13 , 14 , 21 , e, de fato, descobrimos que todos os indivíduos mais jovens que o LGM (ou seja, pós-namorando 23 ka) eram mais semelhantes entre si do que lobos anteriores a ~ 28 ka (Extended Dados Fig. 1b). No entanto, o mesmo padrão também é visível ao contrastar afinidades com lobos mais jovens versus lobos mais velhos em qualquer ponto durante os últimos 100.000 anos (Fig. 4 Suplementar ). Usando simulações, confirmamos que as relações temporais observadas são muito semelhantes ao que seria esperado em uma população panmítica (Fig. 5 Suplementar ). Um processo de longa data de homogeneização de ancestralidade devido à conectividade parece ter impulsionado as relações de lobo do Pleistoceno. As mudanças durante o LGM representam, portanto, não uma mudança na dinâmica populacional de longo prazo, mas a manifestação mais recente desse processo.
Sibéria como fonte de fluxo gênico global
Em seguida, testamos a direcionalidade no fluxo gênico que conectou a ascendência do lobo ao longo do tempo. Análises usando estatísticas de f 4 mostraram que todos os lobos pós-data de 23 ka são mais semelhantes aos lobos siberianos do que aos lobos europeus ou da Ásia Central de ~ 30 ka (Dados Estendidos Fig. 1c e Fig. 6 Suplementar ). Isso sugere que a ascendência relacionada à Sibéria se expandiu para a Europa, de acordo com as evidências mitocondriais 21 . A mesma dinâmica do fluxo de genes siberianos para a Europa se desenrolou entre 50 e 35 ka (Fig. 6 Suplementar ). Descobrimos que um modelo de gráfico de mistura com fluxo gênico recorrente e unidirecional da Sibéria para a Europa poderia explicar essas relações (Fig. 2ae Fig. 8 Suplementar ). Embora não pudéssemos distinguir o fluxo gênico contínuo do tipo pulso, nossos resultados sugerem que a Sibéria atuou como fonte e a Europa como sumidouro para a migração ao longo do Pleistoceno Superior e não mostram evidências de fluxo gênico na outra direção (Dados Estendidos Fig. 1d e Fig. 7 Complementar ).
a , Gráfico de mistura ajustado por qpGraph para lobos antigos selecionados, com duas estatísticas atípicas (| Z | > 3) f (pior = 3,16). b , Modelos qpAdm mais adequados para lobos pós-LGM e atuais. Um antigo dhole foi usado como grupo externo para lobos eurasianos para capturar qualquer ancestralidade divergente não amostrada, enquanto um coiote foi usado como grupo externo para lobos norte-americanos. As barras denotam ±1 erro padrão estimado a partir de um canivete de bloco. c , F ST para pares de grupos amostrais com médias de datas separadas por ≤12.500 anos. As barras denotam ±1,96 erros padrão d, resultados de MSMC2 para pares de cromossomos X masculinos, com idades da amostra indicadas por linhas azuis. Um pico acentuado para cima na curva corresponde à divergência da população, com os tempos estimados indicados por linhas vermelhas. Curvas de exemplo para dois pares de lobos (esquerda e meio) e um resumo dos resultados para todos os pares (direita) são mostrados. kyr, mil anos.
Embora esses resultados demonstrem um fluxo gênico abrangente, eles também mostram que as substituições de ancestralidade foram incompletas e que frações minoritárias de ascendência européia profunda persistiram até os dias atuais (Fig. 2a,b ). A maioria dos lobos eurasianos modernos analisados provavelmente retém ascendência local do Pleistoceno, pois são melhor modelados pelo qpAdm como tendo 10-40% de ancestralidade que é mais divergente do que os lobos siberianos mais antigos neste estudo em ~ 100 ka (Figs. 11 e 12 complementares ). Além da ancestralidade local do lobo cinzento não representada entre nossos genomas antigos, isso pode incluir ancestralidade relacionada ao lobo dourado africano no Oriente Próximo e no sul da Ásia 22 e ancestralidade de origem canídeo desconhecida no Tibete 23 (Fig.10 ). Embora todos os lobos eurasianos hoje compartilhem a maioria de seus ancestrais nos últimos 25.000 anos, a persistência de ancestrais locais profundos fornece evidências contra a extinção local generalizada no final do Pleistoceno Eurásia e sugere que a espécie como um todo, ao contrário de muitas outras megafaunas, não veio perto da extinção.
Muitos lobos norte-americanos modernos e antigos mostram evidências de mistura de coiote ( Canis latrans ) 24 , 25 (Dados estendidos Fig. 1e ), o que explica por que alguns deles não se agrupam com lobos de idade semelhante no PCA (Fig. 1c ). Com base nas taxas de coalescência 26 entre cromossomos X masculinos, que têm fase de haplótipo perfeito, estimamos que lobos e coiotes começaram a divergir ~ 700 ka (Fig. 14 suplementar ), amplamente alinhado com uma divergência fóssil de ~ 1 milhão de anos atrás 27. Nossos dados mostram que a mistura de coiotes ocorreu pelo menos desde 100-80 ka, e dois lobos do Pleistoceno analisados do Yukon também carregavam linhagens mitocondriais de coiotes. Essas descobertas implicam que ou a faixa de coiotes do Pleistoceno se estendeu mais ao norte do que se pensava atualmente ou que a mistura ocorrendo mais ao sul se propagou para o norte através da população de lobos. Em nossos lobos euro-asiáticos, nenhum influxo de ancestralidade de coiotes é observado ao longo do tempo (Dados Estendidos Fig. 1e ). Encontramos um leve gradiente oeste-leste de crescente afinidade de coiotes entre os lobos da Eurásia, mas esse padrão provavelmente reflete a mistura em coiotes de lobos norte-americanos (que estão relacionados aos lobos no leste da Sibéria) (Fig. 9 suplementar ).
Depois de contabilizar a mistura de coiotes, descobrimos que a ascendência do lobo no Alasca e no Yukon estava altamente ligada à Sibéria ao longo do tempo (Fig. 2a ). Isso reflete a história do lobo europeu, mas, embora persista alguma ancestralidade europeia local profunda, nenhuma ancestralidade norte-americana profunda parece persistir até o presente. A ponte terrestre de Bering provavelmente permitiu um influxo de lobos siberianos no Alasca de forma intermitente entre 70 e 11 ka 28 , 29 , mas não encontramos evidências de fluxo gênico na outra direção. Todos os lobos norte-americanos atuais podem ser modelados como tendo 10 a 20% de ascendência de coiotes e a ascendência restante de lobos siberianos com menos de ~ 23 ka, sem contribuição de lobos norte-americanos anteriores (Fig. 2b). Descobrimos que os lobos vermelhos e algonquins se encaixam de maneira semelhante quando deslocados para coiotes ao longo dessa clinagem de mistura de duas fontes 11 , 25 , mas não podemos descartar maior complexidade em sua história. Embora os dados genômicos por si só não possam estabelecer a ausência de lobos cinzentos em nenhum momento específico, nossos resultados são consistentes com a extinção local na América do Norte, por exemplo, durante o LGM quando as camadas de gelo cobriram a metade norte do continente 30 , ou, alternativamente, uma ausência de lobos cinzentos ao sul das camadas de gelo até depois que o gelo recuou.
Alta conectividade no Pleistoceno
Para entender como as populações de lobos do passado eram diferenciadas, calculamos a proporção de variação genética entre grupos de lobos agrupados no espaço e no tempo, em vez de dentro (em pares F ST ; ref. 31 ). Antes do LGM, a diferenciação mesmo entre regiões distantes era baixa ( F ST < 3%) (Fig. 2c ). As primeiras populações européias e norte-americanas não eram muito diferentes umas das outras nem dos lobos relacionados à Sibéria que, com o tempo, substituíram grande parte de seus ancestrais. Também estimamos as taxas de coalescência do cromossomo X 26 , o que sugeriu que quaisquer dois lobos do Pleistoceno compartilhavam ancestralidade dentro de ~ 10.000 anos da data do lobo mais velho (Fig. 2de Fig. 15 Complementar ). O fluxo gênico generalizado impediu assim divergências profundas entre as populações de lobos no Pleistoceno Superior.
Nos últimos 10.000 anos (o Holoceno), a dinâmica populacional foi diferente daquela do Pleistoceno, sem evidência de fluxo gênico siberiano adicional para a Europa; em vez disso, os ancestrais relacionados aos europeus se espalharam para o leste e contribuíram para os lobos modernos na China e na Sibéria (Fig. 2b ). Níveis mais altos de diferenciação hoje ( F ST de ~ 10-60%) provavelmente refletem em grande parte gargalos populacionais após invasão de habitat e perseguição por humanos nos últimos séculos 32 , 33 , embora haja alguma evidência de crescente diferenciação já durante os últimos 20.000 anos (Fig. 2c). As estimativas de MSMC2 dos genomas atuais sugerem declínios generalizados no tamanho da população efetiva neste período (Fig. 13 suplementar ), mas não encontramos declínio simultâneo na heterozigosidade individual (Fig. 1d ). Combinadas, essas evidências sugerem que uma redução geral no fluxo gênico, como mostrado pelos resultados de F ST , em vez de um declínio populacional em toda a espécie 21 , pode ter resultado em tamanhos populacionais locais efetivos mais baixos.
Seleção natural ao longo de 100.000 anos
A forte conectividade observada entre os lobos do Pleistoceno tardio aumenta a possibilidade de adaptação em toda a espécie. A seleção natural é tipicamente inferida indiretamente da variação genética atual, mas nosso conjunto de dados de 100.000 anos (~ 30.000 gerações) permite a detecção direta de alelos selecionados. Testando cada variante para uma associação entre a frequência alélica e o tempo em 72 lobos antigos e 68 lobos modernos, e aplicando o controle genômico 34 para corrigir a variação da frequência alélica causada pela deriva genética, encontramos 24 regiões genômicas com evidências de seleção (Fig. 3a e Extended Dados Tabela 1 ). Confirmamos a robustez do nosso método à história demográfica aplicando-o a dados simulados na ausência de seleção, não encontrando falsos positivos (Fig.3b e Fig. 17 Suplementar ).
a , –log 10 ( valores P ) (bilateral, não ajustado para comparações múltiplas) da regressão linear para associação entre frequência alélica e idade da amostra. b , gráfico quantil-quantil comparando os valores de P em a com os esperados sob uma distribuição uniforme (topo) e da mesma forma para resultados de uma população simulada de evolução neutra (tamanho efetivo da população ( N e ) = 50.000) (abaixo). c , Observações de alelos em lobos antigos e frequências nas populações atuais para variantes de chumbo dos três picos mais fortes. As barras denotam CIs binomiais de 95%. d , valores P locais (de um) e TMRCA inferido usando Relate em lobos e cães modernos para a região ao redor do IFT88 . O histograma de todo o genoma (quantis em linhas cinzas) mostra que esse locus tem o TMRCA mais recente no genoma.
O sinal mais forte foi observado no cromossomo 25, onde as variantes que se sobrepõem ao gene IFT88 aumentaram rapidamente de cerca de 0% a 100% na frequência 40-30 ka e ainda são fixadas em lobos e cães hoje (Fig. 3c ). A inferência genealógica em lobos modernos 35 , 36 mostrou ainda que IFT88 teve o tempo mais jovem para o ancestral comum mais recente (TMRCA) (~ 70.000 anos) no genoma (Fig. 3d ). A interrupção do IFT88 leva a defeitos de desenvolvimento craniofacial em camundongos e a fissura labiopalatina em humanos 37 . Se futuros estudos fósseis revelarem uma rápida mudança craniodental neste período de tempo, isso poderia implicar o IFT88sweep como um driver, potencialmente em resposta a alterações de disponibilidade de presas. Mas também é possível que a seleção tenha como alvo traços não esqueléticos desconhecidos associados à variação do IFT88 . O segundo sinal mais forte no genoma foi de 2,5 Mb a jusante de IFT88 , onde as frequências alélicas mudaram em um período de tempo semelhante de 40 a 20 ka (Fig. 3c ), mas não está claro se essa região pode estar envolvida na regulação de longo alcance de IFT88 .
Três regiões com evidência de seleção sobrepõem genes de receptores olfativos, com variantes no cromossomo 15 aumentando em frequência de cerca de 0% a 100% 45-25 ka (Fig. 3c ), sugerindo que o olfato era um alvo recorrente de adaptação em lobos. A maioria dos episódios de seleção detectados ocorreu antes da divergência dos cães, e os cães compartilham os alelos selecionados (Fig. 18 suplementar ). No entanto, variantes em YME1L1 aumentaram em frequência de <5% para 50-70% em lobos de 20-0 ka, mas não são observadas em cães. Uma região no cromossomo 10, onde a variação entre os cães está associada ao tamanho do corpo, orelhas caídas e outras características 38 , 39 , 40 , estava sob seleção recente em raças específicas de cães41 , e descobrimos que também foi selecionado em lobos nos últimos 20.000 anos. Embora não tenha sido detectada em nossa varredura de seleção, a deleção de KB subjacente à pele preta 42 foi identificada em um lobo de 14.000 anos de Tumat, na Sibéria (Fig. 19 suplementar ). Essa deleção provavelmente introgrediu em lobos de cães no Holoceno 42 , mas nosso resultado também levanta a possibilidade de que sua origem final possa ter sido em lobos selvagens do Pleistoceno.
A ascendência canina tem afinidades com os lobos orientais
Descobrimos que os cães compartilham mais deriva genética com lobos que viveram após 28 ka do que com aqueles que viveram antes desse período, o que implica que os progenitores de cães foram geneticamente conectados a outros lobos pelo menos até 28 ka (Fig. 1c e Extended Data Fig. . 1b ). Uma divergência nessa época também é consistente com nossas análises MSMC2 de cromossomos X (Fig. 16 suplementar ). No entanto, até que a natureza do processo de divergência seja melhor compreendida, não se pode descartar que a domesticação tenha começado antes desse ponto.
A origem geográfica da atual linhagem canina Canis familiaris permaneceu controversa. Estudos genéticos têm argumentado que os lobos da Ásia Oriental 1 , 2 , Ásia Central 4 , Oriente Médio 6 , Europa 5 , Sibéria 16 , ou tanto a Eurásia oriental quanto a ocidental independentemente 3 , contribuíram com a ancestralidade dos cães primitivos, enquanto outros têm sido consistentes com uma população progenitora única, mas geograficamente desconhecida 8 , 9. Dada a nossa descoberta de que parte da estrutura da população de lobos é mais antiga do que o tempo provável de domesticação dos cães, podemos esperar que os cães sejam geneticamente mais próximos de alguns lobos antigos do que de outros. Para reduzir os efeitos do fluxo gênico desde o surgimento dos cães, realizamos uma PCA em lobos e cães dos últimos 25.000 anos, com base em estatísticas de f 4 quantificando suas relações apenas com lobos que vivem antes de 28 ka (ou seja, antes da LGM ), e descobriu que os cães mostraram perfis de relacionamento semelhantes aos dos lobos siberianos de 23-13 ka (Fig. 4a , Dados Estendidos Fig. 2 e Métodos ). Direto f 4-testes também mostraram que os cães estão mais próximos dos lobos siberianos do que dos lobos europeus deste período (Fig. 4b e Dados Estendidos Fig. 3 ). Os lobos europeus posteriores a 28 ka têm uma afinidade com os lobos europeus pré-LGM, refletindo a persistência da ancestralidade do lobo eurasiano do oeste profundo (Fig. 2a ). A ausência de tais afinidades ocidentais em cães sugere que eles não se originaram das populações de lobos europeus amostradas aqui.
a , PCA em lobos pós-LGM e atuais (X), com base em f 4 -estatísticas apenas da forma f 4 (X,A;B,C), onde A, B e C são qualquer um dos 21 lobos anteriores 28 k. Os cães são projetados e coloridos por f 4 (AndeanFox,X;Zhokhov dog 9,5 ka, Tel Hreiz dog 7,2 ka). b , Para cães (X), f 4 (AndeanFox,X;Zhokhov dog 9,5 ka,Tel Hreiz dog 7,2 ka) horizontalmente contra f 4 (AndeanFox,X;Belaya Gora wolf 18 ka,Hohle Fels wolf 13 ka) verticalmente (Pearson's r = 0,86, P = 3 × 10-23 ). As barras denotam ±1 erro padrão estimado a partir de um canivete de bloco. Silhuetas dephylopic.org . c , log 10 ( valores P ) para modelos qpAdm ajustados a alvos de cães, onde um valor P baixo significa que o modelo pode ser rejeitado. Um dhole antigo foi usado para representar ancestralidade divergente e não amostrada; modelos incluindo esta fonte têm contornos pretos. Os pontos são agitados horizontalmente para evitar a sobreposição. d , proporções de ascendência qpAdm para cães, usando o cão Zhokhov (9,5 ka) e um lobo sírio atual como proxies para ascendência de progenitores de cães orientais e ocidentais, respectivamente. As barras denotam ±1 erro padrão estimado a partir de um canivete de bloco. e , Mapa de cães precoces e posteriores relevantes e suas proporções de ancestralidade como em d. Cruzes pretas indicam a localização de lobos de 25 a 10 ka que podem ser rejeitados como progenitores de cães. Mapa base do pacote mapdata R. k, mil anos. f , Modelo gráfico de mistura das principais relações de linhagem de cães, ajustado por qpGraph sem estatísticas f atípicas. Os comprimentos das arestas estão em unidades de F ST (×1.000).
Enquanto os lobos siberianos do nordeste de 23 a 13 ka exibem a maior afinidade geral com os cães, descobrimos que eles não eram os ancestrais imediatos dos cães. Quando um amplo conjunto de lobos antigos foi testado como fontes candidatas usando qpWave/qpAdm 43 , todos os modelos de fonte única, incluindo um usando um lobo siberiano de 18.000 anos, foram fortemente rejeitados para todos os cães estudados ( P < 1 × 10 − 6 ) ( Métodos e Fig. 4c ). No entanto, um modelo com o lobo siberiano e 10-20% de ancestralidade de um componente aproximado pelo grupo externo dhole fit cães, como o indivíduo siberiano Zhokhov 17 de 9.500 anos de idade ( P = 0,29) (Fig. 4c). Embora use uma espécie de grupo externo, este modelo de duas fontes não implica necessariamente a mistura de duas populações ou espécies distintas. Em vez disso, pode refletir cães sendo derivados de alguma ancestralidade de lobo local que não é amostrada e até certo ponto divergente dos lobos antigos disponíveis (Dados Estendidos Fig. 4 ). Validando essa interpretação, descobrimos que os lobos europeus recentes, que têm um pequeno grau de ancestralidade europeia local e profunda (Fig. 2a ), obtêm resultados muito semelhantes aos dos cães, exigindo 10-20% de ancestralidade não amostrada, se apenas os lobos siberianos fossem disponíveis como fontes (Fig. 11 Suplementar e Informações Suplementares). Portanto, interpretamos os resultados para cães como refletindo de forma semelhante alguma ascendência de lobo não amostrada que não é totalmente representada pelos antigos lobos siberianos amostrados aqui. Esta ascendência não amostrada parece ter mantido um grau parcial de diferenciação dos lobos antigos amostrados desde antes de 100 ka (Fig. 12 suplementar ), e nossos resultados implicam que provavelmente viveu fora das regiões da Europa, nordeste da Sibéria e América do Norte amostradas aqui.
Os resultados obtidos para o cão Zhokhov também se aplicaram a cães antigos do Lago Baikal, América do Norte e nordeste da Europa (um cão careliano de 10.900 anos) e cães cantores modernos da Nova Guiné. como tendo se originado de um único 'fluxo' de diversidade de lobos antigos, em uma abordagem que não requer uma fonte próxima (Tabela de Dados Estendida 2 ). Esse resultado mostra que genomas de lobos antigos podem contornar as complexidades de processos mais recentes, pois os mesmos modelos foram rejeitados quando lobos modernos foram usados como fontes (Tabela de Dados Estendida 2 ), provavelmente devido ao fluxo gênico de cães para lobos 8 .
A mistura recente e as mudanças populacionais complicam as análises dos lobos modernos. Mesmo assim, se a estrutura populacional de lobos não foi completamente remodelada desde a época da domesticação do cão, é possível que parte da ancestralidade dos progenitores do cão ainda possa ser representada e detectável entre os lobos hoje, mesmo que a localização geográfica anterior dessa ancestralidade seria desconhecido. Testamos isso de duas maneiras. Primeiro, projetamos cães em um lote de PCA construído usando genótipos de lobos modernos e descobrimos que eles se projetavam mais perto dos lobos da China, Mongólia e Altai do que dos lobos da Yakutia (Dados Estendidos Fig. 5). Em segundo lugar, estendemos nossas análises de qpAdm para fontes modernas de lobos e descobrimos que alguns lobos chineses forneceram melhores ajustes do que o lobo siberiano de 18.000 anos e poderiam servir como fontes únicas de ancestralidade de cães Zhokhov sem a necessidade de um componente de ancestralidade não amostrado (Extended Dados Fig. 6 ). Esses resultados podem ser tomados para apoiar uma origem do cão eurasiano oriental ou central fora do nordeste da Sibéria, mas não podemos tirar conclusões geográficas firmes na ausência de genomas de lobos antigos dessas e de outras regiões candidatas.
Uma segunda fonte para ascendência de cães ocidentais
Estendemos nossas análises para um conjunto global de cães antigos e modernos, para testar quaisquer contribuições de ancestralidade de progenitores de lobos geneticamente distintos. A evidência mais forte de múltiplos progenitores seria se alguns cães tivessem afinidades diferentes com lobos anteriores à domesticação, pois esses lobos não podem ser afetados pelo fluxo gênico do cão. Aplicando esse raciocínio, descobrimos que os antigos cães do Oriente Próximo e os atuais africanos, e em menor grau os cães europeus, são deslocados para os lobos da Eurásia ocidental no PCA de estatísticas f 4 com base em relacionamentos com lobos anteriores ao LGM (Fig. 4a ). Este clino recapitula o eixo primário da estrutura populacional dentro dos cães (entre os antigos cães do Oriente Próximo e do leste da Eurásia 8 ) (Fig.4b ), mesmo quando os lobos dos últimos 28.000 anos são excluídos (Fig. 20 Suplementar ). A linhagem de ancestralidade do cão, portanto, pelo menos em parte, reflete as diferenças de ancestralidade do lobo que antecedem o provável período de domesticação. Testando as observações do PCA explicitamente, o qpWave rejeitou fortemente um único progenitor de lobo ao incluir cães do Oriente Próximo ( P < 10 -4 ) (Tabela de Dados Estendida 2 ). Os modelos qpAdm mais adequados para esses cães envolveram uma fonte relacionada aos antigos lobos europeus, além da ancestralidade encontrada no cão Zhokhov (Fig. 4c ).
Para testar se os lobos europeus antigos amostrados poderiam ser a fonte real desse segundo componente da ascendência canina, testamos modelos qpAdm apresentando o cão Zhokhov siberiano como uma fonte - representando a ascendência canina relacionada ao leste - e um antigo lobo europeu como um segundo fonte. Esses modelos não se encaixavam em cães do Oriente Próximo e africanos, a menos que um terceiro componente de grupo externo também fosse incluído para representar ancestralidade divergente e não amostrada (Fig. 21 suplementar ), o que significa que os lobos europeus não são páreo para a ancestralidade ausente. Expandindo para todos os lobos pós-LGM e atuais, apenas os lobos atuais da Síria, Israel, Irã e Índia alcançaram bons ajustes (Dados Estendidos Fig. 7). De acordo com uma fonte desta parte do mundo, quando projetados na estrutura do lobo atual, os cães do Oriente Próximo e da África são deslocados para os lobos caucasianos e do Oriente Próximo, em vez dos europeus (Dados estendidos Fig. 5 ). Usando um lobo sírio atual como fonte, estimamos 56% (erro padrão, 10%) ancestralidade de lobo relacionado ao Oriente Próximo no primeiro cão disponível (7,2 ka) do Levante, 37% (erro padrão, 3,5%) na raça Africana Basenji e 5-25% em cães neolíticos e europeus posteriores (Fig. 4d ). Embora a evidência de ancestralidade dupla seja baseada em lobos antigos que antecedem a domesticação e, portanto, não são afetados pelo potencial fluxo gênico posterior, essas estimativas exatas podem ser infladas se houver mistura de cães no lobo sírio.
Em seguida, testamos exaustivamente modelos de gráficos de mistura de relacionamentos de cães, permitindo até dois eventos de mistura entre quatro populações de cães e o lobo sírio. Obtivemos resultados consistentes com as inferências qpAdm, pois um único gráfico apresentando a mistura de lobo sírio em cães primitivos do Oriente Próximo se encaixa nos dados (Fig. 4f ), com uma linhagem de cães separada dando origem aos primeiros cães da Carélia e do Oriente. Neste gráfico, o cão da Carélia está mais intimamente relacionado com a fonte 'oriental' que também contribuiu com a ancestralidade do cão do Oriente Próximo.
As assimetrias de ancestralidade generalizadas observadas entre lobos e cães hoje têm sido interpretadas como reflexo da mistura local recente 8 , 9 . Nossa descoberta de que os cães têm proporções variáveis de dois componentes distintos da ancestralidade do lobo pode fornecer uma explicação unificadora para muitas dessas assimetrias. Por exemplo, estudos anteriores explicaram uma afinidade entre lobos siberianos do Pleistoceno e cães do Ártico, sugerindo a mistura nestes últimos 13 , 17 . O modelo de ancestralidade dupla provavelmente pode explicar essa assimetria sem tal mistura, com os cães do Ártico tendo menos do componente ocidental (Fig. 22 suplementar).). Por outro lado, níveis mais altos do componente ocidental em cães do Oriente Próximo e africanos provavelmente explicam pelo menos parte de sua afinidade observada anteriormente com os lobos do Oriente Próximo 8 , 9 , 10 . Uma observação de que os lobos em Xinjiang, na Ásia central, não apresentam assimetrias para diferentes cães foi interpretada como sugerindo que outras assimetrias são principalmente devido ao fluxo gênico cão-lobo 8 . Nossos resultados, em vez disso, sugerem que um equilíbrio de ancestrais de lobos orientais e ocidentais na Ásia central (Fig. 2b ) causa uma relativa simetria para os ancestrais de cães orientais e ocidentais. Os lobos de Xinjiang, portanto, não são evidências contra o modelo de ancestralidade dupla.
Conclusão
Mostramos que as populações de lobos foram geneticamente conectadas ao longo do Pleistoceno Superior, provavelmente devido à alta mobilidade dos lobos em uma paisagem aberta 44 . O LGM não correspondeu necessariamente a um tempo de mudança sem precedentes para a população interconectada de lobos, o que pode fornecer uma pista de sua perseverança quando outros carnívoros do norte da Eurásia foram extintos. Além disso, a razão pela qual os lobos do Pleistoceno parecem basais para a diversidade atual não é que eles foram extintos 13 , 14, mas esse fluxo gênico contínuo homogeneizou a ancestralidade posterior. Nossa descoberta de que vários alelos selecionados rapidamente atingiram a fixação mostra que as adaptações se espalharam para toda a população de lobos do Pleistoceno, um processo que pode ter contribuído para a sobrevivência da espécie. Ao mesmo tempo, nossos resultados mostram que essas varreduras seletivas rápidas em toda a espécie ocorreram apenas algumas vezes nos últimos 100.000 anos.
Nossos resultados também fornecem insights sobre questões de longa data sobre a origem dos cães. Primeiro, acredita-se que cães e lobos eurasianos atuais sejam linhagens monofiléticas recíprocas 9 . Descobrimos que, em geral, os cães estão mais próximos dos lobos da Eurásia oriental. Em segundo lugar, porque nenhum lobo moderno é uma boa combinação para ancestralidade de cães, a população de origem foi considerada extinta. Nossos resultados implicam que este não é necessariamente o caso, já que a homogeneização contínua da ancestralidade do lobo pode ter obscurecido as relações anteriores com os cães. Terceiro, não está claro se mais de uma população de lobos contribuiu para os cães primitivos e atuais 3 , 7 , 8 , 9. Descobrimos que uma fonte relacionada ao leste da Eurásia, 'progenitor do cão oriental', parece ter contribuído com ~ 100% da ascendência dos primeiros cães na Sibéria, nas Américas, no leste da Ásia e no nordeste da Europa. Além disso, uma fonte relacionada à Eurásia ocidental, 'progenitor do cão ocidental', contribuiu com 20 a 60% da ascendência dos primeiros cães do Oriente Próximo e da África e 5 a 25% da ascendência dos cães neolíticos e europeus posteriores. A ancestralidade ocidental posteriormente se espalhou pelo mundo com, por exemplo, a expansão pré-histórica da agricultura na Eurásia ocidental 8 e a expansão dos cães europeus na era colonial.
Um estudo anterior propôs que o aparecimento arqueológico anterior de cães na Eurásia ocidental e oriental do que na Eurásia central foi devido à domesticação independente de lobos ocidentais e orientais, mas que a ancestralidade do primeiro foi extinta ou quase extinta nos cães atuais 3 . Nossos resultados apoiam a noção de dois ancestrais distintos de cães, mas diferem dessa hipótese anterior. Primeiro, demonstramos que a ascendência de pelo menos duas populações de lobos existe e é onipresente em cães modernos, e é o principal determinante da estrutura populacional de cães hoje. Em segundo lugar, somos capazes de rejeitar os lobos europeus do Pleistoceno relacionados aos aqui amostrados como fonte para o C. familiarislinhagem. Terceiro, o estudo anterior sugeriu que um cão neolítico irlandês tinha mais ascendência da domesticação ocidental do que cães posteriores 3 , enquanto descobrimos que esse cão tinha menos ascendência do progenitor ocidental identificado aqui do que os cães europeus atuais (Fig. 4d ). A falta de genomas dos primeiros cães da Europa, no entanto, significa que estudos futuros podem revelar que eles surgiram de um processo de domesticação independente que não contribuiu substancialmente para populações posteriores 3 , 45 , 46 .
Nossos resultados são consistentes com dois cenários: (1) domesticação independente dos progenitores orientais e ocidentais que mais tarde se fundiram no oeste ou (2) domesticação única do progenitor oriental, seguida pela mistura de lobos ocidentais quando os cães chegaram ao sudoeste da Eurásia. Nossos resultados não podem distinguir entre esses cenários, mas, em ambos os casos, a fusão ou mistura deve ter ocorrido antes de 7,2 ka, a idade do mais antigo genoma de cão do Oriente Próximo disponível 8 . Uma única domesticação do progenitor ocidental seguida pela mistura de lobos orientais não parece compatível com nossos resultados, pois exigiria a substituição de 100% da ancestralidade dos cães orientais. Se cães com 100% de ancestralidade de progenitores ocidentais fossem descobertos, por exemplo, no antigo Oriente Próximo 47ou contextos europeus 15 , isso implicaria domesticação independente. Alternativamente, os primeiros cães no oeste poderiam ser de ascendência progenitora oriental, semelhante ao cão careliano de 10,9 ka, em linha com um único processo de domesticação. Genomas de lobos antigos adicionais, inclusive de fora das regiões cobertas aqui, onde o DNA geralmente preserva menos bem, também serão necessários para identificar ainda mais os progenitores de lobos de cães.
Métodos
Amostragem, preparação de DNA e sequenciamento
Estocolmo
As amostras LOW002, LOW003, LOW006, LOW007, LOW008 e PON012 foram processadas no Laboratório de Pesquisa Arqueológica da Universidade de Estocolmo, Suécia, seguindo métodos previamente descritos 8 . Em resumo, isso envolveu a extração de DNA incubando o pó de osso por 24 h a 37 ° C em 1,5 ml de tampão de digestão (0,45 M EDTA (pH 8,0) e 0,25 mg ml -1 proteinase K), concentrando o sobrenadante em Amicon Ultra-4 (30 kDa peso molecular cut-off (MWCO)) colunas de filtro (MerckMillipore) e purificação em colunas Qiagen MinElute. As bibliotecas Illumina de fita dupla foram preparadas usando o protocolo descrito na ref. 48 , com a inclusão da enzima USER e as modificações descritas na ref. 49 .
As amostras 367, PDM100, Taimyr-1 e Yana-1 foram processadas no Museu Sueco de História Natural em Estocolmo, Suécia, seguindo métodos previamente descritos 8 . Em resumo, isso envolveu a extração de DNA usando um método à base de sílica com concentração em filtros Vivaspin (Sartorius), de acordo com um protocolo otimizado para recuperação de DNA antigo 50 . As bibliotecas Illumina de fita dupla foram preparadas usando o protocolo descrito na ref. 48 , com a inclusão da enzima USER.
As amostras ALAS_024, VAL_033, ALAS_016, VAL_008, HMNH_007, HMNH_011, VAL_050, VAL_005, DS04, VAL_037, VAL_012, VAL_011, VAL_18A, IN18_016 e IN18_005 foram processadas no Museu Sueco de História Natural em Estocolmo, Suécia, seguindo métodos descritos anteriormente para permafrost amostras de osso e dente 51 . Em resumo, isso envolveu extração de DNA usando a metodologia da ref. 52 e preparação de biblioteca de dupla fita Illumina como descrito na ref. 48, com índices únicos duplos e a inclusão da enzima USER. Entre oito e dez reações de PCR separadas com índices únicos foram realizadas para cada amostra para maximizar a complexidade da biblioteca. As bibliotecas foram sequenciadas ao lado das amostras HOV4, AL2242, AL2370, AL2893, AL3272 e AL3284 em três pistas Illumina NovaSeq 6000 com uma configuração de extremidade pareada S4 de 100 pb no SciLifeLab em Estocolmo.
Potsdam
As amostras JAL48, JAL65, JAL69, JAL358, AH574, AH575 e AH577 foram processadas na Universidade de Potsdam. As etapas de pré-amplificação (extração de DNA e preparação da biblioteca) foram realizadas em salas de laboratório separadas, especialmente equipadas para o processamento de DNA antigo. As etapas de amplificação e pós-amplificação foram realizadas em diferentes salas de laboratório. O DNA foi extraído do pó de osso (29–54 mg) seguindo um protocolo especialmente adaptado para recuperar fragmentos curtos de DNA 52 . Bibliotecas duplamente indexadas de fita simples foram construídas a partir de 20 µl de extrato de DNA de acordo com o protocolo na ref. 53 . As bibliotecas foram sequenciadas em uma plataforma HiSeq X no SciLifeLab em Estocolmo.
Tübingen/Jena
As amostras JK2174, JK2175, JK2179, JK2181, JK2183, TU144, TU148, TU839 e TU840 foram processadas na Universidade de Tübingen, com extração de DNA e etapas de pré-amplificação realizadas em instalações de sala limpa e etapas de pós-amplificação realizadas em um laboratório de DNA separado . Ambos os laboratórios cumprem as normas para trabalhar com DNA antigo 54 , 55 . Todas as superfícies das amostras de dentes e ossos foram inicialmente irradiadas com UV por 30 min, para minimizar o risco potencial de contaminação do DNA moderno. Posteriormente, o DNA foi extraído aplicando-se um protocolo baseado em sílica de guanidina bem estabelecido para amostras antigas 52 . As bibliotecas de sequenciamento Illumina foram preparadas usando 20 µl de extrato de DNA por biblioteca 48; posteriormente, códigos de barras duplos (índices) foram adicionados quimicamente às extremidades primárias das bibliotecas 56 . Para as amostras de Auneau (TU839 e TU840), foram preparadas cinco bibliotecas de sequenciamento cada; para todas as outras amostras processadas em Tübingen, três bibliotecas de sequenciamento foram preparadas cada. Para detectar a contaminação potencial dos produtos químicos, foram realizados controles negativos para extração e preparação da biblioteca. Após a preparação das bibliotecas de sequenciamento, a concentração de DNA foi medida com qPCR (Roche LightCycler) usando primers correspondentes 48 . A concentração de DNA foi dada pelo número de cópias dos fragmentos de DNA em 1 µl da amostra.
A amplificação das bibliotecas de sequenciamento indexadas foi realizada usando Herculase II Fusion sob as seguintes condições: 1x tampão Herculase II, primer IS5 0,4 µM e primer IS6 0,4 µM 48, Herculase II Fusion DNA polimerase (Agilent Technologies), 0,25 mM dNTPs (100 mM; 25 mM cada dNTP) e 0,5–4 µl de biblioteca com código de barras como modelo em um volume total de reação de 100 µl. O perfil térmico de amplificação aplicado foi processado da seguinte forma: desnaturação inicial por 2 min a 95 °C; desnaturação por 30 s a 95 °C, anelamento por 30 s a 60 °C e alongamento por 30 s a 72 °C por 3 a 20 ciclos; e uma etapa final de alongamento por 5 min a 72°C. Em seguida, o DNA amplificado foi purificado usando uma etapa de purificação MinElute e o DNA foi eluído em 20 µl de TET. A concentração das bibliotecas de sequenciamento de DNA amplificado foi medida usando um Bioanalyzer (Agilent Technologies) e um chip de laboratório DNA1000 da Agilent Technologies.
As bibliotecas de sequenciamento foram sequenciadas em uma plataforma Illumina HiSeq 4000 no Max Planck Institute for Science of Human History em Jena. As amostras de Auneau (TU839 e TU840) foram sequenciadas em pares aplicando 2 × 50 + 8 + 8 ciclos. Todas as outras bibliotecas preparadas em Tübingen foram sequenciadas de extremidade única usando 75 + 8 + 8 ciclos.
Oxford
As amostras AL2657, AL2541, AL2741, AL2744, AL3185, AL2350, CH1109, AL2370, AL3272 e AL3284 foram processadas na instalação dedicada de DNA antigo no laboratório PalaeoBARN da Universidade de Oxford, seguindo métodos descritos anteriormente 8 . Em resumo, as bibliotecas de fita dupla foram construídas seguindo o protocolo na ref. 48. Essas bibliotecas foram sequenciadas em um instrumento HiSeq 2500 (AL2657, AL2541, AL2741, AL2744) ou HiSeq 4000 (AL3185, AL2350, CH1109) no Centro Nacional de Seqüenciamento Dinamarquês ou em um instrumento NextSeq 550 (AL2741) no Museu de História Natural de Londres. Para as amostras AL2370, AL3272 e AL3284, entre seis e oito reações de PCR separadas com índices únicos foram realizadas em suas bibliotecas e foram sequenciadas juntamente com as amostras HOV4, VAL_18A e IN18_016 em uma via Illumina NovaSeq 6000 com uma extremidade pareada S4 100-bp instalação no SciLifeLab em Estocolmo.
Copenhague
As amostras CGG13, CGG17, CGG19, CGG20, CGG21, CGG25, CGG26, CGG27, CGG28, CGG34, Tumat1 e IRK foram processadas no GLOBE Institute, University of Copenhagen. Todo o trabalho pré-PCR foi realizado em antigas instalações de DNA seguindo as diretrizes antigas de DNA 57 . Os detalhes da extração, construção da biblioteca e sequenciamento para as amostras com códigos CGG estão descritos na ref. 21 , em relação à publicação de dados mitocondriais desses espécimes. A amostra Tumat1 foi processada seguindo exatamente o mesmo protocolo. Em resumo, a extração de DNA foi realizada usando um tampão contendo uréia, EDTA e proteinase K 50 , bibliotecas de fita dupla foram preparadas com NEBNext DNA Sample Prep Master MixSet 2 (E6070S, New England Biolabs) e adaptadores específicos para Illumina 48, e o sequenciamento foi realizado em uma plataforma Illumina HiSeq 2500 usando química de leitura única de 100 pb. Para a amostra IRK, o DNA foi extraído de três subamostras e purificado conforme descrito na ref. 21 . Os três extratos de DNA e a pré-digestão purificada de uma subamostra foram incorporados em bibliotecas de fita dupla seguindo o protocolo BEST 58 , com as modificações descritas na ref. 59 e sequenciado em uma plataforma BGISEQ-500 usando química de leitura única de 100 pb.
Santa Cruz
As amostras SC19.MCJ017, SC19.MCJ015, SC19.MCJ010 e SC19.MCJ014 foram processadas no Laboratório de Paleogenômica da UCSC e foram fornecidas pelo programa de Paleontologia do Governo de Yukon. Todo o trabalho pré-PCR foi realizado em uma instalação dedicada de DNA antigo na Universidade da Califórnia, Santa Cruz, seguindo métodos padrão de DNA antigo 60 . Subamostras (250–350 mg) foram enviadas para a instalação UCI KECK AMS para datação por radiocarbono, e as quantidades restantes foram pulverizadas em um Retsch MM400 para extração. Para cada amostra, ~100 mg de pó foi tratado com uma solução de hipoclorito de sódio a 0,5% antes da extração para remover contaminantes de superfície 61 e depois combinado com 1 ml de tampão de lise para extração, seguindo o protocolo na ref. 52. As amostras foram processadas em paralelo com um controle negativo. Nós quantificamos os extratos usando um kit Qubit 1× dsDNA HS Assay (Q33231) antes de preparar as bibliotecas. Preparamos bibliotecas de fita simples seguindo o protocolo na ref. 62 e amplificou as bibliotecas por 9-16 ciclos conforme informado por qPCR. Após a amplificação, limpamos as bibliotecas usando uma solução de esferas 1,2 × SPRI e as agrupamos em uma razão equimolar para sequenciamento de controle de qualidade raso interno em uma execução de 75 pb de extremidade pareada NextSeq 550. Em seguida, enviamos as bibliotecas para a Fulgent Genetics para um sequenciamento mais profundo em duas pistas de 150 bp de extremidade pareada em um instrumento HiSeq X.
Viena
A amostra HOV4 foi processada no Departamento de Antropologia da Universidade de Viena. A amostra é um dente canino, que após o sequenciamento foi determinado para derivar de um dhole ( Cuon alpinus ). O DNA foi extraído de seu cemento pelos métodos descritos na ref. 63 com um tempo de incubação modificado de ~18 h. A biblioteca foi preparada de acordo com o protocolo na ref. 48 com as modificações da ref. 64 . Cinco reações de PCR separadas com índices exclusivos foram realizadas na biblioteca e foram sequenciadas ao lado das amostras VAL_18A, IN18_016, AL2242, AL2370, AL2893, AL3272 e AL3284 em uma pista Illumina NovaSeq 6000 com uma configuração de extremidade pareada S4 de 100 bp em SciLifeLab em Estocolmo.
Uma visão geral de todas as amostras e seus metadados associados está disponível em Dados Suplementares 1 .
Processamento de dados de sequência de genoma
Para dados de extremidade emparelhada, os pares de leitura foram mesclados e os adaptadores foram aparados usando SeqPrep ( https://github.com/jstjohn/SeqPrep ), descartando leituras que não puderam ser mescladas com êxito. As leituras foram mapeadas para o genoma de referência canFam3.1 do cão usando BWA aln (v.0.7.17) 65 com parâmetros permissivos, incluindo uma semente desabilitada (-l 16500 -n 0,01 -o 2). As duplicatas foram removidas mantendo apenas uma leitura de qualquer conjunto de leituras que tivessem a mesma orientação, comprimento e coordenadas inicial e final. Para a amostra Taimyr-1, os dados 13 publicados anteriormente foram mesclados com os dados recém-gerados. Os dados das amostras processadas em Copenhague foram processados conforme descrito anteriormente 66exceto que eles também foram mapeados para canFam3.1. O dano post-mortem foi quantificado usando PMDtools (v0.60) 67 com os argumentos '--first' e '--CpG'.
Genotipagem e integração com genomas publicados anteriormente
Para construir um conjunto de dados comparativos para análises genéticas populacionais, partimos de um conjunto de chamadas variante publicado compilando 722 genomas modernos de cães, lobos e outros canídeos de vários estudos anteriores (NCBI BioProject acesso PRJNA448733 ) 40 . Para isso, adicionamos genomas inteiros modernos adicionais de outros estudos: 4 lobos dourados africanos e 15 cães da aldeia nigeriana (Genome Sequence Archive ( http://gsa.big.ac.cn/ ), acesso PRJCA000335) 68 , 12 lobos escandinavos ( Acesso ao Arquivo Europeu de Nucleotídeos PRJEB20635 ) 69 , 9 Lobos e coiotes norte-americanos (acesso ao Arquivo Europeu de Nucleotídeos PRJNA496590 ) 25e 8 outros canídeos (cão de caça africano, dhole, lobo etíope, chacal dourado, lobos cinzentos do Oriente Médio) (Acesso do Arquivo de Nucleotídeos Europeus PRJNA494815 ) 22 . As leituras desses genomas foram mapeadas para o genoma de referência do cão usando bwa mem (versão 0.7.15) 70 , marcado para duplicatas usando Picard Tools (v2.21.4) ( http://broadinstitute.github.io/picard ), genotipado no sites presentes no conjunto de dados acima usando GATK HaplotypeCaller (v3.6) 71 com o argumento '-gt_mode GENOTYPE_GIVEN_ALLELES' e, em seguida, mesclados no conjunto de dados usando bcftools merge ( http://www.htslib.org/). Os seguintes filtros foram então aplicados a locais e genótipos em todo o conjunto de dados: locais com excesso de heterozigosidade (bcftools fill-tags 'ExcHet' valor P < 1 × 10 −6) foram removidos; os alelos indel foram removidos definindo o genótipo de qualquer indivíduo portador de tal alelo como ausente; genótipos em locais com uma profundidade (tomada como a soma dos campos 'AD' VCF) inferior a um terço ou mais do que o dobro da média do genoma para o genoma dado ou inferior a 5 foram definidos como ausentes; genótipos contendo qualquer alelo diferente dos dois alelos de maior frequência no local foram definidos como ausentes; a representação dos alelos foi normalizada usando a norma bcftools; e, finalmente, os locais em que 130 ou mais indivíduos tinham um genótipo ausente foram removidos. Isso resultou em um conjunto de dados final de 67,8 milhões de SNPs bialélicos. Nas análises de ancestralidade (ou seja, aquelas envolvendo f-estatísticas), os lobos modernos foram tratados como indivíduos enquanto que para cães modernos até quatro indivíduos com a maior cobertura de sequenciamento de uma determinada raça foram usados e combinados em populações. Uma lista dos genomas modernos usados em análises e seus metadados associados está incluída em Dados Suplementares 2 .
Todos os genomas antigos receberam genótipos pseudo-haplóides nos locais variantes no conjunto de dados acima usando htsbox pileup r345 ( https://github.com/lh3/htsbox ), exigindo um comprimento mínimo de leitura de 35 bp ('-l 35') , qualidade de mapeamento de 20 ('-q 20') e qualidade de base de 30 ('-Q 30'). Se um genoma antigo carregasse um alelo não presente no conjunto de dados, seu genótipo era definido como ausente. Os genomas antigos e históricos de lobos e cães gerados anteriormente mapeados para a referência do cão foram obtidos das respectivas publicações 3 , 7 , 8 , 13 , 17 , 66 , 72 , 73 (European Nucleotide Archive study accessions PRJEB7788, PRJEB13070 , PRJNA319283 , PRJEB22026 , PRJNA608847 , PRJEB38079 , PRJEB39580 , PRJEB41490 ) e genotipados da mesma maneira. Uma lista dos genomas antigos usados nas análises e seus metadados associados está incluída em Dados Suplementares 2 .
Análise filogenética do genoma mitocondrial e datação evolutiva
Extraímos reads mapeados para o genoma mitocondrial para as amostras de lobos antigos usando samtools (v1.9) 74 . Chamamos sequências de consenso usando um limite de 75%, chamando quaisquer sites com cobertura inferior a 3 como 'N', usando Geneious (v9.0.5) e removemos quaisquer amostras com mais de 10% de dados ausentes. Incluímos um conjunto de genomas mitocondriais publicados anteriormente de lobos antigos e modernos 5 , 9 , 13 , 21 , 75 , 76 , 77 , 78 , 79 , 80 , o que levou a um conjunto de dados final de 183 indivíduos (62 14Indivíduos antigos datados em C, 24 indivíduos antigos não datados, dos quais 7 tinham infinitas datas de 14 C e 90 indivíduos modernos). Também incluímos três sequências semelhantes a coiotes como grupos externos (de um coiote moderno e dois lobos antigos com sequências mitocondriais semelhantes a coiotes: SC19.MCJ015, datado de 14 C e SC19.MCJ017, com data infinita de 14 C). Alinhamos todas as sequências usando Clustal Omega (v1.2.4) 81 . Uma filogenia Bayesiana foi construída usando BEAST (v1.10.1) 82 , com um modelo de substituição HKY + I + G escolhido por JModelTest2 (v2.1.10) 83, relógio log-normal relaxado não correlacionado e árvore de tamanho constante coalescente anterior. Combinamos 20 cadeias MCMC (cada uma executada por 200 milhões de iterações), após excluir os primeiros 25% dos valores como burn-in. Para amostras com data de 14 C, incluímos datas de ponta anteriores que correspondiam a uma distribuição normal com a mesma média e distribuição de confiança de 95% das datas de 14 C. Estimamos as idades de amostras sem data a partir de uma distribuição a priori da seguinte forma: (1) para n = 24 amostras antigas sem informação de 14 C, usamos uma anterior uniforme de 0 a 1.000.000 anos antes do presente ( BP ); (2) para o n = 7 amostras antigas com infinitos 14C, usamos uma anterior uniforme como em (1), mas com o limite inferior como a data mínima dada pela datação por radiocarbono; (3) todas as n = 90 amostras modernas já haviam sido publicadas anteriormente 21 , e as datas anteriores da ponta para essas amostras eram as mesmas que as anteriores uniformes usadas no estudo anterior (0 a 100 ou 0 a 500 BP ). As sequências de consenso mitocondrial para as amostras de lobo recentemente relatadas aqui (excluindo aquelas que foram removidas porque tinham muitos dados ausentes) estão disponíveis como Dados Suplementares 4 .
f -estatísticas e gráficos de mistura
As estatísticas f 3 - ef 4 - foram calculadas com ADMIXTOOLS (v5.0) 84 , usando apenas sites de transversão e com o argumento 'numchrom: 38'. Para superar as limitações de memória ao calcular um grande número de estatísticas de f 4 , o jackknifing de bloco foi executado externamente ao ADMIXTOOLS em 225 blocos de 10 Mb de tamanho. Gráficos de mistura foram ajustados usando qpGraph, com argumentos 'outpop: NULL', 'useallsnps: NO', 'blgsize: 0.05', 'forcezmode: YES', 'lsqmode: NO', 'diag: 0.0001', 'bigiter: 6' , 'contrata: SIM' e 'lambdascale: 1'. As estatísticas do grupo externo f 3 foram calculadas usando apenas os locais verificados como heterozigotos no indivíduo CoyoteCalifornia.
A PCA foi realizada nas estatísticas f 3 do grupo externo, transformando os valores em distâncias tomando 1 – f 3 e, em seguida, executando a função prcomp R na matriz de distância resultante. Apenas lobos antigos foram incluídos no cálculo dos PCs; lobos atuais e cães antigos e atuais foram então projetados individualmente nos PCs re-executando a análise uma vez para cada um desses indivíduos independentemente com aquele único indivíduo adicionado e salvando suas coordenadas. Para evitar sobrecarregar a parcela com cães, apenas os seguintes cães foram incluídos: Basenji, Boxer, BullTerrier, NewGuineaSingingDog, SiberianHusky, Germany.HXH (7.000 BP ), Germany.CTC (4,7 ka), Ireland.Newgrange (4.800 BP ), Israel .THRZ02 (7.200 PB ), Baikal.OL4223 (6.900 PB ), Zhokhov.CGG6 (9.500 PB ) e PortauChoix.AL3194 (4.000 PB ).
Heterozigosidade e estimativas de F ST
A heterozigosidade condicional foi estimada em 1.250.173 locais de transversão verificados como heterozigotos no indivíduo CoyoteCalifornia, escolhido porque é em grande parte um grupo externo à diversidade do lobo. Para cada indivíduo, exatamente duas leituras foram amostradas em cada um desses locais (se disponíveis), e a fração de locais onde essas duas leituras exibiram alelos diferentes foi calculada (alelos diferentes dos observados no coiote foram ignorados). Os erros padrão foram obtidos por jackknifing de blocos através dos 38 cromossomos.
F ST foi calculado com smartpca do pacote EIGENSOFT (v7.2.1) 85 , usando a opção 'inbreed: YES' para contabilizar os genótipos pseudohaplóides dos genomas antigos (esta opção também foi aplicada aos genomas diplóides atuais). F ST foi calculado aos pares para pools de pelo menos dois genomas, formados a partir de indivíduos selecionados por estarem próximos no tempo e no espaço (Tabela Complementar 1). Alguns pares de indivíduos mostraram alta similaridade indicando possível parentesco, conforme avaliado comparando as taxas de incompatibilidade de leitura entre indivíduos e um indivíduo de cada um desses pares foi excluído dessas análises (JK2174 foi excluído devido à alta semelhança com JK2183, TU839 porque de alta similaridade com TU840, e CGG17 por causa de alta similaridade com Yana-1). Os valores de F ST para pares de pools com pontos médios de idade separados por menos de 12.500 anos foram incluídos no gráfico.
Análises de tempo de divergência e tamanho efetivo da população com MSMC2
Usamos MSMC2 (v2.1.2) 26 para inferir tempos de divergência populacional e históricos de tamanho efetivo da população. Os genótipos de entrada para isso foram chamados usando GATK HaplotypeCaller (v3.6) 86 em genomas antigos e modernos com cobertura de sequenciamento >5,8×. Para análises de tempo de divergência, os cromossomos X haploides de dois genomas masculinos diferentes foram combinados e o ponto em que o tamanho efetivo da população inferida para este cromossomo 'pseudodiplóide' aumentou acentuadamente foi considerado como correspondendo a uma divergência populacional. Os resultados foram escalonados usando uma taxa de mutação de 0,4 × 10 −8 mutações por local por geração 13 , 87 (com uma taxa 25% menor para análises de cromossomo X) e um intervalo geracional médio de 3 anos 13. Para inferências de tamanho efetivo da população, as variantes de transição foram ignoradas e os resultados foram dimensionados usando uma taxa de mutação apenas de transversões inferida a partir de resultados em genomas modernos. Para obter mais detalhes sobre as análises do MSMC2, consulte a seção 3 de Informações Complementares .
Análises de seleção
A análise de seleção foi realizada usando PLINK (v1.90b5.2) 88 . Esta análise usou os 72 genomas de lobos antigos e 68 genomas de lobos modernos (com o último incluindo um genoma de lobo japonês histórico 73 tratado como antigo para fins de análise, com idade definida para 200 PB ). Uma lista dos genomas usados para esta análise está disponível em Dados Suplementares 2 (coluna "Usado para varredura de seleção"). Todos os SNPs, não apenas as transversões, foram usados para esta análise. A idade de cada lobo foi definida como o fenótipo, com valores de 0 para lobos modernos, e o argumento '--linear' foi usado para testar uma associação entre genótipos SNP e idade, aplicando também o argumento '--adjust' para P corretovalores usando controle genômico. A aplicação do controle genômico 34 aqui teve como objetivo usar a magnitude da variação de frequência do alelo temporal observada em todo o genoma para explicar o que foi observado apenas a partir da deriva genética, dada a história demográfica do lobo. Apenas os resultados para os seguintes conjuntos de sítios foram retidos e incluídos no gráfico de Manhattan: sítios onde pelo menos 40 genomas antigos tinham uma chamada de genótipo, sítios com uma frequência alélica menor entre os lobos antigos de ≥5% e sítios que tinham pelo menos 7 sites vizinhos dentro de uma janela de 50 kb com um valor P que era pelo menos 90% tão grande (em uma escala log 10 ) quanto o Pvalor do próprio site. O último 'filtro de vizinhança' visava reduzir falsos positivos, exigindo evidências semelhantes em vários locais próximos. Como ponto de corte de significância do valor P para corrigir o teste genômico, usamos 5 × 10 −8 , que é comumente usado em estudos de associação genômica em humanos e também em cães 89 . Excluímos 15 regiões onde apenas uma única variante atingiu significância. Uma tabela detalhada com as 24 regiões detectadas está disponível em Dados Complementares 3 . Para testar a robustez desta análise para falsos positivos decorrentes apenas da deriva genética, aplicamos a mesma análise a dados de simulações coalescentes neutras geradas usando ms 90e não encontrou nenhum falso positivo. Para obter mais detalhes, consulte a seção 4 de Informações Complementares .
Modelagem de ancestralidade com qpAdm e qpWave
Usamos os métodos qpAdm e qpWave 43 de ADMIXTOOLS (v5.0) 84 para testar modelos de ancestralidade para lobos e cães alvos após 23 ka. Para as análises primárias, usamos o seguinte conjunto de populações-fonte candidatas (estimativa de idade entre parênteses, anos PB): Armenia_Hovk1.HOV4 (ancient dhole), Siberia_UlakhanSular.LOW008 (70,772), Germany_Aufhausener.AH575 (57,233), Siberia_BungeToll.CGG29 (48,210), Germany_HohleFels.JK2183 (32,366), Siberia_BelayaGora.IN18_016 (32,020), Yukon_QuartzCreek.SC19.MCJ010 (29.943), Altai_Razboinichya.AL2744 (28.345), Siberia_BelayaGora.IN18_005 (18.148) e Germany_HohleFels.JK2179 (13.229). Usamos uma abordagem rotativa na qual, para cada alvo, testamos todos os modelos possíveis de uma, duas e três fontes que poderiam ser enumerados a partir do conjunto acima. Indivíduos do conjunto que não foram usados como fonte em um determinado modelo serviram como conjunto de referência (ou a população 'correta' no framework qpAdm). Isso significa que, em todos os modelos, cada um dos indivíduos acima estava sempre na lista de fontes ou na lista de referências. Classificamos os modelos com base em suasValores de P , mas priorizando modelos com menos fontes usando um limiar de valor- P de 0,01: se um modelo mais simples (ou seja, um modelo com menos fontes) tivesse um valor de P acima desse limiar, ele seria classificado acima de um modelo mais complexo (ou seja, um modelo com mais fontes) independentemente do Pvalor deste último. Também falhamos em modelos com proporções de ancestralidade inferidas maiores que 1,1 ou menores que -0,1. Para modelos de fonte única, o qpWave foi executado em vez do qpAdm. Ambos os programas foram executados com a opção 'allsnps: YES' (sem esta opção, havia muito pouco poder para rejeitar modelos). Descrevemos a ancestralidade atribuída à antiga fonte dhole (Armenia_Hovk1.HOV4) como ancestral 'não amostrada'; observe que isso não implica que tal ancestralidade seja de origem não-lobo, apenas que não é representada por (ou seja, divergiu cedo e não tem deriva genética compartilhada com) os genomas de lobos antigos no conjunto de referência.
Para testar se qualquer genoma de lobo pós-23 ka ou moderno disponível pode ser um bom substituto para a ancestralidade relacionada ao lobo da Eurásia ocidental identificada em cães do Oriente Próximo e da África, adicionamos o cão Zhokhov 17 de 9.500 anos ao conjunto rotativo de populações-fonte candidatas. Escolhido por sua alta cobertura, data inicial e localização oriental, isso pressupõe que o cão Zhokhov seja um bom representante para o componente de ancestralidade canina oriental. Usando o cão africano Basenji como alvo, os modelos envolvendo o cão Zhokhov mais outro lobo nos permitiram testar se aquele lobo era uma boa combinação para o componente adicional de ancestralidade. Para obter mais detalhes sobre as análises qpAdm e qpWave, consulte as seções 2 de Informações Complementares (alvos de lobo) e 5 (alvos de cães).
Resumo do relatório
Mais informações sobre o projeto de pesquisa estão disponíveis no Nature Research Reporting Summary vinculado a este artigo.
Disponibilidade de dados
Os dados de sequenciamento de DNA gerados estão disponíveis no European Nucleotide Archive (ENA) sob o acesso de estudo PRJEB42199 . Previously published genomic data analysed here are available under accession numbers PRJNA448733 , PRJCA000335, PRJEB20635 , PRJNA496590 , PRJNA494815 , PRJEB7788 , PRJEB13070 , PRJNA319283 , PRJEB22026 , PRJNA608847 , PRJEB38079 , PRJEB39580 and PRJEB41490 , with individual genomes used listed in Supplementary Data 2. O genoma de referência canFam3.1 está disponível sob o NCBI assembly accession GCF_000002285.3 .
Referências
Savolainen, P., Zhang, Y.-P., Luo, J., Lundeberg, J. & Leitner, T. Evidência genética para uma origem do leste asiático de cães domésticos. Ciência 298 , 1610-1613 (2002).
CASPubMedGoogle ScholarWang, G.-D. et ai. Fora do sul da Ásia Oriental: a história natural dos cães domésticos em todo o mundo. Célula Res. 26 , 21-33 (2016).
Google ScholarFrantz, LAF et ai. Evidências genômicas e arqueológicas sugerem uma origem dupla de cães domésticos. Ciência 352 , 1228-1231 (2016).
CASPubMedGoogle ScholarShannon, LM et ai. A estrutura genética em cães da aldeia revela uma origem de domesticação da Ásia Central. Proc. Natl Acad. Sci. EUA 112 , 13639–13644 (2015).
CASPubMedPubMed CentralGoogle ScholarThalmann, O. et ai. Genomas mitocondriais completos de canídeos antigos sugerem uma origem europeia de cães domésticos. Ciência 342 , 871-874 (2013).
CASPubMedGoogle ScholarVonholdt, BM et ai. Análises de SNP e haplótipos em todo o genoma revelam uma rica história subjacente à domesticação de cães. Natureza 464 , 898-902 (2010).
CASPubMedPubMed CentralGoogle ScholarBotigué, LR et al. Os genomas de cães europeus antigos revelam continuidade desde o início do Neolítico. Nat. Comum. 8 , 16082 (2017).
PubMedPubMed CentralGoogle ScholarBergstrõm, A. et ai. Origens e legado genético de cães pré-históricos. Ciência 370 , 557-564 (2020).
PubMed CentralGoogle ScholarTian, H. et ai. O transporte intraflagelar 88 ( IFT88 ) é crucial para o desenvolvimento craniofacial em camundongos e é um gene candidato para fissura labiopalatina humana. Zumbir. Mol. Genet. 26 , 860-872 (2017).
PubMedPubMed CentralGoogle ScholarFan, Z. et ai. Padrões mundiais de variação genômica e mistura em lobos cinzentos. Genoma Res. 26 , 163-173 (2016).
PubMedPubMed CentralGoogle Scholarvon Holdt, BM et ai. A análise da sequência do genoma inteiro mostra que duas espécies endêmicas de lobo norte-americano são misturas do coiote e do lobo cinzento. Sci. Av. 2 , e1501714 (2016).
PubMedPubMed CentralGoogle ScholarHughes, PD & Gibbard, PL Uma base estratigráfica para o Último Máximo Glacial (LGM). Quat. Int. 383 , 174-185 (2015).
Skoglund, P., Ersmark, E., Palkopoulou, E. & Dalén, L. Antigo genoma do lobo revela uma divergência precoce de ancestrais de cães domésticos e mistura em raças de alta latitude. atual Biol. 25 , 1515-1519 (2015).
PubMedGoogle ScholarRamos-Madrigal, J. et ai. Genomas de lobos siberianos do Pleistoceno descobrem várias linhagens de lobos extintos. atual Biol. 31 , 198-206 (2020).
Google ScholarJanssens, L. et ai. Um novo olhar para um cachorro velho: Bonn-Oberkassel reconsiderado. J. Archaeol. Sci. 92 , 126-138 (2018).
Perri, AR et ai. A domesticação de cães e a dupla dispersão de pessoas e cães nas Américas. Proc. Natl Acad. Sci. EUA 118 , e2010083118 (2021).
PubMedPubMed CentralGoogle ScholarSinding, M.-HS et ai. Cães adaptados ao Ártico surgiram na transição Pleistoceno-Holoceno. Ciência 368 , 1495-1499 (2020).
CASPubMedPubMed CentralGoogle ScholarPečnerová, P. et ai. A sexagem baseada no genoma fornece pistas sobre o comportamento e a estrutura social do mamute lanoso. atual Biol. 27 , 3505-3510 (2017).
Google ScholarGower, G. et ai. Preconceito generalizado do sexo masculino em coleções de fósseis e museus de mamíferos. Proc. Natl Acad. Sci. EUA 116 , 19019–19024 (2019).
PubMedPubMed CentralGoogle ScholarDrummond, AJ & Rambaut, A. BEAST: Análise evolutiva Bayesiana por amostragem de árvores. BMC Evol. Biol. 7 , 214 (2007).
PubMed CentralGoogle ScholarLoog, L. et ai. O DNA antigo sugere que os lobos modernos traçam sua origem a uma expansão do Pleistoceno Superior da Beringia. Mol. Eco 29 , 1596-1610 (2020).
PubMed CentralGoogle ScholarGopalakrishnan, S. et ai. O fluxo gênico interespecífico moldou a evolução do gênero Canis . atual Biol. 28 , 3441-3449 (2018).
PubMedPubMed CentralGoogle ScholarWang, M.-S. et ai. A hibridização antiga com uma população desconhecida facilitou a adaptação de canídeos em grandes altitudes. Mol. Biol. Evoluir 37 , 2616-2629 (2020).
PubMedGoogle Scholarvon Holdt, BM et ai. Uma perspectiva de todo o genoma sobre a história evolutiva de enigmáticos canídeos semelhantes a lobos. Genoma Res. 21 , 1294-1305 (2011).
PubMedPubMed CentralGoogle ScholarSinding, M.-HS et ai. Genômica populacional de lobos cinzentos e canídeos semelhantes a lobos na América do Norte. PLoS Genet. 14 , e1007745 (2018).
PubMed CentralGoogle ScholarWang, K., Mathieson, I., O'Connell, J. & Schiffels, S. Rastreamento da estrutura da população humana ao longo do tempo a partir de sequências genômicas inteiras. PLoS Genet. 16 , e1008552 (2020).
PubMedPubMed CentralGoogle ScholarKurtén, B. & Anderson, E. Mamíferos do Pleistoceno da América do Norte (Columbia University Press, 1980).
Hu, A. et ai. Influência do fluxo do Estreito de Bering e da circulação do Atlântico Norte nas mudanças glaciais do nível do mar. Nat. Geosci. 3 , 118-121 (2010).
CASGoogle ScholarVershinina, AO et ai. Genomas de cavalos antigos revelam o momento e a extensão das dispersões pela Ponte Terrestre de Bering. Mol. Eco 30 , 6144-6161 (2021).
Google ScholarLeonard, JA et ai. Extinções da megafauna e o desaparecimento de um lobo ecomorfo especializado. atual Biol. 17 , 1146-1150 (2007).
PubMedGoogle ScholarHudson, RR, Slatkin, M. & Maddison, WP Estimativa de níveis de fluxo gênico a partir de dados de sequência de DNA. Genetics 132 , 583-589 (1992).
PubMedPubMed CentralGoogle ScholarPiloto, M. et ai. Assinaturas de todo o genoma de gargalos populacionais e seleção diversificada em lobos europeus. Hereditariedade 112 , 428-442 (2014).
PubMedGoogle ScholarDufresnes, C. et ai. Uivando do passado: filogeografia histórica e perdas de diversidade em lobos cinzentos europeus. Proc. Biol. Sci. 285 , 20181148 (2018).
PubMed CentralGoogle ScholarDevlin, B. & Roeder, K. Controle genômico para estudos de associação. Biometrics 55 , 997-1004 (1999).
PubMedMATEMÁTICAGoogle ScholarSpeidel, L., Forest, M., Shi, S. & Myers, SR Um método para estimativa de genealogia de todo o genoma para milhares de amostras. Nat. Genet. 51 , 1321-1329 (2019).
PubMedPubMed CentralGoogle ScholarStern, AJ, Wilton, PR & Nielsen, R. Um método de probabilidade total aproximado para inferir seleção e trajetórias de frequência de alelos de dados de sequência de DNA. PLoS Genet. 15 , e1008384 (2019).
PubMed CentralGoogle ScholarFreedman, AH et ai. O sequenciamento do genoma destaca a dinâmica da história inicial dos cães. PLoS Genet. 10 , e1004016 (2014).
PubMed CentralGoogle ScholarRimbault, M. et ai. Variantes derivadas de seis genes explicam quase metade da redução de tamanho em raças de cães. Genoma Res. 23 , 1985-1995 (2013).
PubMedPubMed CentralGoogle ScholarWebster, MT et ai. Variantes genéticas ligadas no cromossomo 10 controlam a morfologia da orelha e a massa corporal entre raças de cães. BMC Genomics 16 , 474 (2015).
PubMed CentralGoogle ScholarPlassais, J. et ai. O sequenciamento completo do genoma de canídeos revela regiões genômicas sob seleção e variantes que influenciam a morfologia. Nat. Comum. 10 , 1489 (2019).
PubMedPubMed CentralGoogle ScholarBoyko, AR et al. Uma arquitetura genética simples está subjacente à variação morfológica em cães. PLoS Biol. 8 , e1000451 (2010).
PubMed CentralGoogle ScholarAnderson, TM et ai. História molecular e evolutiva do melanismo em lobos cinzentos norte-americanos. Ciência 323 , 1339-1343 (2009).
CASPubMedPubMed CentralGoogle ScholarHaak, W. et ai. A migração maciça da estepe foi uma fonte de línguas indo-européias na Europa. Natureza 522 , 207-211 (2015).
CASPubMedPubMed CentralGoogle ScholarMech, LD Padrões inexplicáveis de dispersão natal do lobo cinzento Canis lupus . Mam. Rev. 50 , 314-323 (2020).
Baumann, C. et ai. Uma proposta refinada para a origem dos cães: o estudo de caso de Gnirshöhle, uma caverna magdaleniana. Sci. Rep. 11 , 5137 (2021).
CASPubMedPubMed CentralGoogle ScholarGermonpre, M. et ai. Cães e lobos fósseis de sítios paleolíticos na Bélgica, Ucrânia e Rússia: osteometria, DNA antigo e isótopos estáveis. J. Archaeol. Sci. 36 , 473-490 (2009).
Davis, SJM & Valla, FR Evidência para domesticação do cão há 12.000 anos no Natufian de Israel. Nature 276 , 608-610 (1978).
Google ScholarMeyer, M. & Kircher, M. Preparação da biblioteca de sequenciamento Illumina para captura e sequenciamento de alvos altamente multiplexados. Porto da Primavera Fria. Protocolo 2010 , db.prot5448 (2010).
Rodríguez-Varela, R. et al. Análises genômicas de restos humanos da conquista pré-europeia das Ilhas Canárias revelam grande afinidade com os modernos norte-africanos. atual Biol. 27 , 3396-3402 (2017).
Google ScholarErsmark, E. et ai. Demografia populacional e diversidade genética no leão das cavernas do Pleistoceno. Open Quatern ., https://doi.org/10.5334/oq.aa (2015).
Stanton, DWG et ai. Origem do Pleistoceno inicial e extensa diversidade intra-espécies do extinto leão das cavernas. Sci. Rep. 10 , 12621 (2020).
PubMedPubMed CentralGoogle ScholarDabney, J. et ai. Sequência completa do genoma mitocondrial de um urso das cavernas do Pleistoceno Médio reconstruído a partir de fragmentos de DNA ultracurtos. Proc. Natl Acad. Sci. EUA 110 , 15758–15763 (2013).
CASPubMedPubMed CentralGoogle ScholarGansauge, M.-T. & Meyer, M. Preparação de biblioteca de DNA de fita simples para o sequenciamento de DNA antigo ou danificado. Nat. Protocolo 8 , 737-748 (2013).
Google ScholarPoinar, HN & Cooper, A. DNA antigo: faça certo ou não faça nada. Science 5482 , 416 (2000).
Knapp, M. & Hofreiter, M. Seqüenciamento de próxima geração de DNA antigo: requisitos, estratégias e perspectivas. Genes 1 , 227-243 (2010).
PubMedPubMed CentralGoogle ScholarKircher, M. Análise de dados de sequenciamento de DNA antigo de alto rendimento. Métodos Mol. Biol. 840 , 197-228 (2012).
PubMedGoogle ScholarOrlando, L. et ai. Recalibrando a evolução do Equus usando a sequência do genoma de um cavalo do início do Pleistoceno Médio. Natureza 499 , 74-78 (2013).
CASPubMedGoogle ScholarCaroe, C. et ai. Preparação de biblioteca de tubo único para DNA degradado. Métodos Eco. Evoluir 9 , 410-419 (2018).
Mak, SST et ai. Desempenho comparativo das plataformas de sequenciamento BGISEQ-500 vs Illumina HiSeq2500 para sequenciamento paleogenômico. Gigascience 6 , 1–13 (2017).
PubMedPubMed CentralGoogle ScholarFulton, TL & Shapiro, B. Criação de um antigo laboratório de DNA. Métodos Mol. Biol. 1963 , 1-13 (2019).
PubMedGoogle ScholarKorlević, P. & Meyer, M. Pré-tratamento: remoção de contaminação de DNA de ossos e dentes antigos usando hipoclorito de sódio e fosfato. Métodos Mol. Biol. 1963 , 15-19 (2019).
Google ScholarKapp, JD, Green, RE & Shapiro, B. Um método de preparação de biblioteca genômica de fita simples rápido e eficiente otimizado para DNA antigo. J. Hered. 112 , 241-249 (2021).
PubMed CentralGoogle ScholarHarney, É. et ai. Um protocolo minimamente destrutivo para extração de DNA de dentes antigos. Genoma Res. 31 , 472-483 (2021).
PubMed CentralGoogle ScholarGamba, C. et ai. Fluxo e estase do genoma em um transecto de cinco milênios da pré-história europeia. Nat. Comum. 5 , 5257 (2014).
CASPubMedGoogle ScholarLi, H. & Durbin, R. Alinhamento rápido e preciso de leitura curta com a transformada de Burrows–Wheeler. Bioinformática 25 , 1754-1760 (2009).
PubMedPubMed CentralGoogle ScholarRamos Madrigal, J. et ai. Genomas de lobos siberianos do Pleistoceno extintos fornecem informações sobre a origem dos lobos atuais. atual Biol. 31 , 199-206 (2021).
Skoglund, P., Northoff, BH & Shunkov, MV Separando DNA endógeno antigo da contaminação moderna em um neandertal siberiano. Proc. Natl Acad. Sci. EUA 111 , 2229–2234 (2014).
CASPubMedPubMed CentralGoogle ScholarLiu, Y.-H. et ai. O sequenciamento de todo o genoma de cães africanos fornece informações sobre adaptações contra parasitas tropicais. Mol. Biol. Evoluir 35 , 287-298 (2018).
PubMedGoogle ScholarKardos, M. et ai. Consequências genômicas da endogamia intensiva em uma população isolada de lobos. Nat. Eco Evoluir 2 , 124-131 (2018).
Google ScholarLi, H. Leituras de sequência de alinhamento, sequências de clone e contigs de montagem com BWA-MEM. Pré-impressão em https://arxiv.org/abs/1303.3997 (2013).
McKenna, A. et ai. O Genome Analysis Toolkit: uma estrutura MapReduce para analisar dados de sequenciamento de DNA de última geração. Genoma Res. 20 , 1297-1303 (2010).
PubMedPubMed CentralGoogle ScholarNí Leathlobhair, M. et al. A história evolutiva dos cães nas Américas. Ciência 361 , 81-85 (2018).
PubMedGoogle ScholarNiemann, J. et ai. Sobrevivência estendida de lobos siberianos do Pleistoceno até o início do século 20 na ilha de Honshū. iScience 24 , 101904 (2021).
PubMedGoogle ScholarLi, H. et ai. O formato Sequence Alignment/Map e SAMtools. Bioinformática 25 , 2078–2079 (2009).
PubMed CentralGoogle ScholarArnason, U., Gullberg, A., Janke, A. & Kullberg, M. Análises mitogenômicas de relacionamentos caniformes. Mol. Filogeneto. Evoluir 45 , 863-874 (2007).
PubMedGoogle ScholarBjörnerfeldt, S., Webster, MT & Vilà, C. Relaxamento da restrição seletiva no DNA mitocondrial do cão após a domesticação. Genoma Res. 16 , 990-994 (2006).
PubMed CentralGoogle ScholarMatsumura, S., Inoshima, Y. & Ishiguro, N. Reconstruindo a história de colonização de linhagens perdidas de lobos pela análise do genoma mitocondrial. Mol. Filogeneto. Evoluir 80 , 105-112 (2014).
Google ScholarMeng, C., Zhang, H. & Meng, Q. Genoma mitocondrial do lobo tibetano. DNA mitocondrial 20 , 61-63 (2009).
PubMedGoogle ScholarPang, J.-F. et ai. Os dados do mtDNA indicam uma origem única para cães ao sul do rio Yangtze, há menos de 16.300 anos, de vários lobos. Mol. Biol. Evoluir 26 , 2849-2864 (2009).
PubMedPubMed CentralGoogle ScholarZhang, H. et ai. Genoma mitocondrial completo de Canis lupus campestris . DNA mitocondrial 26 , 255-256 (2015).
PubMedGoogle ScholarSievers, F. et ai. Geração rápida e escalável de alinhamentos de múltiplas sequências de proteínas de alta qualidade usando Clustal Omega. Mol. Sistema Biol. 7 , 539 (2011).
PubMed CentralGoogle ScholarSuchard, MA et ai. Integração de dados filogenéticos e filodinâmicos bayesianos usando BEAST 1.10. Evolução do vírus. 4 , vey016 (2018).
PubMed CentralGoogle ScholarDarriba, D., Taboada, GL, Doallo, R. & Posada, D. jModelTest 2: mais modelos, novas heurísticas e computação paralela. Nat. Métodos 9 , 772 (2012).
PubMedPubMed CentralGoogle ScholarPatterson, N. et ai. Mistura antiga na história humana. Genética 192 , 1065-1093 (2012).
PubMed CentralGoogle ScholarPatterson, N., Price, AL & Reich, D. Estrutura populacional e autoanálise. PLoS Genet. 2 , e190 (2006).
PubMed CentralGoogle ScholarPoplin, R. et ai. Escalando a descoberta precisa de variantes genéticas para dezenas de milhares de amostras. Pré -impressão em bioRxiv https://doi.org/10.1101/201178 (2018).
Koch, E. et ai. Estimativa da taxa de mutação de novo em lobos de pedigree conhecido. Mol. Biol. Evoluir 36 , 2536-2547 (2019).
PubMed CentralGoogle ScholarChang, CC et ai. PLINK de segunda geração: enfrentando o desafio de conjuntos de dados maiores e mais ricos. GigaScience 4 , 7 (2015).
Deane-Coe, PE, Chu, ET, Slavney, A., Boyko, AR & Sams, AJ Teste de DNA direto ao consumidor de 6.000 cães revela duplicação de 98,6 kb associada a olhos azuis e heterocromia em Huskies Siberianos. PLoS Genet. 14 , e1007648 (2018).
PubMed CentralGoogle ScholarHudson, RR Gerando amostras sob um modelo neutro de variação genética de Wright-Fisher. Bioinformática 18 , 337-338 (2002).
PubMedGoogle Scholar
Reconhecimentos
Este trabalho foi financiado por doações a P. Skoglund do Conselho Europeu de Pesquisa (concessão nº 852558), da Fundação Erik Philip Sörensen e do Laboratório Ciência para a Vida, Programa Sueco de Biodiversidade, disponibilizados pelo apoio da Fundação Knut e Alice Wallenberg. AB, LS, P. Swali e P. Skoglund foram apoiados pelo financiamento central do Francis Crick Institute (FC001595) da Cancer Research UK, do UK Medical Research Council e do Wellcome Trust. P. Skoglund também foi apoiado pela Vallee Foundation, pela European Molecular Biology Organization e pelo Wellcome Trust (217223/Z/19/Z). Os cálculos foram suportados pelo SNIC-UPPMAX. Também agradecemos o apoio do Science for Life Laboratory, da Fundação Knut e Alice Wallenberg, a Infraestrutura Nacional de Genômica, financiada pelo Conselho de Pesquisa Sueco e pelo Centro Multidisciplinar de Uppsala para Ciência Computacional Avançada, para assistência com sequenciamento massivamente paralelo e acesso à infraestrutura computacional UPPMAX. Agradecemos à comunidade de mineração de ouro de Yukon e às Primeiras Nações, incluindo os Tr'ondëk Hwëch'in, pelo apoio contínuo à nossa pesquisa de paleontologia nos territórios de Yukon, Canadá. Agradecemos ao Danish National High-Throughput Sequencing Center e BGI-Europe pela assistência na geração de dados de sequenciamento e ao Danish National Supercomputer for Life Sciences–Computerome ( incluindo o Tr'ondëk Hwëch'in, pelo apoio contínuo à nossa pesquisa de paleontologia nos territórios de Yukon, Canadá. Agradecemos ao Danish National High-Throughput Sequencing Center e BGI-Europe pela assistência na geração de dados de sequenciamento e ao Danish National Supercomputer for Life Sciences–Computerome ( incluindo o Tr'ondëk Hwëch'in, pelo apoio contínuo à nossa pesquisa de paleontologia nos territórios de Yukon, Canadá. Agradecemos ao Danish National High-Throughput Sequencing Center e BGI-Europe pela assistência na geração de dados de sequenciamento e ao Danish National Supercomputer for Life Sciences–Computerome (https://computerome.dtu.dk) para recursos computacionais. Agradecemos ao Museu Nacional do País de Gales pelo apoio contínuo à amostragem. M. Germonpré agradece o apoio do projeto Brain.be 2.0 ICHIE (BELSPO B2/191/P2/ICHIE). O MTPG foi apoiado pelo Conselho Europeu de Pesquisa (concessão nº 681396). O M.-HSS foi apoiado pelas Fundações Velux através do Projeto Qimmeq, o Aage og Johanne Louis-Hansens Fond e o Independent Research Fund Denmark (8028-00005B). A LD reconhece o apoio da FORMAS (2018-01640). O DWGS recebeu financiamento para este projeto do programa de pesquisa e inovação Horizon 2020 da União Europeia sob o acordo de subvenção Marie Skłodowska-Curie no. 796877. MP foi apoiado pela Agência Nacional Polonesa para Intercâmbio Acadêmico – NAWA (concessão nº PPN/PPO/2018/1/00037). VJS foi apoiado pelo Programa de Prioridade de Pesquisa Universitária da Universidade de Zurique 'Evolução em Ação: Dos Genomas aos Ecossistemas'. Esta pesquisa foi realizada com a participação da ZIN RAS (bolsa nº 075-15-2021-1069). Agradecemos ao museu do Instituto de Ecologia Vegetal e Animal UB RAS (Ekaterinburg, Rússia) pelo fornecimento de amostras. RPJ e C.O'D. foram apoiados pelo Comitê Permanente de Arqueologia da Royal Irish Academy através do Archaeological Excavation Research Grant Scheme. EYP, PN e VVP são apoiados pela Russian Science Foundation (concessão nº 16-18-10265-RNF e 21-18-00457-RNF). YVK foi apoiado pela Russian Science Foundation (concessão nº 20-17-00033). O MH foi apoiado pelo European Research Council (consolidator grant GeneFlow no. 310763). ML-G. foi apoiado pela Fundação de Ciência Tcheca GAČR (concessão nº 15-06446S) e financiamento institucional do Museu Morávio do Ministério da Cultura Tcheco (IP DKRVO 2019-2023, MK000094862). O LS é apoiado pela bolsa Sir Henry Wellcome (220457/Z/20/Z). Agradecemos ao Staatliches Museum für Naturkunde Stuttgart pelo acesso às amostras. LF e GL foram apoiados por subsídios do Conselho Europeu de Pesquisa (ERC-2013-StG-337574-UNDEAD e ERC-2019-StG-853272-PALAEOFARM) e subsídios do Conselho de Pesquisa Ambiental Natural (NE/K005243/1, NE/K003259/1, NE/S007067/1 e NE/S00078X/1). A LF também foi apoiada pelo Wellcome Trust (210119/Z/18/Z). Esta pesquisa foi financiada no todo ou em parte pelo Wellcome Trust (FC001595). Para fins de acesso aberto,
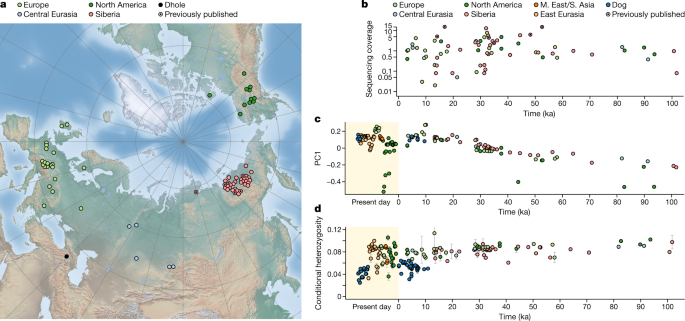
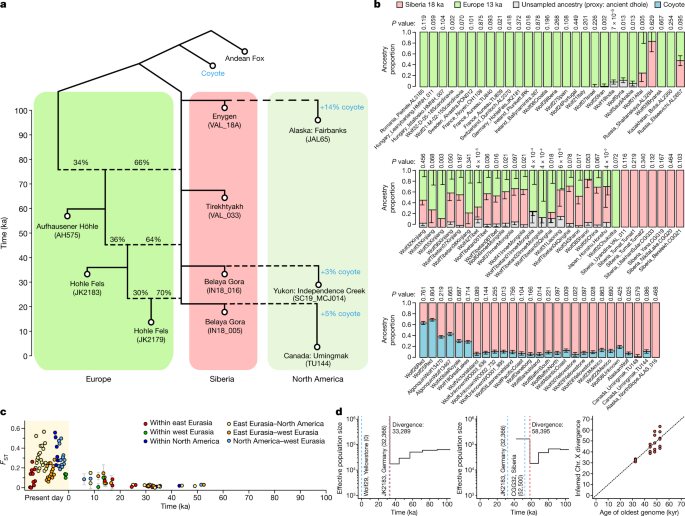
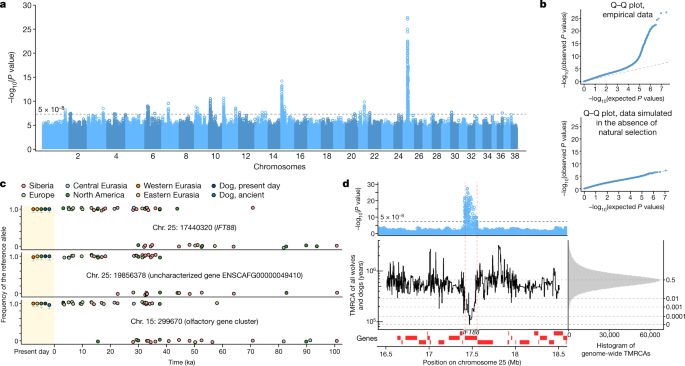
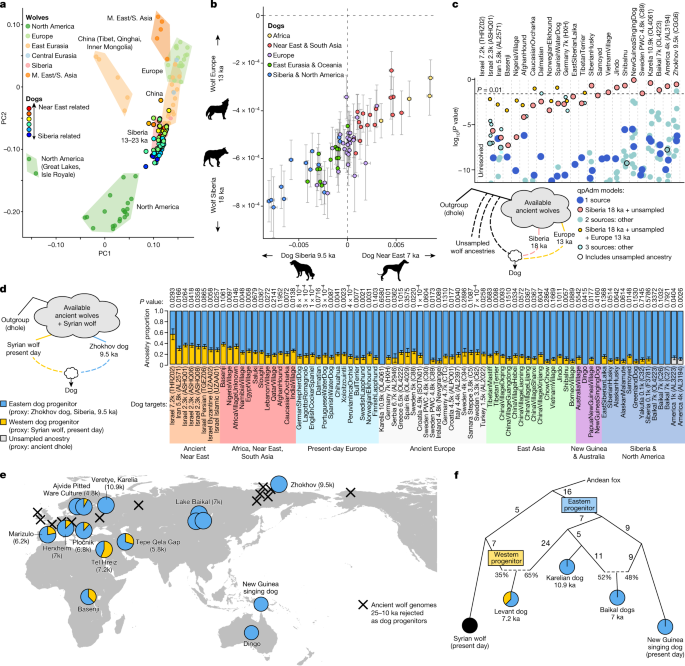
Nenhum comentário:
Postar um comentário
Observação: somente um membro deste blog pode postar um comentário.