10 anos de descoberta do GWAS: biologia, função e tradução
Resumo
A aplicação do desenho experimental de estudos de associação genômica ampla (GWASs) já tem 10 anos (jovem), e aqui revisamos a notável gama de descobertas que ele facilitou na genética de populações e de características complexas, na biologia de doenças e na tradução em direção a novas terapêuticas. Prevemos as prováveis descobertas nos próximos 10 anos, quando os GWAS serão baseados em milhões de amostras com dados de array imputados a um grande painel de referência totalmente sequenciado e em centenas de milhares de amostras com dados de sequenciamento do genoma completo.
Palavras-chave: estudo de associação genômica ampla, SNP, obesidade, doença autoimune, esquizofrenia, herdabilidade
Texto principal
Introdução
Aqui, revisamos a notável gama de descobertas que os estudos de associação genômica ampla (GWASs) facilitaram na genética de populações e de características complexas, na biologia de doenças e na tradução para novas terapêuticas. Nas seções introdutórias, fornecemos os antecedentes desta revisão, resumimos seu escopo e layout e revisitamos a justificativa científica para os GWASs. Em seguida, revisamos as conclusões gerais que podem ser tiradas das descobertas do GWAS em uma ampla gama de características. Posteriormente, destacamos resultados mais específicos de descobertas e métodos no caminho do GWAS para a biologia e revisamos o progresso em três doenças exemplares, nomeadamente diabetes tipo 2 (DT2 [MIM: 125853 ]), doenças autoimunes (MIM: 109100 ) e esquizofrenia (MIM: 181500 ). Terminamos a revisão com uma série de seções sobre as limitações dos projetos experimentais atuais e possíveis maneiras de superá-las e uma previsão sobre o futuro dos GWASs para características humanas.
Background
Há cinco anos, vários de nós revisamos (e demos nossa opinião sobre) os primeiros 5 anos de descobertas resultantes do projeto experimental do GWAS. 1 That review sought to set the record straight on the discoveries made by GWASs because at that time, there was still a level of misunderstanding and distrust about the purpose of and discoveries made by GWASs. There is now much more acceptance of the experimental design because the empirical results have been robust and overwhelming, as reviewed here.
Escopo e Estrutura
Os dados gerados a partir de pesquisas de SNP em todo o genoma foram explorados para abordar muitas questões científicas além das associações de características de SNP. Não temos espaço para dar uma cobertura adequada das descobertas em genética evolutiva e populacional, nem podemos cobrir completamente os muitos desenvolvimentos em métodos analíticos, embora mencionemos brevemente alguns desenvolvimentos recentes. O escopo da nossa revisão são novas descobertas sobre a genética e a biologia resultante de doenças comuns em adultos (doenças autoimunes, metabólicas e psiquiátricas em particular) e seus fatores de risco e as implicações mais amplas dessas descobertas. As descobertas do GWAS afetaram e estão afetando uma ampla variedade de doenças e características, muitas das quais foram abordadas em outras análises aprofundadas. Nosso foco está nas associações entre características complexas e SNPs, mas notamos que tem havido muitas associações relatadas entre características e variantes de número de cópias (CNVs) e que existem mecanismos conhecidos pelos quais as CNVs podem ser associadas a doenças. 2 Os resultados de outras pesquisas genômicas, incluindo estudos de exoma e sequenciamento do genoma completo (WGS), não são revisados aqui.
Fundamentação e base científica do GWAS
O GWAS é um desenho experimental utilizado para detectar associações entre variantes genéticas e características em amostras de populações. O objetivo principal destes estudos é compreender melhor a biologia das doenças, partindo do pressuposto de que uma melhor compreensão levará à prevenção ou a um melhor tratamento. O caminho do GWAS para a biologia não é simples porque uma associação entre uma variante genética num locus genómico e uma característica não é diretamente informativa no que diz respeito ao gene alvo ou ao mecanismo pelo qual a variante está associada a diferenças fenotípicas. No entanto, tal como aqui revisto, novos tipos de dados, novas tecnologias moleculares e novos métodos analíticos proporcionaram oportunidades para colmatar a lacuna de conhecimento da sequência à consequência. Os GWAS também foram implementados com sucesso para definir melhor o papel relativo dos genes e do ambiente no risco de doenças, auxiliando na previsão de riscos (permitindo a medicina preventiva e personalizada) e investigando a seleção natural e as diferenças populacionais ( Tabela 1 ).
Tabela 1.
O papel dos arranjos GWAS SNP nas descobertas genéticas humanas
| Análise | Propósito | Descobertas |
|---|---|---|
| GWAS | detecção de associações traço-SNP | ∼10.000 associações robustas com doenças e distúrbios, características quantitativas e características genômicas |
| Análise CNV em todo o genoma | detecção de associações traço-CNV | centenas de associações com doenças e distúrbios |
| Avaliação de LD em todo o genoma | quantificando a arquitetura do genoma | grande variação em LD no genoma |
| Estimativa da herdabilidade do SNP um | arquitetura genética | grande proporção de variação genética capturada por SNPs comuns |
| Estimativa de correlação genética um | detectando e quantificando pleiotropia | pleiotropia é onipresente |
| Pontuações de risco poligênico um | detecção de pleiotropia; validando descobertas do GWAS | a previsão fora da amostra funciona conforme o esperado; detecção de novas associações de características |
| Mendelian randomizationa | testing causal relationships | replication of known causal relationships; empirical evidence of observational associations that are not causal |
| Population differences in allele frequencies | reconstructing human population history; detecting selection | genetic structure can mimic geographical structure; evidence of natural selection |
| Trait GWAS with -omics GWASa | fine-mapping; detecting target genes; function | two-thirds of GWAS-associated loci implicate a gene that is not the nearest gene to the most associated SNP |
Estas análises podem ser realizadas com estatísticas resumidas do GWAS.
Até o momento, os GWAS dependem e exploram o desequilíbrio de ligação (LD), a estrutura de correlação que existe entre as variantes de DNA no genoma humano atual como resultado de forças evolutivas históricas, particularmente tamanho finito da população, mutação, taxa de recombinação e seleção natural. O poder estatístico para detectar associações entre variantes de DNA e uma característica depende do tamanho da amostra experimental, da distribuição dos tamanhos de efeito de variantes genéticas causais (desconhecidas) que estão segregando na população, da frequência dessas variantes e do LD entre os genotipados observados. Variantes de DNA e as variantes causais desconhecidas. Portanto, o potencial de sucesso de um GWAS para uma determinada característica ou doença depende de (1) quantos loci que afetam a característica segregam na população, (2) da distribuição conjunta do tamanho do efeito e da frequência alélica nesses loci (às vezes chamados de genéticos). arquitetura), (3) o tamanho da amostra experimental, (4) o painel de variantes do genoma que são usadas no GWAS e (5) quão heterogênea é a característica ou doença que está sendo estudada. A última diz respeito tanto à biologia da característica quanto à capacidade de diagnosticá-la ou medi-la com precisão.
Se a arquitetura genética de uma característica ou doença específica fosse conhecida, os experimentos ideais poderiam ser projetados para detectar variantes específicas. No entanto, apesar de muitos estudos teóricos sobre a provável relação entre a frequência alélica e os loci característicos, até o início dos GWASs, havia muito poucos dados empíricos para validar a previsão dos modelos teóricos.
Os GWASs foram facilitados pelo desenvolvimento de matrizes SNP relativamente baratas. Matrizes SNP comumente usadas variam em seu conteúdo, mas geralmente contêm de 200.000 a mais de 2.000.000 SNPs. Até o momento, a maioria das variantes genéticas pesquisadas por meio de GWASs são comuns na população, pois apresentam uma frequência alélica menor (MAF), normalmente maior que 1%. Para os fins desta revisão, definimos arbitrariamente variantes comuns como tendo MAF ≥ 1% e variantes raras como tendo MAF <1%. O GWAS como projeto experimental é mais do que apenas um estudo baseado em array de variantes comuns. Por exemplo, estudos de associação utilizando dados WGS também são GWASs. Há um continuum de GWASs baseados em matrizes SNP até aqueles que usam WGS, e a única diferença (além do custo) é a densidade de cobertura da variação no genoma e o espectro MAF das variantes.
LD entre variantes genéticas é comumente medido como uma correlação quadrada (r 2 ) porque esta medida é linear no tamanho da amostra necessária para detectar associação entre uma variante causal genotipada observada e uma variante causal não observada. LD r 2 pode ser grande apenas se as frequências alélicas nos dois loci corresponderem, 3 , 4 e esta é a razão pela qual os GWAS de matrizes SNP comuns não são poderosos o suficiente para detectar associações devido a variantes causais raras (além de considerações sobre o tamanho da amostra; veja abaixo). Imputação estatística 5 , 6 , 7 de variantes não observadas pode recuperar algumas das informações perdidas devido ao LD imperfeito entre genótipos observados e variantes causais não observadas. A imputação é possibilitada pelo fato de que os genótipos de variantes genéticas não observadas podem ser previstos pelos haplótipos inferidos a partir de múltiplos SNPs observados e pelos haplótipos observados a partir de um painel de referência totalmente sequenciado.
Na Figura 1 , resumimos os cálculos de poder (ver Apêndice A para teoria) do tamanho mínimo da amostra necessário para detectar uma associação em função do método genotípico (matriz SNP mais imputação ou WGS), frequência alélica e tamanho do efeito. Dado que a imputação estatística de variantes tão pouco frequentes como 1/1.000 ainda é razoavelmente precisa, 8 não é possível obter muito poder de detecção com o WGS. Para variantes ultra-raras, por exemplo, aquelas com frequência de 1/100.000, o WGS pode identificar associações, mas apenas quando os tamanhos de efeito dos polimorfismos (mutações) são muito grandes. Por exemplo, para essas variantes raras com um tamanho de efeito de 1 unidade de desvio padrão fenotípico (cerca de 7 cm para altura ou 5 unidades de IMC), é necessário um tamanho de amostra superior a um milhão (ou seja, uma contagem alélica de dez). Para estudos de caso-controle de doenças, os tamanhos dos efeitos de β = 0,01, 0,1 e 1 desvio padrão fenotípico na Figura 1 correspondem aproximadamente a razões de chances de 1,02, 1,2 e 4, respectivamente, se assumirmos que tanto a frequência alélica quanto a população prevalência são 0,01 ou menos. 9 A segregação de variantes raras com efeitos muito grandes pode ser observável em certas famílias, e então um desenho experimental baseado em família seria mais eficiente na localização e identificação de tais características (quase) monogênicas. Além disso, outras varreduras genômicas, como os estudos WES e WGS, permitem testar uma carga de variantes raras em unidades funcionais compartilhadas (por exemplo, genes) de uma forma que não é acessível aos GWASs.
Figura 1.
Minimum Sample Sizes for Detecting Trait-SNP Associations from Imputed and WGS Data
Required sample sizes for detecting association were calculated with (Equation A1), (Equation A2), (Equation A3), (Equation A4), (Equation A5) under the assumption of a type I error rate of 5 × 10−8, 80% power, and Hardy-Weinberg equilibrium. Effect sizes (β) are in phenotypic standard deviation units. For genotyped SNPs imputed to a fully sequenced reference, we have used the average imputation Rimp2 values reported by the Haplotype Reference Consortium8 in their Figure S3. This is a conservative estimate of imputation accuracy because it is based on a less dense genotyping array. For the WGS data, we have assumed no sequencing errors. Note that for some combinations of allele frequency and effect size, the required minimum number of individuals for detecting association exceeds 100 million.
Os resultados da Figura 1 baseiam-se em amostras populacionais não selecionadas ou amostras de caso-controle baseadas na população e na detecção de associação entre a característica ou doença e a mesma variante genética. A potência será aumentada para casos altamente comprovados e enriquecida para casos extremos ou estudos familiares com múltiplos casos de uma doença rara. Além disso, a utilização de dados WGS para análise de associação de variantes raras tem o potencial de aumentar o poder através da combinação de alelos de impacto semelhante (por exemplo, através de testes de carga através de um gene) sob a suposição de múltiplas variantes causais independentes numa região genética. Esta estratégia justifica-se a partir do conhecimento de doenças monogénicas, onde é típico que diferentes variantes do mesmo gene se segregam com doenças em famílias diferentes. No entanto, tais testes também apresentam desafios porque é necessário conhecimento prévio sobre a função ou frequência para determinar quais alelos num gene devem ser incluídos na contagem de carga.
Resultados Gerais
Características complexas são altamente poligênicas
Os resultados do GWAS já foram relatados para centenas de características complexas em uma ampla gama de domínios, incluindo doenças comuns, características quantitativas que são fatores de risco para doenças, fenótipos de imagens cerebrais, medidas genômicas, como expressão gênica e metilação do DNA, e características sociais e comportamentais. como o bem-estar subjetivo e o nível de escolaridade. Cerca de 10.000 associações fortes foram relatadas entre variantes genéticas e uma ou mais características complexas, 10 onde “forte” é definido como estatisticamente significativo no limiar do valor p de todo o genoma de 5 × 10 −8 , excluding other genome-wide-significant SNPs in LD (r2 > 0.5) with the strongest association (Figure 2). GWAS associations have proven highly replicable, both within and between populations,11, 12 sob a suposição de tamanhos de amostra adequados.
Figure 2.
Cronograma de descoberta de características SNP do GWAS
Os dados utilizados para geração do gráfico foram retirados do Catálogo GWAS. 10 SNPs e características foram selecionados de acordo com os seguintes filtros. SNPs foram selecionados com valor de p <5 × 10 −8 . Para cada característica com dois ou mais SNPs selecionados, os SNPs foram removidos se tivessem um LD r 2 > 0,5 (calculado a partir de dados da fase 3 de 1000 genomas) com outros SNPs selecionados e seu valor de p foi maior. Para cada ano de descoberta, apenas as três principais características e doenças com o maior número de SNPs são rotuladas no círculo.
Uma conclusão inequívoca dos GWASs é que, para quase todas as características complexas que foram estudadas, muitos loci contribuem para a variação genética permanente. Por outras palavras, para a maioria das características e doenças estudadas, o alvo mutacional no genoma parece grande, de modo que os polimorfismos em muitos genes contribuem para a variação genética na população. Isto significa que, em média, a proporção da variância explicada nas variantes individuais é pequena. Por outro lado, como previsto anteriormente, 1, 13 this observation implies that larger experimental sample sizes will lead to new discoveries, and that is exactly what has occurred over the last decade. For example, in 2009 the first genomic locus robustly associated with liability to schizophrenia was discovered with a sample of 3,000 cases;14 by 2014, this had increased to 108 with a sample size of 35,000 cases.15 Similarly, when the concept of “missing heritability”16 was introduced, it highlighted that in 2008, only 40 genome-wide-significant SNPs had been identified for height, and together these explained about 5% of heritability.17 In 2014, the number of associated SNPs had increased to ∼700, explaining more than 20% of heritability,18 e a partir da relação entre o tamanho da amostra e as descobertas nos últimos 10 anos, é razoável prever que, nos próximos anos, este número aumentará para milhares de variantes, o que explicará cumulativamente uma proporção substancial (por exemplo, mais de um terço ) de herdabilidade.
O termo poligênico descreve a arquitetura genética que sustenta a variação de uma característica entre indivíduos de uma população, mas o que isso significa para cada indivíduo? Isso significa que cada indivíduo carregará um número de alelos que aumentam (+) e um número de alelos que diminuem (-) a característica ou o risco de doença. Existem tantas combinações possíveis desses conjuntos de alelos que é provável que cada indivíduo tenha uma combinação única e, em estudos projetados para detectar loci associados, o tamanho do efeito de cada alelo é medido no contexto de um contexto médio, e o efeito o tamanho de cada locus é pequeno.
A pleiotropia é generalizada
The number of segregating variants in the human population is large but finite. It is not known what proportion of the segregating variants are associated with complex-trait genetic variation, but the fact that each of the many studied traits is associated with variants at hundreds to thousands of loci in the genome strongly suggests that some of the underlying causal variants are the same. Multiple lines of evidence are consistent with widespread pleiotropy for complex traits. First, Mendelian mutations that cause specific syndromes or diseases are frequently associated with multiple phenotypes in an affected individual. Second, pedigree studies have reported genetic correlations between traits, implying that a number of the same variants affect two or more traits in a consistent direction.19 Third, GWASs have shown that the same genetic variants can be significantly associated with multiple diseases and traits when the phenotypes are measured on different individuals (so that no environmental associations are driving the results).20, 21, 22 In the case of auto-immune diseases, evidence implies that at some loci, the same causal variants are driving the observations of associations across diseases.23, 24, 25 Fourth, analytical methods that estimate genetic correlations from GWAS data have provided evidence for widespread pleiotropy.20, 26
O corolário da pleiotropia generalizada para características complexas é que o paradigma de “um gene, uma função, uma característica” é a forma errada de ver a variação genética no genoma humano (e o mesmo se aplica ao estudo de doenças em organismos experimentais). 27 Implica também que estudar características ou doenças isoladamente em relação à seleção natural passada ou presente pode levar a uma inferência errada. A verdadeira natureza da pleiotropia é atualmente desconhecida mas, em alguns casos, pode implicar um impacto das variantes em diferentes tecidos e/ou em diferentes idades.
Nova metodologia de análise que sustenta nova descoberta
Os dados do GWAS levaram a novos métodos de análise que se enquadram em uma série de categorias dependendo de sua finalidade: (1) métodos para modelar melhor a estrutura populacional e o parentesco entre indivíduos em uma amostra durante análises de associação, 28 , 29 , 30 , 31 , 32 , 33 , 34 (2) métodos de detecção de novas variantes e loci genéticos com base nas estatísticas resumidas do GWAS, 35 , 36 , 37 (3) métodos de estimativa e particionamento da (co)variância genética, 38 , 39 e (4) métodos de inferir causalidade. 40 , 41 , 42 Além disso, as descobertas e a interpretação do GWAS se beneficiaram substancialmente de algoritmos aprimorados na imputação estatística de genótipos não observados e na imputação estatística de genes do antígeno leucocitário humano (HLA) e polimorfismos de aminoácidos. 43 , 44 , 45 , 46
Variantes comuns juntas marcam uma proporção substancial de variação genética aditiva
Além de permitir a descoberta de associações características-locus específicas, os GWASs facilitaram a estimativa de quanto da variação genética aditiva total devido a variantes segregantes na população é marcada por SNPs genotipados e imputados. Esta quantificação da “herdabilidade do SNP” é informativa no que diz respeito à arquitetura genética desconhecida da característica. A herdabilidade do SNP forneceu orientação objetiva para informar decisões sobre quais projetos experimentais são mais eficientes na detecção de novas associações traço-locus com base em dados empíricos, ou seja, aumentando o tamanho da amostra de GWASs. A estimativa clássica da herdabilidade total em sentido restrito (estimada a partir de registros fenotípicos de amostras que incluem membros da família) captura a quantidade total de variância genética aditiva na população, independentemente da distribuição conjunta da frequência alélica e do tamanho do efeito 47 (we acknowledge a potential for bias by common environmental effects and non-additive genetic variation). In contrast, SNP heritability (estimated from tiny genetic relationships from unrelated individuals) captures only the proportion of additive genetic variance due to LD between the assayed and imputed SNPs and the unknown causal variants. Estimation and partitioning of additive genetic variation for quantitative traits and liability to disease have implied that one-third to two-thirds of genetic variation at causal variants can be tagged by common genotyped and imputed SNPs through LD.1, 48 Atualmente, não se sabe quanto da variação genética aditiva total se deve a variantes causais com frequências inferiores a 1%. As evidências dos dados genotípicos imputados para a altura implicam que uma variação genética mais aditiva é explicada por variantes com MAF <10% do que o esperado sob um modelo evolutivo neutro, consistente com a seleção purificadora dos loci associados à altura. 49 Num futuro próximo, quando a variância genética aditiva for estimada a partir de dados WGS em grandes amostras, a contribuição das variantes raras e de baixa frequência observadas será estimada explicitamente. As estimativas dos dados disponíveis até à data fornecem a primeira evidência de diferentes arquitecturas genéticas entre doenças, 50 por exemplo, há mais sinais de variantes raras para esclerose lateral amiotrófica (doença do neurônio motor [MIM: 105400 ]) do que para esquizofrenia 51 e mais loci previstos para esquizofrenia do que para distúrbios imunológicos 52 e hipertensão. 53
Observações teóricas e empíricas sugerem um lugar para variação genética não aditiva, e tem havido muitas tentativas malsucedidas de detectar epistasia com dados de GWAS. Existem várias explicações prováveis. Primeiro, há evidências limitadas de que a variação genética não aditiva constitui uma grande fração da variação genética total, portanto a detecção requer tamanhos de amostra maiores do que os necessários para os efeitos principais. Em segundo lugar, a perda de informação devido ao LD imperfeito entre SNPs genotipados e variantes causais é maior para interações do que para efeitos principais. Por exemplo, a perda de informação para efeitos aditivos é proporcional ao LD r 2 , enquanto a perda de informação para efeitos de dominância e interação aditivo-aditivo é proporcional a r 4 . A primeira observação também se aplica às interações entre genes e fatores ambientais. Um exemplo replicável de interação epistática é a interação ERAP1 -HLA para psoríase (MIM: 177900 ) e espondilite anquilosante (MIM: 106300 ). 54
The Utility of GWAS-Derived Genetic Predictors
Em 2007, foi demonstrado que era possível usar dados GWAS de estudos humanos para criar preditores genéticos para doenças e outras características complexas, estimando o tamanho do efeito em múltiplos loci em uma amostra de descoberta e usando esses efeitos SNP estimados em amostras independentes. 13, 55 para gerar uma pontuação de risco poligênico (PRS) por indivíduo. Uma revisão completa dos diferentes métodos de geração de PRS está fora do escopo desta revisão, mas atualmente a principal força motriz que influencia a precisão da previsão é o tamanho da amostra descoberta usada para estimar os efeitos de variantes individuais. Os PRS têm sido aplicados extensivamente nos últimos 5 anos, não num ambiente clínico para a previsão do risco de doença de um indivíduo saudável, mas em aplicações que facilitam novos desenhos experimentais e descobertas. As previsões poligênicas não são particularmente informativas para um indivíduo, mas explicam uma proporção suficiente de variação (entre 1% e 15% atualmente para características altamente poligênicas sem um gene principal) para separar grupos, por exemplo, amostras com os valores mais altos e mais baixos. risco. Eles também são úteis para detectar novas associações de características, correlacionando fenótipos observados em uma amostra ou coorte com a previsão genética de outra característica. Este desenho é poderoso, porque se a amostra descoberta for totalmente independente da nova amostra, uma associação observada entre uma característica complexa e um preditor genético da amostra descoberta deve ser devida a factores genéticos, uma vez que não existem factores ambientais partilhados. O paradigma dos PRSs também pode ser aplicado à predição de fenótipos moleculares como a expressão gênica mesmo quando não são observados 56 para explorar o “fenômeno” humano para associação com preditores derivados de doenças e outras características 57 ou investigando genótipo (procurado por PRS) por interação ambiental. 58
A disponibilidade pública de dados permitiu novas pesquisas e descobertas
O compartilhamento de dados genéticos na comunidade de mapeamento genético tem sido um fator importante para o sucesso do mapeamento genético. Neste momento, a grande maioria dos dados disponíveis provém de estudos de populações de ascendência europeia, e espera-se que os dados de outros grupos étnicos sejam depositados de forma mais extensiva nos próximos anos.
A disponibilidade de estatísticas resumidas do GWAS (os tamanhos dos efeitos e seus erros padrão ou valores p em milhões de SNPs) no domínio público aumentou dramaticamente nos últimos 5 anos e, em 2017, centenas desses conjuntos de dados estão disponíveis publicamente. 59 There are a number of reasons for this. Previous concerns about potential individual identification from GWAS summary data have proven to be unfounded, either because the sample size from GWAS summary statistics is typically very large or because a simple step such as providing average allele frequencies from a reference sample negates potential identification. The entire genomics field benefits from wide availability of genetic data. When a GWAS is published, full genome-wide summary statistics (at the very least) should be available for uncontrolled download. Funding bodies and journals could play a stronger role in enforcing such a requirement. The availability of summary statistics in the public domain has enabled discoveries of novel associations,37, 60, 61, 62 estimativa da herdabilidade baseada em SNP, 63 quantificação da pleiotropia em muitas características, 20 , 21 , 38 e criação de escores de predição mais precisos, bem como acompanhamento com ferramentas computacionais, ensaios funcionais e sistemas modelo para identificação de genes candidatos.
Para um futuro próximo, o Biobank do Reino Unido está a ampliar ainda mais as barreiras, ao divulgar genótipos de todo o genoma e dados fenotípicos ricos sobre 500.000 pessoas para a comunidade de investigação internacional.
Do GWAS à Biologia
By design, associations detected by GWASs do not yield a particular gene target or mechanism. This is in contrast to the detection of Mendelian coding mutations in family studies, where the variant, target gene, and mechanism (change in protein) are identified simultaneously. Moreover, the sheer number of associated variants means that the battery of follow-up functional studies traditionally applied to new discoveries from Mendelian disease is not appropriate or achievable for discoveries of genes associated with complex traits. It should be noted that although the effect sizes of individual genetic variants are small in populations, their effect sizes on molecular phenotypes can be large, and the drug effects of gene targets can also be magnified (e.g., statins). Notably, the last 5 years have witnessed some clever laboratory experiments that have followed up on GWAS association, and these have led to the discovery of the target gene, for example, the targets of the associations between FTO (MIM: 610966) and obesity (MIM: 601665)64 and between the major histocompatibility complex (MHC) and schizophrenia.65 A realização de experimentos laboratoriais novos ou semelhantes para muitos loci poderia ser possível, mas seria demorada e cara.
Até recentemente, os esforços para compreender os mecanismos biológicos através dos quais estas diversas variantes de risco agem foram frustrados por limitações na capacidade de realizar avaliações em larga escala do impacto funcional. 66 The advent of sequence-based -omic analyses have been transformative by allowing functional analyses of risk variants to be pursued on the same genome scale (which has fueled their discovery) and allowing mechanistic inferences to be based on the behavior of the full set of risk loci for a given trait.67 The maps of regulatory annotations and connections in disease-relevant tissues, generated by projects such as ENCODE,68 Epigenome RoadMap,69 and GTEx,70 têm sido cruciais para a interpretação das variantes não codificantes que representam a maioria dos alelos de risco identificados pelo GWAS. Os recursos específicos dos tecidos podem tornar-se cada vez mais importantes e, em particular, para as perturbações neuropsiquiátricas, são essenciais recursos cerebrais humanos adequados. Novas iniciativas como CommonMind e PsychENCODE estão a fornecer dados e ferramentas para a comunidade de investigação em neuropsiquiatria acompanhar os sinais do GWAS. Novos métodos analíticos fornecem agora os primeiros passos do acompanhamento funcional in silico, explorando a disponibilidade de conjuntos de dados de recursos que detalham a expressão genética, marcas epigenéticas, contatos de cromatina 3D, 71 ou outras anotações genômicas, incluindo alvos de medicamentos. Uma área fértil de desenvolvimento de métodos é a integração de dados de GWASs e estudos de locus de características quantitativas de expressão (eQTL) para identificar associações entre transcrições e características complexas. 56 , 61 , 62 Esses métodos são úteis para priorizar genes de loci GWAS conhecidos para acompanhamento funcional, detectar novas associações de características genéticas e inferir as direções das associações. 21 , 27 , 62 Os resultados analíticos de que apenas cerca de um terço dos genes associados são os genes mais próximos 61 , 62 são informativos para o projeto de experimentos de mapeamento preciso.
Um dos objetivos finais da pesquisa genética é impulsionar avanços translacionais que permitam uma prevenção e/ou tratamento mais eficaz de doenças. Apesar do inevitável lapso de tempo entre as descobertas da investigação básica e a implementação clínica, um número crescente de exemplos destaca as diversas vias pelas quais a genética humana pode informar a medicina translacional.
Três exemplos de sucesso do GWAS
Here, we focus on three examples of adult-onset disease to demonstrate some of the significant advances that have followed as a direct result of GWASs. Figure 3 illustrates examples of an overlap between GWAS signals that are known drug targets. In general, drug targets that are genetically informed have a higher probability of making it to phase III trial or to market, implying potential huge cost savings to the pharmaceutical industry.72
Figure 3.
Exemplos de ligações entre descobertas do GWAS e drogas
Diabetes tipo 2
Variant and Gene Discovery. Scores of genes have been causally implicated in monogenic forms of diabetes (e.g., neonatal diabetes mellitus [MIM: 601410]73), but GWASs have now identified over 100 common variant signals.74, 75, 76 Recent efforts to extend GWASs beyond array-based genotyping and to access a broader range of variants through sequencing (particularly those of lower frequency) have revealed that most genetic variation influencing T2D appears to reside at common variant sites.74, 77 This chimes with the view of T2D as a largely post-reproductive trait and is consistent with a failure to detect compelling empirical evidence that T2D risk alleles have been subject to marked purifying selection.78, 79 In keeping with the age of these common risk alleles (which predates the diaspora of modern humans out of Africa), most common variant associations for T2D are replicated across major ethnic groups.75, 80 No entanto, à medida que populações cada vez mais diversas são genotipadas e sequenciadas, mais alelos étnicos específicos estão a ser identificados. Vários desses alelos têm um impacto fenotípico relativamente grande e atingiram alta frequência em populações específicas, incluindo variantes em PAX4 (MIM: 167413 ) em asiáticos orientais 74 e TBC1D4 (MIM: 612465 ) em Inuit. 81 Os esforços para identificar evidências convincentes de interações gene-gene e gene-ambiente têm sido em grande parte malsucedidos. 82
Do GWAS à Biologia . Informações regulatórias sobre os principais tecidos da ação da insulina (gordura, músculo e fígado) 82 , 83 e dados equivalentes de material de ilhotas pancreáticas 67 , 84 forneceram evidências convincentes de que as variantes mais fortemente associadas ao DM2 (bem como à glicemia de jejum e outras características quantitativas relacionadas) estão preferencialmente localizadas em intensificadores ativos (particularmente intensificadores de estiramento) em ilhotas pancreáticas 67 , 84 e, em menor grau, em intensificadores ativos na gordura, no músculo e no fígado. 83 , 85 O refinamento crescente da anotação regulatória trouxe uma localização mais precisa desses efeitos regulatórios globais, por exemplo, enfatizando genes de fatores de transcrição específicos (como FOXA2 [MIM: 600288 ]). 86 Esses padrões de enriquecimento genômico específico do tecido estão associados a estudos dos correlatos fisiológicos dos alelos de risco de DM2, conforme observado em dados fisiológicos de indivíduos não diabéticos; estes indicaram que, embora alguns alelos de risco de DM2 tenham um efeito primário na ação da insulina, a maioria parece estar associada à redução da secreção de insulina. 87 Essas abordagens geraram alguns avanços notáveis, por exemplo, o mapeamento da expressão cis destacou o KLF14 (MIM: 609393 ) como o mediador de um sinal T2D do cromossomo 7 que está associado à resistência à insulina e à hiperlipidemia (apropriadamente, esse sinal de expressão é específico do tecido adiposo tecido). 85 Equivalent data from human islets have characterized the likely effector transcripts at several T2D GWAS loci (such as ZMIZ1 [MIM: 607159], MTNR1B [MIM: 600804], and ADCY5 [MIM: 600293]), where the major impact is to reduce insulin secretion.86, 88 Additional clues to the identification of the causal transcripts at certain GWAS loci have come from examining the credentials of the regional transcripts themselves, assigning candidacy on the basis of known biology (e.g., NOTCH2 [MIM: 600275] and GIPR [MIM: 137241]),89 involvement in related monogenic conditions (WFS1 [MIM: 606201], HNF1 [MIM: 142410], and HNF4A [MIM: 600281]),90, 91 or data from animal models (CDKAL1 [MIM: 611259]).92 Finally, the accumulation of data on coding variants (via exome sequencing and/or exome array genotyping) has highlighted several instances where GWAS signals previously attributed to non-coding variants can be reassigned to causal coding variants (e.g., TM6SF2 [MIM: 606563] 74). Para outros, como RREB1 (MIM: 602209 ), a identificação de variantes de codificação associadas a T2D, estatisticamente independentes do sinal GWAS original, sinaliza os prováveis transcritos efetores. 74 Em suma, é possível apontar para uma transcrição efetora convincente em cerca de um terço dos 100 loci T2D identificados pelos GWASs. Esses genes representam alvos legítimos para validação empírica detalhada e exploração mecanicista. Também apoiam esforços, através de abordagens baseadas em redes, para estabelecer até que ponto a biologia da predisposição para DM2 converge para um conjunto restrito de vias.
Tradução . Exemplos de pesquisas sobre DM2 destacam as diversas rotas pelas quais a genética humana pode informar a medicina translacional: (1) a combinação de GWASs de variante comum e ressequenciamento de genes candidatos demonstrou que mutações de perda de função em SLC30A8 (MIM: 611145 ; codificando um transportador de zinco expresso em ilhotas pancreáticas) são protetores para DM2, levando a esforços de várias empresas farmacêuticas para desenvolver antagonistas de ZnT-8; 93 (2) o uso de variantes genéticas como instrumentos que “simulam” a variação nas exposições ambientais e bioquímicas esclareceu até que ponto a ingestão de vitamina D, a nutrição precoce, os níveis lipídicos circulantes e a inflamação crônica desempenham papéis causais no que diz respeito ao desenvolvimento de DM2 94 , 95 , 96 , 97 , 98 e definiu a relação entre a resistência à insulina e a distribuição do tecido adiposo; 99 (3) a identificação de variantes genéticas associadas à variação individual em resposta a agentes terapêuticos comumente utilizados aprimorou nossa compreensão dos mecanismos através dos quais esses agentes operam 100 , 101 e, em alguns casos, levou à otimização terapêutica com base no fenótipo genético e/ou clínico; 102 and (4) the combination of -omic measurements, longitudinal clinical phenotypes, and GWAS data has highlighted sets of molecules (e.g., branched-chain amino acids) that not only are prospectively associated with T2D progression but could also play a causal role in T2D development and thereby provide valuable clinical tools for stratification and prognostication.103, 104
Auto-immune Diseases
Variant and Gene Discovery. In the last 5 years, GWASs have been undertaken for nearly all major immune-mediated diseases (with sample sizes of tens of thousands of case and control individuals for more common immune-mediated diseases studied either by GWASs or by more targeted chips, such as Illumina’s Immunochip105), resultando em centenas de loci associados. O desenvolvimento de abordagens estatísticas para estudos de doenças cruzadas para identificar loci pleiotrópicos tem sido particularmente produtivo na identificação de novos genes e na melhor compreensão da relação patogênica de doenças imunomediadas. Um recente estudo cruzado de doenças envolvendo as condições espondilite anquilosante (EA [MIM: 106300 ]), doença inflamatória intestinal (DII [MIM: 266600 ]), colangite esclerosante primária (MIM: 613806 ) e psoríase identificou sem qualquer genotipagem adicional 30 novos loci com significado em todo o genoma. 24 Estudos transétnicos demonstraram sobreposição genética substancial entre populações etnicamente remotas; 106 , 107 , 108 por exemplo, foram estimadas correlações genéticas de 0,76 e 0,79 entre europeus e asiáticos orientais para a doença de Crohn e a colite ulcerativa (UC). 106 Comparações transétnicas de associações em loci compartilhados têm sido bastante úteis na identificação de variantes causais; por exemplo, a variação específica da população nas HLA-DRB1 (MIM: 142857 associações ) na artrite reumatóide (AR [MIM: 180300 ]) ajudou a definir os principais aminoácidos que sustentam essa associação.
Do GWAS à Biologia . Os resultados do GWAS fizeram contribuições importantes para uma compreensão biológica mais profunda das doenças imunomediadas nos últimos 5 anos. Por exemplo, em um estudo de doenças cruzadas, novos loci incluíram genes que pela primeira vez implicam a patogênese associada à variação de metilação ( DNMT3A [MIM: 602769 ] e DNMT3B [MIM: 602900 ]), genes de detecção de bactérias ( TLR4 [MIM: 603030 ]), genes que influenciam o microbioma hospedeiro ( FUT2 [MIM: 182100 ]) e genes da via NFKB ( NFKB1 [MIM: 164011 ], NFKBIA [MIM: 164008 ], TNFAIP3 [MIM: 191163 ]). 24 A evidência de pleiotropia extensa inclui variantes que apresentam diferentes direções de associação em diferentes doenças ou variantes específicas de doenças nos mesmos loci. Isto tem relevância para o provável impacto do direcionamento terapêutico desses loci. Por exemplo, o SNP rs1800693 no principal gene do receptor de TNF TNFR1 (MIM: 191190 ) está associado em diferentes direções à esclerose múltipla (MS [MIM: 126200 ]) e EA. 109 O SNP leva à perda do domínio transmembrana do receptor, e o SNP de risco para EM (protetor para EA) leva ao aumento do receptor de TNF solúvel no soro. 110 A inibição do TNF, incluindo terapias com receptores chamariz de TNF, é altamente eficaz para EA e muitas outras doenças imunomediadas, mas seu uso pode ser complicado pelo desenvolvimento de novo da EM e, na própria EM, pode exacerbar a doença. Embora este seja um exemplo retrospectivo, demonstra o potencial do uso da genética para prever toxicidades. Existem vários agentes em desenvolvimento onde a genética apontaria para a probabilidade de toxicidades. Os exemplos incluem CD40 e seu ligante, onde o SNP rs1883832 no alelo C de CD40 (MIM: 109535 ) é um fator de risco para AR e distúrbio autoimune da tireoide (AITD), mas é protetor contra EM e DII, e o PTPN22 (MIM: 600716 ) variante c.1858C>T (p.Arg620Trp), que aumenta o risco de diabetes tipo 1 (MIM: 222100 ), lúpus eritematoso sistêmico (MIM: 152700 ), vitiligo (MIM: 606579 ), AITD e UC, mas é protetor contra a doença de Crohn. 23 No mínimo, isto sugere que quaisquer ensaios clínicos nestas condições devem examinar cuidadosamente o desenvolvimento das doenças com as associações genéticas inversas.
O MHC, bem como os genes HLA nele codificados, é o principal locus para a maioria das doenças imunomediadas. Embora os principais tipos de HLA altamente penetrantes envolvidos em diferentes doenças tenham sido estabelecidos há muito tempo, nos últimos 5 anos, a capacidade de imputar os aminoácidos compostos e depois testá-los quanto à associação de doenças permitiu pesquisas que definiram melhor os componentes-chave do HLA. proteínas envolvidas em doenças. Na AR, sabia-se há cerca de 30 anos que uma sequência de aminoácidos nas posições 70-74 do HLA-DRB1 determina em grande parte, embora não completamente, a associação diferencial entre os tipos de HLA-DRB1 e a doença. 111 Através do uso de imputação de aminoácidos e estudos de associação, esta sequência de “epítopo compartilhado” foi estendida, 112 e esta informação é usada para fornecer uma explicação molecular para a propensão dos peptídeos com aminoácidos componentes citrulinados para induzir doenças. 113 Há muito se sabe que as variantes HLA são os principais determinantes de reações adversas medicamentosas imunologicamente mediadas graves. Por exemplo, a toxicidade do anti-retroviral abacavir está largamente restrita aos portadores do HLA-B57. Com o uso de imputação de GWAS e HLA, um HLA-DQA1 ∗ 0201-HLA-DRB1 ∗ 0701 está fortemente associado ao risco de pancreatite induzida por tiopurina, de modo que os homozigotos para este haplótipo têm um risco de 17% deste efeito colateral importante. Foi demonstrado que o haplótipo 114 É provável que, com o uso crescente do perfil genético na prática clínica, sejam identificados mais exemplos nos próximos anos.
Translation. GWAS results have already proven highly successful at initiating medication repositioning. For example, GWAS discoveries triggered the repositioning of biological medications targeting components of the IL-23 pathway (including IL-12p40, IL-17, and IL-23p19), and now these are mainstay treatments for psoriasis and psoriatic arthritis (MIM: 607507), are highly effective in AS, and (with the exception of IL-17 blockade) are effective in IBD, as suggested by early studies.115, 116, 117 The annual sales of these medications alone are likely to be greater than the total amount spent on GWASs in the past decade.
Muitas outras descobertas do GWAS estimularam programas direcionados de desenvolvimento de terapia, alguns dos quais são descritos aqui. A descoberta da associação entre PADI4 (MIM: 605347 ) e AR forneceu evidências conclusivas de que as reações imunológicas a epítopos que foram citrulinados pelas enzimas PAD estavam causalmente envolvidas na AR. Isto levou ao desenvolvimento de programas de inibidores de PAD na AR, e estes têm se mostrado promissores. 118, 119 Principais programas de desenvolvimento de medicamentos foram iniciados para atingir os genes da aminopepidase M1 ERAP1 (MIM: 606832 ) e ERAP2 (MIM: 609497 ) devido às suas associações genéticas com EA, psoríase, DII, doença de Behcet (MIM: 109650 ) e o raro condição retinopatia de Birdshot (MIM: 605808 ). 120
O acompanhamento bioinformático dos resultados do GWAS também tem sido frutífero. Por exemplo, Okada et al. examinaram a sobreposição entre associações genéticas e alvos de medicamentos conhecidos para demonstrar que as terapias existentes para AR têm como alvo desproporcional os produtos genéticos associados à AR e seus parceiros proteicos em interação. 108 A partir disto, extrapolaram que outros agentes com elevados níveis de efeitos sobre estas proteínas seriam enriquecidos com potenciais novas terapias para AR e forneceram evidências sugestivas de que os inibidores de CDK4 e CDK6 já em uso, particularmente em oncologia, poderiam ser eficazes na AR. Esses agentes demonstraram ser eficazes no modelo de artrite induzida por colágeno da AR e estão agora em testes em humanos na AR no Japão.
Esquizofrenia
Variante e descoberta de genes. Embora as doenças psiquiátricas tenham tido um início lento na identificação do locus GWAS, mais de 50.000 amostras foram genotipadas nos últimos 5 anos; a relação linear típica entre o tamanho da amostra e o número de loci foi observada, e mais de 100 loci de risco foram descobertos até o momento. Esses loci de risco são enriquecidos em genes contendo mutações de novo na esquizofrenia, autismo (MIM: 209850 ) e deficiência intelectual, 15 and several identified loci contain genes relevant to major hypotheses of schizophrenia etiology, including DRD2 (MIM: 126450; the target of anti-psychotic drugs) and genes involved in glutamatergic neurotransmission (GRM3 [MIM: 601115], GRIN2A [MIM: 138253], and GRIA1 [MIM: 138248]), as well as genes that extend previous observations of association with voltage-gated calcium channel subunits (CACNA1C [MIM: 114205], CACNB2 [MIM: 600003], and CACNA1I [MIM: 608230]).15 One of the most striking findings that emerged early in schizophrenia studies—at the stage where there were only a handful of genome-wide-significant loci—was the highly polygenic nature of the common variants contributing to risk.14 This observation has been widely replicated, and estimates are that 71% of 1 Mb genomic regions have at least one variant influencing schizophrenia risk,53 and there is evidence of substantial pleiotropy with other psychiatric disorders.26 No entanto, a arquitetura genética, descrita como a mistura de variantes raras e comuns, provavelmente diferirá entre os transtornos psiquiátricos, como já está sendo observado para as taxas mais altas de CNVs raras, penetrantes de novo e variantes de nucleotídeo único encontradas no autismo do que na esquizofrenia. ou transtorno bipolar. 121 , 122 , 123 , 124 , 125 , 126 , 127 , 128 , 129 Os estudos PRS estão sendo amplamente utilizados para investigar a heterogeneidade da doença e as contribuições dos fatores de risco ambientais.
Do GWAS à Biologia. O acompanhamento funcional é necessariamente mais difícil para transtornos psiquiátricos e, até o momento, as análises bioinformáticas têm sido o foco principal no fornecimento de estratégias para priorização de loci. Os loci de risco de esquizofrenia estão sobre-representados em regiões reguladoras ativas no cérebro 15 , 130 , 131 e são enriquecidos em genes de densidade pós-sináptica, membrana pós-sináptica, espinha dendrítica, axônio e canais de potássio dependentes de voltagem, bem como metilação de histonas H3-K4 132 sobrepõem-se a caminhos identificados em estudos de variantes raras do autismo. A priorização dos resultados do GWAS progrediu através da integração com conjuntos de dados eQTL, implicando genes sinápticos ( SNAP91 [MIM: 607923 ], TSNARE1 e CLCN4 [MIM 302910 ]) e genes com funções no neurodesenvolvimento ( FURIN [MIM: 136950 ] e CNTN4 [MIM: 607280 ]). Contatos 3D entre variantes de risco e promotores, explorados pela captura de conformação cromossômica (Hi-C) na placa subcortical e zona germinativa do córtex humano em desenvolvimento, apoiaram supostas interações entre variantes de risco causais e promotores em genes de sinalização glutamatérgicos e de cálcio ( GRIA1 [MIM : 138248 ], NLGN1 [MIM: 600568 ], GRIN2A [MIM: 138253 ] e CACNA1C [MIM: 114205 ]), em vários genes há muito implicados na esquizofrenia (incluindo DRD2 e DRD6 , que codificam subunidades de receptores de acetilcolina) e nos genes SNAP91 , TSNARE1 , CLCN4 , FURIN e CNTN4 . 71
O mapeamento preciso foi realizado para a associação mais forte e identificada pela primeira vez com a esquizofrenia na região do MHC, um desafio devido ao seu alto conteúdo gênico e alto LD. A posição do sinal de associação dentro da região do MHC levou à investigação de haplótipos estruturais comuns dos genes do fator 4 do complemento C4A (MIM: 120810 ) e C4B (MIM: 120820 ), combinações dos quais se correlacionaram bem com o risco de esquizofrenia e aumentaram a expressão de C4 e mostraram expressão cerebral diferencial entre indivíduos caso e controle. 65 Several other complement proteins play a role in synapse elimination, and decreased numbers of synapses have long been suggested as a primary abnormality in schizophrenia. Observations that, in mice, a complement gene that shares features with human C4A and C4B is expressed in neurons and promoted synapse elimination in a developmental brain circuit strongly implicate this gene and its protein.
Tradução . Nenhum novo alvo molecular para a esquizofrenia foi identificado com sucesso desde que os primeiros medicamentos antipsicóticos foram identificados há várias décadas. As razões são provavelmente múltiplas, mas a maior parte do desenvolvimento de medicamentos para a esquizofrenia concentrou-se na obtenção de medicamentos de alta potência para um único alvo – uma metodologia bem-sucedida em muitas outras áreas da medicina – o que exige uma escolha entre as hipóteses concorrentes da fisiopatologia da esquizofrenia. Os resultados do GWAS forneceram evidências inequívocas de poligenicidade e, como muitos dos loci GWAS contêm genes que codificam proteínas entre aquelas indicadas por múltiplas hipóteses anteriores, por exemplo, dopamina, glutamato, modulação imunológica, sinalização de cálcio e colinérgico nicotínico, o desenvolvimento futuro de medicamentos poderia beneficiar de uma abordagem multi-alvo. Um enriquecimento de conjunto de genes de prova de conceito de alelos de risco de esquizofrenia em conjuntos de genes para alvos de medicamentos identificou várias oportunidades potenciais de reaproveitamento. 133 Medicamentos de alvo único podem ser apropriados para subgrupos genéticos específicos, embora a identificação de subtipos genéticos ainda não faça parte do paradigma dos ensaios clínicos.
Discussão
O Presente
We have summarized the major kinds of discoveries made from GWASs focusing on adult traits and have reviewed the new biology and emerging translational outcomes for three diseases. Over the last decade, this experimental design has delivered a remarkably diverse set of discoveries in human genetics. For most traits and diseases studied, the mutational target in the genome appears large, in that polymorphisms in many genes contribute genetic variation in the population. Furthermore, the empirical evidence of widespread pleiotropy implies that many segregating variants affect multiple traits. A precise estimate of the proportion of all segregating genetic variants that are “functional” in the context of being associated with one or more traits, conditional on all other causal variants, remains elusive. For the highlighted traits, disorders, and diseases, we have given examples of routes from GWAS to biology and translation. For an experimental design only a decade old, this is an example of rapid translation of genetic findings toward clinical application.
A relação entre o tamanho da amostra e o número de loci de risco detectados varia entre as características, mas todas mostram um aumento acentuado em um tamanho de amostra crítico. Até o momento, não houve nenhuma característica com evidência de um platô no número de loci de risco descobertos com o aumento do tamanho da amostra. Para algumas características, como altura, esquizofrenia e DII, as amostras descobertas nos próximos 5 anos provavelmente continuarão a aumentar, talvez a uma taxa mais baixa por amostra adicional. Uma taxa decrescente de descoberta de novos loci fornecerá um quadro mais completo da arquitetura genética e satisfará melhor a compreensão das vias biológicas contribuintes. De acordo com o conhecimento da doença mendeliana, a expectativa é que múltiplas variantes de risco sejam detectadas em loci já identificados. Assim, à medida que o tamanho das amostras aumenta, as novas descobertas de vias associadas serão saturadas primeiro, seguidas pelos genes e, por último, pelas variantes.
Os GWASs foram aplicados com sucesso a características moleculares como expressão gênica, metilação do DNA, 134 e metabólitos. 135 As conclusões destes estudos são que a maioria dos fenótipos moleculares são iguais a outras características complexas, na medida em que as diferenças entre os indivíduos são devidas a uma combinação de factores genéticos e exposições ambientais e que os loci genéticos podem ser mapeados por GWASs. 136 Isto torna a descoberta de caminhos causais desde genomas até fenómenos um desafio, na medida em que a variação entre pessoas em factores de risco modificáveis pode estar parcialmente ancorada na variação da sequência de ADN para estas “exposições”. No entanto a combinação da variação de sequências com fenótipos moleculares e dados de doenças com novos métodos analíticos como a randomização mendeliana 42 has great potential to unravel cause and consequence and to improve phenotypic prediction.137
Até o momento, os GWAS foram baseados em matrizes SNP projetadas para marcar variantes comuns no genoma. Estas matrizes não cobrem todas as variantes genéticas da população e parece natural que os futuros GWAS sejam baseados no WGS. No entanto, o diferencial de preço entre os arrays SNP e o WGS ainda é substancial, e a tecnologia dos arrays permanece mais robusta do que o sequenciamento. No entanto, agora centenas de milhares de genomas estão a ser sequenciados como parte de grandes iniciativas, e os próximos 5 anos permitirão comparações directas de descobertas feitas a partir de estudos de sequenciação e arranjos. Curiosamente, matrizes personalizadas sem uma “espinha dorsal” de GWAS (como Immunochip, Metabochip e matrizes somente exoma) em geral falharam em identificar variantes raras (MAF <1%) em loci que foram inicialmente descobertos a partir de um GWAS, um dos seus objetivos. A razão para isso não está clara. Pode ser porque não existem variantes raras de efeito principal, porque o tamanho da amostra é demasiado pequeno para detectar variantes raras e/ou estimar o seu tamanho de efeito, porque a cobertura do chip de variantes raras é inadequada, ou devido a uma combinação destas. No entanto, essas matrizes personalizadas levaram à descoberta de novos loci e ao mapeamento preciso dos loci existentes, impulsionados principalmente pelo aumento do tamanho da amostra experimental (ver Apêndice A sobre a relação entre tamanho da amostra, precisão de imputação e frequência alélica no poder de detecção). Um estudo recente de altura usando matrizes exoma SNP e um tamanho de amostra de 700.000 relatou 83 variantes de codificação associadas à altura com uma frequência inferior a 5% e tamanhos de efeito de até 2 cm. 138 Cada uma dessas variantes explica, em média, aproximadamente a mesma quantidade de variação que as variantes comuns, cujos tamanhos de efeito são da ordem de 1 mm, porque é a combinação de frequência e tamanho de efeito que determina a variação ( Apêndice A ).
Uma limitação do array atual e da tecnologia WGS é que a precisão da detecção de variantes estruturais (indels ou inversões> 50 pb) é menor que a da detecção de SNP. As novas tecnologias que permitem a haplotipagem de longo alcance estão a ajudar a superar a fraqueza das tecnologias de leitura curta, e tecnologias baratas que abrangem todo o genoma para variantes estruturais constituiriam um avanço importante.
O mapeamento fino de associações de características SNP é a tentativa de identificar uma ou mais variantes causais que são responsáveis pelos sinais GWAS observados. O mapeamento fino apenas por associação estatística é limitado pelo tamanho da amostra experimental e LD, dado que a evidência estatística para separar uma variante causal de uma variante em LD com ela é proporcional a n(1 – r 2 ) (ver Equação A1 no Apêndice A ). Se as variantes causais não estiverem nos dados (por exemplo, não foram genotipadas), então o erro de imputação também limita o mapeamento preciso. Com a provável disponibilidade de dados GWAS baseados em array SNP em tamanhos de amostra muito grandes e dados WGS em tamanhos de amostra grandes, o poder de mapeamento preciso estatístico melhorará, e um pequeno número de variantes que estão em LD extremamente alto podem ser identificadas como um conjunto plausível de variantes com alta probabilidade de conter uma ou mais variantes causais. A utilização de informações adicionais, tais como o conhecimento prévio da função provável de variantes específicas, dada a sua localização e o(s) motivo(s) de ADN circundante(s), 139, 140 could help to reduce the set of statistical candidates to a smaller number. This is already a fertile area of statistical and bioinformatic research56, 62, 131, 141, 142 reunir resultados de características ou doenças de GWAS com aqueles de expressão gênica tecidual. São necessárias mais pesquisas sobre a resolução do mapeamento fino, e isso será alimentado por um aumento esperado nos dados do GWAS sobre a expressão gênica específica de tecidos e células.
A maioria dos GWAS até à data foram realizados em indivíduos de ascendência europeia, mas há um número crescente de estudos sobre populações de ascendência asiática e africana. Como as variantes comuns contribuem para a arquitetura genética de características complexas, a expectativa é que essas variantes comuns sejam evolutivamente antigas e compartilhadas entre etnias, o que é encorajador para a generalização dos tratamentos. A demonstração mais clara disto, conforme discutido acima, foi no caso da DII, para a qual a correlação genética entre amostras asiáticas e europeias é próxima de 0,8, embora alguns loci de risco individuais difiram em frequência ou tamanho do efeito. 106 Uma característica das análises GWAS até à data é excluir estritamente indivíduos fora dos limites étnicos com base em unidades de desvio padrão nas dimensões dos componentes principais. No entanto, à medida que os tamanhos das amostras aumentam, torna-se possível não só utilizar, mas também tirar partido de etnias mistas e mistas. As diferentes frequências alélicas e estrutura de LD entre as populações devem ajudar no mapeamento preciso das variantes causais. Novos métodos surgiram para lidar com esses dados, 75 , 143 e esperamos que esta seja uma área fértil de desenvolvimento e descoberta de métodos nos próximos anos.
O Futuro
O GWAS tem futuro? Extrapolando as descobertas dos últimos 10 anos para o futuro, se mantivéssemos a atual estratégia experimental de matrizes e imputação de SNP, então tamanhos de amostra cada vez maiores levariam, sem dúvida, a novas descobertas genéticas, particularmente (1) a descoberta de mais variantes e mais genes associados a uma ou mais características, (2) contabilizando mais variação genética, (3) preditores genéticos mais precisos e (4) uma maior capacidade de avaliar a heterogeneidade da doença e de derivar diagnósticos geneticamente informados que possam estar mais alinhado a tratamentos específicos. Para análises de enriquecimento biológico e a descoberta ou ajuste fino de caminhos envolvidos em características quantitativas e doenças, é provável que mais loci aumentem a resolução. Em campos onde os critérios diagnósticos não são baseados em marcadores biológicos, como a psiquiatria, os GWAS viraram o campo de cabeça para baixo, pela primeira vez, contribuindo com dados quantitativos que podem ser usados para reavaliar completamente a relação de distúrbios anteriormente distintos.
O futuro dos GWAS terá desafios novos e antigos. Com estudos cada vez maiores, os novos loci identificados normalmente terão tamanhos de efeito menores (por exemplo, menos de 0,5 mm para uma característica como altura e uma razão de chances de 1,01 para doenças comuns) ou, para variantes raras, terão níveis muito baixos. freqüência. 138 For disorders with population prevalence of the order of 0.1%, discovery will still be limited by experimental sample size, given that it will take many years to accumulate sample sizes of 100,000 cases or more. One challenge is how such loci can be fine-mapped or studied for mechanism. Upscaling of technology, either through interfacing with sequenced-based -omic data or through upscaling by experimental perturbations (e.g., multiple-locus or genome-wide CRISPR) are likely to be key to overcoming the challenges of small effect size. What is likely to change in the near future is that GWASs by SNP arrays will be gradually replaced by GWASs by WGS, particularly for quantitative traits and very common diseases. Nonetheless, given a finite budget, larger sample sizes that are phenotypically more informative and genotyped on a SNP chip remain a powerful strategy for maximizing discovery. Fifteen years ago, genotyping technology was the limiting step to discovery, but now discovery is limited by phenotypic descriptors that could link with genetic data to allow disease stratification that might be more aligned with treatments. Furthermore, the emphasis in research will need to shift from gene discovery to translation into biological understanding and patient-focused outcomes, such as better diagnostic tests and novel treatments.
Em conclusão, o desenho experimental dos GWAS levou a uma notável gama de descobertas na genética humana na última década. Cumpriu o seu objectivo original de detectar associações entre variantes comuns de ADN e doenças e distúrbios humanos. Isso levou a uma melhor compreensão da arquitetura genética de características complexas e, portanto, da seleção natural passada em características associadas à aptidão. Isso levou à descoberta de variantes, genes e vias biológicas que desempenham um papel em doenças e distúrbios específicos. Isso levou a novas descobertas na epidemiologia de doenças e à descoberta ou reaproveitamento de possíveis terapêuticas. Tal como previsto em 2007, foi de facto um caso de beber da mangueira de incêndio. 144 Para o futuro, a combinação de pesquisas de variação genética de todo o genoma e dados fenotípicos e ómicos detalhados sobre milhões de indivíduos será um tesouro para fazer novas descobertas fundamentais na genética humana. Algumas dessas descobertas serão totalmente inesperadas e outras detectarão ou desvendarão mecanismos biológicos. As descobertas específicas de doenças continuarão a estimular o desenvolvimento e os ensaios de novas terapêuticas, a compreensão dos caminhos desde a sequência até à consequência e, para algumas doenças, a prevenção ou intervenção precoce. Daqui a 10 anos, a medicina genômica “personalizada” ou de “precisão” provavelmente será difundida e incluirá algumas aplicações para doenças comuns, seja diretamente através da estratificação de risco para prevenção direcionada ou estratégias de intervenção ou indiretamente através de novos tratamentos onde os resultados do GWAS fornecem o primeiro etapa no pipeline de descoberta. O projeto experimental de GWASs, que começou como um exercício teórico há mais de 20 anos, 145 amadureceu e entregou.
Acknowledgments
Esta pesquisa foi apoiada pelo Conselho Nacional Australiano de Saúde e Pesquisa Médica (1107258, 1078901, 1078037, 1056929, 1048853 e 1113400), pela gs2:Sylvia and Charles Viertel Charitable Foundation, pelo NIH (GM099568, MH100141-01, MH095034, MH109897, DK098032, DK085545, MH101814 e DKD105535), o Wellcome Trust (090532, 090367, 098381 e 106130), o Conselho de Pesquisa Médica do Reino Unido (L020149, 004422 e J010642), a Innovative Medicines Initiative e a Accelerating Medicines Partnership (através da Foundation for the NIH). MIM é um investigador sênior do Wellcome Trust. Agradecemos aos revisores por seus muitos comentários úteis, que melhoraram o artigo. Devido às limitações de espaço, não pudemos fazer justiça completa a todos os artigos relevantes e importantes do GWAS que foram publicados nos últimos 10 anos. Pedimos desculpas a muitos de nossos colegas cujo trabalho não é citado.
Apêndice A: Poder Experimental para Detectar Associação
Revisitamos o poder estatístico porque a interação do tamanho da amostra experimental, frequência da variante causal e tamanho do efeito e plataforma (genótipos, genótipos imputados e sequência do genoma completo) permanece essencial para julgar o projeto experimental ideal para descoberta. O poder de detectar uma associação variante-característica de LD entre uma variante causal não observada e um genótipo observado pode ser quantificado no parâmetro de não centralidade (NCP) de um teste estatístico para detectar associação (ou seja, o valor esperado da estatística de teste sob a hipótese alternativa). É definido como
| NCP=n×r2×q2/(1−r2q2), | (Equation A1) |
where n is the experimental sample size, q2 is the proportion of phenotypic variance explained by a causal variant in the population, and r2 is the squared LD correlation between the causal variant and a genotyped SNP. This can also be expressed as the proportion of variance explained by the genotyped SNP in the population (R2 = r2q2),
| NCP=n×R2/(1−R2). | (Equation A2) |
If the genotypes at the causal locus are in Hardy-Weinberg equilibrium, then
| q2=2×MAF×(1−MAF)×β2, | (Equation A3) |
onde β é o tamanho do efeito de um alelo no fenótipo em unidades de desvio padrão. Isto pressupõe que a análise para detectar uma associação é feita por regressão do fenótipo na contagem de genótipos (por exemplo, zero, um ou dois alelos menores). Portanto, quando q 2 é pequeno,
| NCP=n×r2×2×MAF×(1−MAF)×β2. | (Equação A4) |
O poder de detectar uma associação entre uma característica e uma variante causal não genotipada, mas imputada, é, da mesma forma,
| NCP=n×R2imp×2×MAF×(1−MAF)×β2, | (Equação A5) |
onde R imp 2 é a correlação quadrática entre os genótipos reais e imputados no locus. Esses cálculos de poder ilustram a compensação entre tamanho da amostra, frequência alélica e tamanho do efeito. No futuro, quando os GWAS forem provavelmente realizados pela WGS, o r 2 e Rimp 2 os valores nas Equações A4 e A5 serão 1 se a variante causal for sequenciada sem erro, e um poder considerável pode ser obtido para detectar a associação entre uma característica e uma variante sequenciada em comparação com dados genotipados ou imputados baseados em array se r 2 ou R imp 2 é pequeno a modesto, mas apenas quando o tamanho da amostra experimental é mantido constante. A equação acima também demonstra que o poder de detectar a associação para uma variante rara é limitado devido à sua baixa frequência alélica, mesmo que o tamanho do efeito seja maior do que o de uma variante comum. Isto significa que para associações de variantes raras, os tamanhos das amostras precisam ser muito grandes ou pelo menos comparáveis aos usados para GWASs com variantes comuns, mesmo com dados WGS.
Web Resources
GWAS Catalog, https://www.ebi.ac.uk/gwas/
OMIM, http://www.omim.org/
Referências
- 1. Visscher PM, Brown MA, McCarthy MI, Yang J. Cinco anos de descoberta do GWAS. Sou. J. Hum. Geneta. 2012;90:7–24. doi: 10.1016/j.ajhg.2011.11.029. [ DOI ] [ artigo gratuito do PMC ] [ PubMed ] [ Google Scholar ]
- 2. Zhang F., Gu W., Hurles ME, Lupski JR Copie a variação do número na saúde, doença e evolução humana. Anu. Rev. Genômica Hum. Geneta. 2009;10:451–481. doi: 10.1146/annurev.genom.9.081307.164217. [ DOI ] [ artigo gratuito do PMC ] [ PubMed ] [ Google Scholar ]
- 3. Propriedades de Hudson RR de um modelo de alelo neutro com recombinação intragênica. Teoria. População. Biol. 1983;23:183–201. doi: 10.1016/0040-5809(83)90013-8. [ DOI ] [ PubMed ] [ Google Scholar ]
- 4. Frequências do alelo Wray NR e medida r2 do desequilíbrio de ligação: impacto no desenho e interpretação de estudos de associação. Res. Gêmea. Zumbir. Geneta. 2005;8:87–94. doi: 10.1375/1832427053738827. [ DOI ] [ PubMed ] [ Google Scholar ]
- 5. Browning SR, Browning BL Faseamento rápido e preciso de haplótipos e inferência de dados faltantes para estudos de associação de genoma completo por meio de agrupamento de haplótipos localizado. Sou. J. Hum. Geneta. 2007;81:1084–1097. doi: 10.1086/521987. [ DOI ] [ artigo gratuito do PMC ] [ PubMed ] [ Google Scholar ]
- 6. Li Y., Willer CJ, Ding J., Scheet P., Abecasis GR MaCH: usando dados de sequência e genótipo para estimar haplótipos e genótipos não observados. Geneta. Epidemiol. 2010;34:816–834. doi: 10.1002/gepi.20533. [ DOI ] [ artigo gratuito do PMC ] [ PubMed ] [ Google Scholar ]
- 7.Marchini J., Howie B., Myers S., McVean G., Donnelly P. A new multipoint method for genome-wide association studies by imputation of genotypes. Nat. Genet. 2007;39:906–913. doi: 10.1038/ng2088. [DOI] [PubMed] [Google Scholar]
- 8.McCarthy S., Das S., Kretzschmar W., Delaneau O., Wood A.R., Teumer A., Kang H.M., Fuchsberger C., Danecek P., Sharp K. A reference panel of 64,976 haplotypes for genotype imputation. Nat. Genet. 2016;48:1279–1283. doi: 10.1038/ng.3643. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 9.Yang J., Wray N.R., Visscher P.M. Comparing apples and oranges: equating the power of case-control and quantitative trait association studies. Genet. Epidemiol. 2010;34:254–257. doi: 10.1002/gepi.20456. [DOI] [PubMed] [Google Scholar]
- 10. Welter D., MacArthur J., Morales J., Burdett T., Hall P., Junkins H., Klemm A., Flicek P., Manolio T., Hindorff L., Parkinson H. O Catálogo NHGRI GWAS, um recurso com curadoria de associações de características SNP. Ácidos Nucleicos Res. 2014;42:D1001–D1006. doi: 10.1093/nar/gkt1229. [ DOI ] [ artigo gratuito do PMC ] [ PubMed ] [ Google Scholar ]
- 11. Torgerson DG, Ampleford EJ, Chiu GY, Gauderman WJ, Gignoux CR, Graves PE, Himes BE, Levin AM, Mathias RA, Hancock DB Meta-análise de estudos de associação genômica ampla de asma em populações norte-americanas etnicamente diversas. Nat. Geneta. 2011;43:887–892. doi: 10.1038/ng.888. [ DOI ] [ artigo gratuito do PMC ] [ PubMed ] [ Google Scholar ]
- 12. Marigorta UM, Navarro A. A alta replicabilidade transétnica dos resultados do GWAS implica variantes causais comuns. PLoS Genet. 2013;9:e1003566. doi: 10.1371/journal.pgen.1003566. [ DOI ] [ artigo gratuito do PMC ] [ PubMed ] [ Google Scholar ]
- 13. Wray NR, Goddard ME, Visscher PM Previsão do risco genético individual para doenças a partir de estudos de associação genômica ampla. Genoma Res. 2007;17:1520–1528. doi: 10.1101/gr.6665407. [ DOI ] [ artigo gratuito do PMC ] [ PubMed ] [ Google Scholar ]
- 14. Consórcio Internacional de Esquizofrenia A variação poligênica comum contribui para o risco de esquizofrenia e transtorno bipolar. Natureza. 2009;460:748–752. doi: 10.1038/nature08185. [ DOI ] [ artigo gratuito do PMC ] [ PubMed ] [ Google Scholar ]
- 15. Grupo de Trabalho sobre Esquizofrenia do Consórcio de Genômica Psiquiátrica Informações biológicas de 108 loci genéticos associados à esquizofrenia. Natureza. 2014;511:421–427. doi: 10.1038/nature13595. [ DOI ] [ artigo gratuito do PMC ] [ PubMed ] [ Google Scholar ]
- 16.Maher B. Personal genomes: The case of the missing heritability. Nature. 2008;456:18–21. doi: 10.1038/456018a. [DOI] [PubMed] [Google Scholar]
- 17.Manolio T.A., Collins F.S., Cox N.J., Goldstein D.B., Hindorff L.A., Hunter D.J., McCarthy M.I., Ramos E.M., Cardon L.R., Chakravarti A. Finding the missing heritability of complex diseases. Nature. 2009;461:747–753. doi: 10.1038/nature08494. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 18.Wood A.R., Esko T., Yang J., Vedantam S., Pers T.H., Gustafsson S., Chu A.Y., Estrada K., Luan J., Kutalik Z. Defining the role of common variation in the genomic and biological architecture of adult human height. Nat. Genet. 2014;46:1173–1186. doi: 10.1038/ng.3097. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 19.Lynch M., Walsh B. Sinauer Associates; 1998. Genetics and analysis of quantitative traits. [Google Scholar]
- 20. Bulik-Sullivan B., Finucane HK, Anttila V., Gusev A., Day FR, Loh PR, Consórcio ReproGen. Consórcio de Genômica Psiquiátrica. Consórcio Genético para Anorexia Nervosa do Wellcome Trust Case Control Consortium 3. Duncan L. Um atlas de correlações genéticas entre doenças e características humanas. Nat. Geneta. 2015;47:1236–1241. doi: 10.1038/ng.3406. [ DOI ] [ artigo gratuito do PMC ] [ PubMed ] [ Google Scholar ]
- 21. Pickrell JK, Berisa T., Liu JZ, Ségurel L., Tung JY, Hinds DA Detecção e interpretação de influências genéticas compartilhadas em 42 características humanas. Nat. Geneta. 2016;48:709–717. doi: 10.1038/ng.3570. [ DOI ] [ artigo gratuito do PMC ] [ PubMed ] [ Google Scholar ]
- [Artigo gratuito do PMC] [PubMed] 22. Sivakumaran S, Agakov F, Theodoratou E, Prendergast JG, Zgaga L, Manolio T, Rudan I, McKeigue P, Wilson JF, Campbell H. Pleiotropia abundante em doenças e características complexas humanas. Sou. J. Hum. Geneta. 2011;89:607–618. doi: 10.1016/j.ajhg.2011.10.004. [ DOI ] [ artigo gratuito do PMC ] [ PubMed ] [ Google Scholar
- 23. Parkes M., Cortes A., van Heel DA, Brown MA Insights genéticos sobre vias comuns e relações complexas entre doenças imunomediadas. Nat. Rev. 2013;14:661–673. doi: 10.1038/nrg3502. [ DOI ] [ PubMed ] [ Google Scholar ]
- 24. Ellinghaus D., Jostins L., Espanha SL, Cortes A., Bethune J., Han B., Park YR, Raychaudhuri S., Pouget JG, Hübenthal M. Análise de cinco doenças inflamatórias crônicas identifica 27 novas associações e destaques padrões específicos da doença em loci compartilhados. Nat. Geneta. 2016;48:510–518. doi: 10.1038/ng.3528. [ DOI ] [ artigo gratuito do PMC ] [ PubMed ] [ Google Scholar ]
- 25.Li Y.R., Zhao S.D., Li J., Bradfield J.P., Mohebnasab M., Steel L., Kobie J., Abrams D.J., Mentch F.D., Glessner J.T. Genetic sharing and heritability of paediatric age of onset autoimmune diseases. Nat. Commun. 2015;6:8442. doi: 10.1038/ncomms9442. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 26.Cross-Disorder Group of the Psychiatric Genomics Consortium Genetic relationship between five psychiatric disorders estimated from genome-wide SNPs. Nat. Genet. 2013;45:984–994. doi: 10.1038/ng.2711. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 27.Visscher P.M., Yang J. A plethora of pleiotropy across complex traits. Nat. Genet. 2016;48:707–708. doi: 10.1038/ng.3604. [DOI] [PubMed] [Google Scholar]
- 28.Yu J., Pressoir G., Briggs W.H., Vroh Bi I., Yamasaki M., Doebley J.F., McMullen M.D., Gaut B.S., Nielsen D.M., Holland J.B. A unified mixed-model method for association mapping that accounts for multiple levels of relatedness. Nat. Genet. 2006;38:203–208. doi: 10.1038/ng1702. [DOI] [PubMed] [Google Scholar]
- 29. Kang HM, Sul JH, Service SK, Zaitlen NA, Kong SY, Freimer NB, Sabatti C., Eskin E. Modelo de componente de variância para explicar a estrutura da amostra em estudos de associação genômica ampla. Nat. Geneta. 2010;42:348–354. doi: 10.1038/ng.548. [ DOI ] [ artigo gratuito do PMC ] [ PubMed ] [ Google Scholar ]
- 30. Lippert C., Listgarten J., Liu Y., Kadie CM, Davidson RI, Heckerman D. Modelos mistos lineares FaST para estudos de associação genômica ampla. Nat. Métodos. 2011;8:833–835. doi: 10.1038/nmeth.1681. [ DOI ] [ PubMed ] [ Google Scholar ]
- 31. Zhou X., Stephens M. Análise eficiente de modelo misto em todo o genoma para estudos de associação. Nat. Geneta. 2012;44:821–824. doi: 10.1038/ng.2310. [ DOI ] [ artigo gratuito do PMC ] [ PubMed ] [ Google Scholar ]
- 32. Svishcheva GR, Axenovich TI, Belonogova NM, van Duijn CM, Aulchenko YS Método baseado em componentes de variância rápida para análise de associação do genoma completo. Nat. Geneta. 2012;44:1166–1170. doi: 10.1038/ng.2410. [ DOI ] [ PubMed ] [ Google Scholar ]
- 33. Yang J., Zaitlen NA, Goddard ME, Visscher PM, Price AL Vantagens e armadilhas na aplicação de métodos de associação de modelos mistos. Nat. Geneta. 2014;46:100–106. doi: 10.1038/ng.2876. [ DOI ] [ artigo gratuito do PMC ] [ PubMed ] [ Google Scholar ]
- 34.Loh P.R., Tucker G., Bulik-Sullivan B.K., Vilhjálmsson B.J., Finucane H.K., Salem R.M., Chasman D.I., Ridker P.M., Neale B.M., Berger B. Efficient Bayesian mixed-model analysis increases association power in large cohorts. Nat. Genet. 2015;47:284–290. doi: 10.1038/ng.3190. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 35.Liu J.Z., McRae A.F., Nyholt D.R., Medland S.E., Wray N.R., Brown K.M., Hayward N.K., Montgomery G.W., Visscher P.M., Martin N.G., Macgregor S., AMFS Investigators A versatile gene-based test for genome-wide association studies. Am. J. Hum. Genet. 2010;87:139–145. doi: 10.1016/j.ajhg.2010.06.009. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 36.Li M.X., Gui H.S., Kwan J.S., Sham P.C. GATES: a rapid and powerful gene-based association test using extended Simes procedure. Am. J. Hum. Genet. 2011;88:283–293. doi: 10.1016/j.ajhg.2011.01.019. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 37.Yang J., Ferreira T., Morris A.P., Medland S.E., Genetic Investigation of ANthropometric Traits (GIANT) Consortium. DIAbetes Genetics Replication And Meta-analysis (DIAGRAM) Consortium. Madden P.A., Heath A.C., Martin N.G., Montgomery G.W. Conditional and joint multiple-SNP analysis of GWAS summary statistics identifies additional variants influencing complex traits. Nat. Genet. 2012;44:369–375. doi: 10.1038/ng.2213. S1–S3. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 38. Finucane HK, Bulik-Sullivan B., Gusev A., Trynka G., Reshef Y., Loh PR, Anttila V., Xu H., Zang C., Farh K. Particionando herdabilidade por anotação funcional usando todo o genoma estatísticas resumidas da associação. Nat. Geneta. 2015;47:1228–1235. doi: 10.1038/ng.3404. [ DOI ] [ artigo gratuito do PMC ] [ PubMed ] [ Google Scholar ]
- 39. Yang J., Benyamin B., McEvoy BP, Gordon S., Henders AK, Nyholt DR, Madden PA, Heath AC, Martin NG, Montgomery GW SNPs comuns explicam uma grande proporção da herdabilidade para a altura humana. Nat. Geneta. 2010;42:565–569. doi: 10.1038/ng.608. [ DOI ] [ artigo gratuito do PMC ] [ PubMed ] [ Google Scholar ]
- 40. Bowden J., Davey Smith G., Burgess S. Randomização mendeliana com instrumentos inválidos: estimativa de efeito e detecção de viés por meio de regressão de Egger. Internacional J. Epidemiol. 2015;44:512–525. doi: 10.1093/ije/dyv080. [ DOI ] [ artigo gratuito do PMC ] [ PubMed ] [ Google Scholar ]
- 41. Burgess S., Butterworth A., Thompson SG Análise de randomização mendeliana com múltiplas variantes genéticas usando dados resumidos. Geneta. Epidemiol. 2013;37:658–665. doi: 10.1002/gepi.21758. [ DOI ] [ artigo gratuito do PMC ] [ PubMed ] [ Google Scholar ]
- 42. Smith GD, Ebrahim S. 'randomização mendeliana': a epidemiologia genética pode contribuir para a compreensão dos determinantes ambientais da doença? Internacional J. Epidemiol. 2003;32:1–22. doi: 10.1093/ije/dyg070. [ DOI ] [ PubMed ] [ Google Scholar ]
- 43.Jia X., Han B., Onengut-Gumuscu S., Chen W.M., Concannon P.J., Rich S.S., Raychaudhuri S., de Bakker P.I. Imputing amino acid polymorphisms in human leukocyte antigens. PLoS ONE. 2013;8:e64683. doi: 10.1371/journal.pone.0064683. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 44.Howie B., Fuchsberger C., Stephens M., Marchini J., Abecasis G.R. Fast and accurate genotype imputation in genome-wide association studies through pre-phasing. Nat. Genet. 2012;44:955–959. doi: 10.1038/ng.2354. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 45.Huang J., Howie B., McCarthy S., Memari Y., Walter K., Min J.L., Danecek P., Malerba G., Trabetti E., Zheng H.F. Improved imputation of low-frequency and rare variants using the UK10K haplotype reference panel. Nat. Commun. 2015;6:8111. doi: 10.1038/ncomms9111. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 46. Browning BL, Imputação de genótipos Browning SR com milhões de amostras de referência. Sou. J. Hum. Geneta. 2016;98:116–126. doi: 10.1016/j.ajhg.2015.11.020. [ DOI ] [ artigo gratuito do PMC ] [ PubMed ] [ Google Scholar ]
- 47. Visscher PM, Hill WG, Wray NR Herdabilidade na era da genômica - conceitos e equívocos. Nat. Rev. 2008;9:255–266. doi: 10.1038/nrg2322. [ DOI ] [ PubMed ] [ Google Scholar ]
- 48. Yang J., Lee T., Kim J., Cho MC, Han BG, Lee JY, Lee HJ, Cho S., Kim H. Poligenicidade ubíqua de características complexas humanas: análise genômica ampla de 49 características em coreanos. PLoS Genet. 2013;9:e1003355. doi: 10.1371/journal.pgen.1003355. [ DOI ] [ artigo gratuito do PMC ] [ PubMed ] [ Google Scholar ]
- 49. Yang J., Bakshi A., Zhu Z., Hemani G., Vinkhuyzen AA, Lee SH, Robinson MR, Perry JR, Nolte IM, van Vliet-Ostaptchouk JV A estimativa de variância genética com variantes imputadas encontra herdabilidade ausente insignificante para humanos altura e índice de massa corporal. Nat. Geneta. 2015;47:1114–1120. doi: 10.1038/ng.3390. [ DOI ] [ artigo gratuito do PMC ] [ PubMed ] [ Google Scholar ]
- 50. Moser G., Lee SH, Hayes BJ, Goddard ME, Wray NR, Visscher PM Descoberta simultânea, estimativa e análise de previsão de características complexas usando um modelo de mistura bayesiana. PLoS Genet. 2015;11:e1004969. doi: 10.1371/journal.pgen.1004969. [ DOI ] [ artigo gratuito do PMC ] [ PubMed ] [ Google Scholar ]
- 51.van Rheenen W., Shatunov A., Dekker A.M., McLaughlin R.L., Diekstra F.P., Pulit S.L., van der Spek R.A., Võsa U., de Jong S., Robinson M.R. Genome-wide association analyses identify new risk variants and the genetic architecture of amyotrophic lateral sclerosis. Nat. Genet. 2016;48:1043–1048. doi: 10.1038/ng.3622. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 52.Ripke S., O’Dushlaine C., Chambert K., Moran J.L., Kähler A.K., Akterin S., Bergen S.E., Collins A.L., Crowley J.J., Fromer M. Genome-wide association analysis identifies 13 new risk loci for schizophrenia. Nat. Genet. 2013;45:1150–1159. doi: 10.1038/ng.2742. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 53.Loh P.R., Bhatia G., Gusev A., Finucane H.K., Bulik-Sullivan B.K., Pollack S.J., Schizophrenia Working Group of Psychiatric Genomics Consortium. de Candia T.R., Lee S.H., Wray N.R. Contrasting genetic architectures of schizophrenia and other complex diseases using fast variance-components analysis. Nat. Genet. 2015;47:1385–1392. doi: 10.1038/ng.3431. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 54.Cortes A., Pulit S.L., Leo P.J., Pointon J.J., Robinson P.C., Weisman M.H., Ward M., Gensler L.S., Zhou X., Garchon H.J. Major histocompatibility complex associations of ankylosing spondylitis are complex and involve further epistasis with ERAP1. Nat. Commun. 2015;6:7146. doi: 10.1038/ncomms8146. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 55. Evans DM, Visscher PM, Wray NR Aproveitar as informações contidas nos estudos de associação genômica ampla para melhorar a previsão individual do risco de doenças complexas. Zumbir. Mol. Geneta. 2009;18:3525–3531. doi: 10.1093/hmg/ddp295. [ DOI ] [ PubMed ] [ Google Scholar ]
- 56. Gamazon ER, Wheeler HE, Shah KP, Mozaffari SV, Aquino-Michaels K., Carroll RJ, Eyler AE, Denny JC, Consórcio GTEx. Nicolae DL Um método de associação baseado em genes para mapear características usando dados de transcriptoma de referência. Nat. Geneta. 2015;47:1091–1098. doi: 10.1038/ng.3367. [ DOI ] [ artigo gratuito do PMC ] [ PubMed ] [ Google Scholar ]
- 57. Evans DM, Brion MJ, Paternoster L., Kemp JP, McMahon G., Munafò M., Whitfield JB, Medland SE, Montgomery GW, GIANT Consortium Mineração do fenômeno humano usando pontuações alélicas que indexam intermediários biológicos. PLoS Genet. 2013;9:e1003919. doi: 10.1371/journal.pgen.1003919. [ DOI ] [ artigo gratuito do PMC ] [ PubMed ] [ Google Scholar ]
- 58. Tyrrell J., Wood AR, Ames RM, Yaghootkar H., Beaumont RN, Jones SE, Tuke MA, Ruth KS, Freathy RM, Davey Smith G. Interações ambientais gene-obesogênicas no estudo UK Biobank. Internacional J. Epidemiol. 2017;46:559–575. doi: 10.1093/ije/dyw337. [ DOI ] [ artigo gratuito do PMC ] [ PubMed ] [ Google Scholar ]
- 59. Zheng J., Erzurumluoglu AM, Elsworth BL, Kemp JP, Howe L., Haycock PC, Hemani G., Tansey K., Laurin C., Early Genetics and Lifecourse Epidemiology (EAGLE) Eczema Consortium LD Hub: um banco de dados centralizado e interface web para realizar regressão de pontuação LD que maximiza o potencial dos dados GWAS de nível resumido para herdabilidade de SNP e análise de correlação genética. Bioinformática. 2017;33:272–279. doi: 10.1093/bioinformática/btw613. [ DOI ] [ artigo gratuito do PMC ] [ PubMed ] [ Google Scholar ]
- 60. Hoffmann TJ, Ehret GB, Nandakumar P., Ranatunga D., Schaefer C., Kwok PY, Iribarren C., Chakravarti A., Risch N. Análises de associação de todo o genoma usando registros eletrônicos de saúde identificam novos loci que influenciam a variação da pressão arterial . Nat. Geneta. 2017;49:54–64. doi: 10.1038/ng.3715. [ DOI ] [ artigo gratuito do PMC ] [ PubMed ] [ Google Scholar ]
- [Artigo gratuito do PMC] [PubMed] 61. Gusev A, Ko A, Shi H, Bhatia G, Chung W, Penninx BW, Jansen R, de Geus EJ, Boomsma DI, Wright FA Abordagens integrativas para associação em grande escala do transcriptoma estudos. Nat. Geneta. 2016;48:245–252. doi: 10.1038/ng.3506. [ DOI ] [ artigo gratuito do PMC ] [ PubMed ] [ Google Scholar
- [Artigo gratuito do PMC] [PubMed ] 62. Zhu Z, Zhang F, Hu H, Bakshi A, Robinson MR, Powell JE, Montgomery GW, Goddard ME, Wray NR, Visscher PM, Yang J. Integração de dados resumidos de GWAS e Os estudos eQTL prevêem alvos genéticos de características complexas. Nat. Geneta. 2016;48:481–487. doi: 10.1038/ng.3538. [ DOI ] [ PubMed ] [ Google Scholar ]
- 63. Bulik-Sullivan BK, Loh PR, Finucane HK, Ripke S., Yang J., Grupo de Trabalho de Esquizofrenia do Consórcio de Genômica Psiquiátrica. Patterson N., Daly MJ, Price AL, Neale BM A regressão do LD Score distingue confusão de poligenicidade em estudos de associação genômica ampla. Nat. Geneta. 2015;47:291–295. doi: 10.1038/ng.3211. [ DOI ] [ artigo gratuito do PMC ] [ PubMed ] [ Google Scholar ]
- 64. Claussnitzer M., Dankel SN, Kim KH, Quon G., Meuleman W., Haugen C., Glunk V., Sousa IS, Beaudry JL, Puviindran V. Circuito variante de obesidade FTO e escurecimento de adipócitos em humanos. N. Engl. J. Med. 2015;373:895–907. doi: 10.1056/NEJMoa1502214. [ DOI ] [ artigo gratuito do PMC ] [ PubMed ] [ Google Scholar ]
- 65. Sekar A., Bialas AR, de Rivera H., Davis A., Hammond TR, Kamitaki N., Tooley K., Presumey J., Baum M., Van Doren V. Risco de esquizofrenia devido à variação complexa do componente 4 do complemento . Natureza. 2016;530:177–183. doi: 10.1038/nature16549. [ DOI ] [ artigo gratuito do PMC ] [ PubMed ] [ Google Scholar ]
- 66.Albert F.W., Kruglyak L. The role of regulatory variation in complex traits and disease. Nat. Rev. Genet. 2015;16:197–212. doi: 10.1038/nrg3891. [DOI] [PubMed] [Google Scholar]
- 67.Pasquali L., Gaulton K.J., Rodríguez-Seguí S.A., Mularoni L., Miguel-Escalada I., Akerman I., Tena J.J., Morán I., Gómez-Marín C., van de Bunt M. Pancreatic islet enhancer clusters enriched in type 2 diabetes risk-associated variants. Nat. Genet. 2014;46:136–143. doi: 10.1038/ng.2870. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 68.ENCODE Project Consortium An integrated encyclopedia of DNA elements in the human genome. Nature. 2012;489:57–74. doi: 10.1038/nature11247. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 69.Roadmap Epigenomics Consortium. Kundaje A., Meuleman W., Ernst J., Bilenky M., Yen A., Heravi-Moussavi A., Kheradpour P., Zhang Z., Wang J. Integrative analysis of 111 reference human epigenomes. Nature. 2015;518:317–330. doi: 10.1038/nature14248. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 70. Consórcio GTEx O projeto Genotype-Tissue Expression (GTEx). Nat. Geneta. 2013;45:580–585. doi: 10.1038/ng.2653. [ DOI ] [ artigo gratuito do PMC ] [ PubMed ] [ Google Scholar ]
- 71. Won H., de la Torre-Ubieta L., Stein JL, Parikshak NN, Huang J., Opland CK, Gandal MJ, Sutton GJ, Hormozdiari F., Lu D. A conformação cromossômica elucida as relações regulatórias no desenvolvimento do cérebro humano. Natureza. 2016;538:523–527. doi: 10.1038/nature19847. [ DOI ] [ artigo gratuito do PMC ] [ PubMed ] [ Google Scholar ]
- 72. Nelson MR, Tipney H., Painter JL, Shen J., Nicoletti P., Shen Y., Floratos A., Sham PC, Li MJ, Wang J. O suporte de evidências genéticas humanas para indicações de medicamentos aprovados. Nat. Geneta. 2015;47:856–860. doi: 10.1038/ng.3314. [ DOI ] [ PubMed ] [ Google Scholar ]
- 73. McDonald TJ, Ellard S. Diabetes com início na maturidade dos jovens: identificação e diagnóstico. Ana. Clin. Bioquímica. 2013;50:403–415. doi: 10.1177/0004563213483458. [ DOI ] [ PubMed ] [ Google Scholar ]
- 74. Fuchsberger C., Flannick J., Teslovich TM, Mahajan A., Agarwala V., Gaulton KJ, Ma C., Fontanillas P., Moutsianas L., McCarthy DJ A arquitetura genética do diabetes tipo 2. Natureza. 2016;536:41–47. doi: 10.1038/nature18642. [ DOI ] [ artigo gratuito do PMC ] [ PubMed ] [ Google Scholar ]
- 75. Consórcio de replicação e meta-análise genética DIAbetes (DIAGRAM). Consórcio da Rede Asiática de Epidemiologia Genética para Diabetes Tipo 2 (AGEN-T2D). Consórcio de Diabetes Tipo 2 do Sul da Ásia (SAT2D). Consórcio Mexicano-Americano de Diabetes Tipo 2 (MAT2D). Exploração genética do diabetes tipo 2 por sequenciamento de geração Nex em consórcio de amostras muylti-étnicas (T2D-GENES). Mahajan A., Go MJ, Zhang W., Below JE, Gaulton KJ A meta-análise trans-ancestral de todo o genoma fornece informações sobre a arquitetura genética da suscetibilidade ao diabetes tipo 2. Nat. Geneta. 2014;46:234–244. doi: 10.1038/ng.2897. [ DOI ] [ artigo gratuito do PMC ] [ PubMed ] [ Google Scholar ]
- 76.Morris A.P., Voight B.F., Teslovich T.M., Ferreira T., Segrè A.V., Steinthorsdottir V., Strawbridge R.J., Khan H., Grallert H., Mahajan A. Large-scale association analysis provides insights into the genetic architecture and pathophysiology of type 2 diabetes. Nat. Genet. 2012;44:981–990. doi: 10.1038/ng.2383. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 77.Steinthorsdottir V., Thorleifsson G., Sulem P., Helgason H., Grarup N., Sigurdsson A., Helgadottir H.T., Johannsdottir H., Magnusson O.T., Gudjonsson S.A. Identification of low-frequency and rare sequence variants associated with elevated or reduced risk of type 2 diabetes. Nat. Genet. 2014;46:294–298. doi: 10.1038/ng.2882. [DOI] [PubMed] [Google Scholar]
- 78.Ayub Q., Moutsianas L., Chen Y., Panoutsopoulou K., Colonna V., Pagani L., Prokopenko I., Ritchie G.R., Tyler-Smith C., McCarthy M.I. Revisiting the thrifty gene hypothesis via 65 loci associated with susceptibility to type 2 diabetes. Am. J. Hum. Genet. 2014;94:176–185. doi: 10.1016/j.ajhg.2013.12.010. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 79. Field Y., Boyle EA, Telis N., Gao Z., Gaulton KJ, Golan D., Yengo L., Rocheleau G., Froguel P., McCarthy MI, Pritchard JK Detecção de adaptação humana durante os últimos 2.000 anos . Ciência. 2016;354:760–764. doi: 10.1126/science.aag0776. [ DOI ] [ artigo gratuito do PMC ] [ PubMed ] [ Google Scholar ]
- 80. Waters KM, Stram DO, Hassanein MT, Le Marchand L., Wilkens LR, Maskarinec G., Monroe KR, Kolonel LN, Altshuler D., Henderson BE, Haiman CA Associação consistente de variantes de risco de diabetes tipo 2 encontradas em europeus em diversos grupos raciais e étnicos. PLoS Genet. 2010;6:e1001078. doi: 10.1371/journal.pgen.1001078. [ DOI ] [ artigo gratuito do PMC ] [ PubMed ] [ Google Scholar ]
- 81. Moltke I., Grarup N., Jørgensen ME, Bjerregaard P., Treebak JT, Fumagalli M., Korneliussen TS, Andersen MA, Nielsen TS, Krarup NT Uma variante TBC1D4 groenlandesa comum confere resistência à insulina muscular e diabetes tipo 2. Natureza. 2014;512:190–193. doi: 10.1038/nature13425. [ DOI ] [ PubMed ] [ Google Scholar ]
- 82. Claussnitzer M., Dankel SN, Klocke B., Grallert H., Glunk V., Berulava T., Lee H., Oskolkov N., Fadista J., Ehlers K. Aproveitando padrões de sítios de ligação de fatores de transcrição de espécies cruzadas: desde locais de risco de diabetes até mecanismos de doença. Célula. 2014;156:343–358. doi: 10.1016/j.cell.2013.10.058. [ DOI ] [ artigo gratuito do PMC ] [ PubMed ] [ Google Scholar ]
- 83. Scott LJ, Erdos MR, Huyghe JR, Welch RP, Beck AT, Wolford BN, Chines PS, Didion JP, Narisu N., Stringham HM A assinatura reguladora genética do diabetes tipo 2 no músculo esquelético humano. Nat. Comum. 2016;7:11764. doi: 10.1038/ncoms11764. [ DOI ] [ artigo gratuito do PMC ] [ PubMed ] [ Google Scholar ]
- 84.Parker S.C., Stitzel M.L., Taylor D.L., Orozco J.M., Erdos M.R., Akiyama J.A., van Bueren K.L., Chines P.S., Narisu N., NISC Comparative Sequencing Program Chromatin stretch enhancer states drive cell-specific gene regulation and harbor human disease risk variants. Proc. Natl. Acad. Sci. USA. 2013;110:17921–17926. doi: 10.1073/pnas.1317023110. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 85.Small K.S., Hedman A.K., Grundberg E., Nica A.C., Thorleifsson G., Kong A., Thorsteindottir U., Shin S.Y., Richards H.B., GIANT Consortium Identification of an imprinted master trans regulator at the KLF14 locus related to multiple metabolic phenotypes. Nat. Genet. 2011;43:561–564. doi: 10.1038/ng.833. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 86.Gaulton K.J., Ferreira T., Lee Y., Raimondo A., Mägi R., Reschen M.E., Mahajan A., Locke A., Rayner N.W., Robertson N. Genetic fine mapping and genomic annotation defines causal mechanisms at type 2 diabetes susceptibility loci. Nat. Genet. 2015;47:1415–1425. doi: 10.1038/ng.3437. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 87. Dimas AS, Lagou V., Barker A., Knowles JW, Mägi R., Hivert MF, Benazzo A., Rybin D., Jackson AU, Stringham HM O impacto das variantes de suscetibilidade ao diabetes tipo 2 nas características glicêmicas quantitativas revela heterogeneidade mecanística . Diabetes. 2014;63:2158–2171. doi: 10.2337/db13-0949. [ DOI ] [ artigo gratuito do PMC ] [ PubMed ] [ Google Scholar ]
- 88. van de Bunt M., Manning Fox JE, Dai X., Barrett A., Gray C., Li L., Bennett AJ, Johnson PR, Rajotte RV, Gaulton KJ Transcrição Expressão Dados de Ilhotas Humanas Links Sinais Regulatórios do Genoma - Amplos estudos de associação para diabetes tipo 2 e características glicêmicas com seus efetores posteriores. PLoS Genet. 2015;11:e1005694. doi: 10.1371/journal.pgen.1005694. [ DOI ] [ artigo gratuito do PMC ] [ PubMed ] [ Google Scholar ]
- 89. Saxena R., Hivert MF, Langenberg C., Tanaka T., Pankow JS, Vollenweider P., Lyssenko V., Bouatia-Naji N., Dupuis J., Jackson AU A variação genética no GIPR influencia as respostas de glicose e insulina a um desafio oral de glicose. Nat. Geneta. 2010;42:142–148. doi: 10.1038/ng.521. [ DOI ] [ artigo gratuito do PMC ] [ PubMed ] [ Google Scholar ]
- 90. Kooner JS, Saleheen D., Sim X., Sehmi J., Zhang W., Frossard P., Been LF, Chia KS, Dimas AS, Hassanali N. Estudo de associação genômica ampla em indivíduos de ascendência do sul da Ásia identifica seis novos loci de suscetibilidade ao diabetes tipo 2. Nat. Geneta. 2011;43:984–989. doi: 10.1038/ng.921. [ DOI ] [ artigo gratuito do PMC ] [ PubMed ] [ Google Scholar ]
- 91.Sandhu M.S., Weedon M.N., Fawcett K.A., Wasson J., Debenham S.L., Daly A., Lango H., Frayling T.M., Neumann R.J., Sherva R. Common variants in WFS1 confer risk of type 2 diabetes. Nat. Genet. 2007;39:951–953. doi: 10.1038/ng2067. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 92.Wei F.Y., Tomizawa K. Functional loss of Cdkal1, a novel tRNA modification enzyme, causes the development of type 2 diabetes. Endocr. J. 2011;58:819–825. doi: 10.1507/endocrj.ej11-0099. [DOI] [PubMed] [Google Scholar]
- 93.Flannick J., Thorleifsson G., Beer N.L., Jacobs S.B., Grarup N., Burtt N.P., Mahajan A., Fuchsberger C., Atzmon G., Benediktsson R. Loss-of-function mutations in SLC30A8 protect against type 2 diabetes. Nat. Genet. 2014;46:357–363. doi: 10.1038/ng.2915. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 94.Fall T., Xie W., Poon W., Yaghootkar H., Mägi R., GENESIS Consortium. Knowles J.W., Lyssenko V., Weedon M., Frayling T.M., Ingelsson E. Using Genetic Variants to Assess the Relationship Between Circulating Lipids and Type 2 Diabetes. Diabetes. 2015;64:2676–2684. doi: 10.2337/db14-1710. [DOI] [PubMed] [Google Scholar]
- 95.Horikoshi M., Beaumont R.N., Day F.R., Warrington N.M., Kooijman M.N., Fernandez-Tajes J., Feenstra B., van Zuydam N.R., Gaulton K.J., Grarup N. Genome-wide associations for birth weight and correlations with adult disease. Nature. 2016;538:248–252. doi: 10.1038/nature19806. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 96.Lotta L.A., Sharp S.J., Burgess S., Perry J.R., Stewart I.D., Willems S.M., Luan J., Ardanaz E., Arriola L., Balkau B. Association Between Low-Density Lipoprotein Cholesterol-Lowering Genetic Variants and Risk of Type 2 Diabetes: A Meta-analysis. JAMA. 2016;316:1383–1391. doi: 10.1001/jama.2016.14568. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 97. Rafiq S., Melzer D., Weedon MN, Lango H., Saxena R., Scott LJ, DIAGRAM Consortium. Palmer CN, Morris AD, McCarthy Variantes genéticas que influenciam medidas de inflamação ou predispõem a doenças autoimunes e inflamatórias não estão associadas ao risco de diabetes tipo 2. Diabetologia. 2008;51:2205–2213. doi: 10.1007/s00125-008-1160-3. [ DOI ] [ artigo gratuito do PMC ] [ PubMed ] [ Google Scholar ]
- 98. Ye Z., Sharp SJ, Burgess S., Scott RA, Imamura F., InterAct Consortium. Langenberg C., Wareham NJ, Forouhi NG Associação entre 25-hidroxivitamina D circulante e diabetes tipo 2 incidente: um estudo de randomização mendeliano. Lancet Diabetes Endocrinol. 2015;3:35–42. doi: 10.1016/S2213-8587(14)70184-6. [ DOI ] [ artigo gratuito do PMC ] [ PubMed ] [ Google Scholar ]
- 99. Lotta LA, Gulati P., Day FR, Payne F., Ongen H., van de Bunt M., Gaulton KJ, Eicher JD, Sharp SJ, Luan J. A análise genômica integrativa implica capacidade limitada de armazenamento adiposo periférico na patogênese da resistência humana à insulina. Nat. Geneta. 2017;49:17–26. doi: 10.1038/ng.3714. [ DOI ] [ artigo gratuito do PMC ] [ PubMed ] [ Google Scholar ]
- 100. Grupo de Estudo de Farmacogenética de Diabetes GoDARTS e UKPDS. Wellcome Trust Case Control Consortium 2. Zhou K., Bellenguez C., Spencer CC, Bennett AJ, Coleman RL, Tavendale R., Hawley SA, Donnelly LA Variantes comuns próximas à ATM estão associadas à resposta glicêmica à metformina no diabetes tipo 2. Nat. Geneta. 2011;43:117–120. doi: 10.1038/ng.735. [ DOI ] [ artigo gratuito do PMC ] [ PubMed ] [ Google Scholar ]
- 101.Zhou K., Yee S.W., Seiser E.L., van Leeuwen N., Tavendale R., Bennett A.J., Groves C.J., Coleman R.L., van der Heijden A.A., Beulens J.W. Variation in the glucose transporter gene SLC2A2 is associated with glycemic response to metformin. Nat. Genet. 2016;48:1055–1059. doi: 10.1038/ng.3632. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 102.Gloyn A.L., Pearson E.R., Antcliff J.F., Proks P., Bruining G.J., Slingerland A.S., Howard N., Srinivasan S., Silva J.M., Molnes J. Activating mutations in the gene encoding the ATP-sensitive potassium-channel subunit Kir6.2 and permanent neonatal diabetes. N. Engl. J. Med. 2004;350:1838–1849. doi: 10.1056/NEJMoa032922. [DOI] [PubMed] [Google Scholar]
- 103. Lynch CJ, Adams SH Aminoácidos de cadeia ramificada na sinalização metabólica e resistência à insulina. Nat. Rev. Endocrinol. 2014;10:723–736. doi: 10.1038/nrendo.2014.171. [ DOI ] [ artigo gratuito do PMC ] [ PubMed ] [ Google Scholar ]
- 104. Pedersen HK, Gudmundsdottir V., Nielsen HB, Hyotylainen T., Nielsen T., Jensen BA, Forslund K., Hildebrand F., Prifti E., Falony G. Micróbios intestinais humanos impactam o metaboloma sérico do hospedeiro e a sensibilidade à insulina. Natureza. 2016;535:376–381. doi: 10.1038/nature18646. [ DOI ] [ PubMed ] [ Google Scholar ]
- 105. Cortes A., Brown MA Promessa e armadilhas do Immunochip. Artrite Res. Lá. 2011;13:101. doi: 10.1186/ar3204. [ DOI ] [ artigo gratuito do PMC ] [ PubMed ] [ Google Scholar ]
- 106. Liu JZ, van Sommeren S., Huang H., Ng SC, Alberts R., Takahashi A., Ripke S., Lee JC, Jostins L., Shah T. As análises da associação identificam 38 loci de suscetibilidade para doença inflamatória intestinal e destacar o risco genético compartilhado entre as populações. Nat. Geneta. 2015;47:979–986. doi: 10.1038/ng.3359. [ DOI ] [ artigo gratuito do PMC ] [ PubMed ] [ Google Scholar ]
- 107. Jiang L., Yin J., Ye L., Yang J., Hemani G., Liu AJ, Zou H., He D., Sun L., Zeng X. Novos locais de risco para artrite reumatóide em chinês Han e congruência com variantes de risco em europeus. Artrite Reumatol. 2014;66:1121–1132. doi: 10.1002/art.38353. [ DOI ] [ PubMed ] [ Google Scholar ]
- 108. Okada Y., Wu D., Trynka G., Raj T., Terao C., Ikari K., Kochi Y., Ohmura K., Suzuki A., Yoshida S. A genética da artrite reumatóide contribui para a biologia e drogas descoberta. Natureza. 2014;506:376–381. doi: 10.1038/nature12873. [ DOI ] [ artigo gratuito do PMC ] [ PubMed ] [ Google Scholar ]
- 109.International Genetics of Ankylosing Spondylitis Consortium (IGAS) Cortes A., Hadler J., Pointon J.P., Robinson P.C., Karaderi T., Leo P., Cremin K., Pryce K., Harris J. Identification of multiple risk variants for ankylosing spondylitis through high-density genotyping of immune-related loci. Nat. Genet. 2013;45:730–738. doi: 10.1038/ng.2667. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 110.Gregory A.P., Dendrou C.A., Attfield K.E., Haghikia A., Xifara D.K., Butter F., Poschmann G., Kaur G., Lambert L., Leach O.A. TNF receptor 1 genetic risk mirrors outcome of anti-TNF therapy in multiple sclerosis. Nature. 2012;488:508–511. doi: 10.1038/nature11307. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 111.Gregersen P.K., Silver J., Winchester R.J. The shared epitope hypothesis. An approach to understanding the molecular genetics of susceptibility to rheumatoid arthritis. Arthritis Rheum. 1987;30:1205–1213. doi: 10.1002/art.1780301102. [DOI] [PubMed] [Google Scholar]
- 112.Raychaudhuri S., Sandor C., Stahl E.A., Freudenberg J., Lee H.S., Jia X., Alfredsson L., Padyukov L., Klareskog L., Worthington J. Five amino acids in three HLA proteins explain most of the association between MHC and seropositive rheumatoid arthritis. Nat. Genet. 2012;44:291–296. doi: 10.1038/ng.1076. [DOI] [PMC free article] [PubMed] [Google Scholar]
- [Artigo gratuito do PMC] [PubMed ] 113. Scally SW, Petersen J, Law SC, Dudek NL, Nel HJ, Loh KL, Wijeyewickrema LC, Eckle SB, van Heemst J, Pike RN Uma base molecular para a associação do HLA- Locus DRB1 com artrite reumatóide por citrulinação. J. Exp. Med. 2013;210:2569–2582. doi: 10.1084/jam.20131241. [ DOI ] [ artigo gratuito do PMC ] [ PubMed ] [ Google Scholar
- 114. Heap GA, Weedon MN, Bewshea CM, Singh A., Chen M., Satchwell JB, Vivian JP, So K., Dubois PC, Andrews JM As variantes HLA-DQA1-HLA-DRB1 conferem suscetibilidade à pancreatite induzida por imunossupressores de tiopurina . Nat. Geneta. 2014;46:1131–1134. doi: 10.1038/ng.3093. [ DOI ] [ artigo gratuito do PMC ] [ PubMed ] [ Google Scholar ]
- 115. Hueber W., Patel DD, Dryja T., Wright AM, Koroleva I., Bruin G., Antoni C., Draelos Z., Gold MH, Psoriasis Study Group Effects of AIN457, um anticorpo totalmente humano para interleucina-17A , na psoríase, artrite reumatóide e uveíte. Ciência. Trad. Med. 2010;2:52ra72. doi: 10.1126/scitranslmed.3001107. [ DOI ] [ PubMed ] [ Google Scholar ]
- 116. McInnes IB, Sieper J., Braun J., Emery P., van der Heijde D., Isaacs JD, Dahmen G., Wollenhaupt J., Schulze-Koops H., Kogan J. Eficácia e segurança do secukinumab, a anticorpo monoclonal anti-interleucina-17A totalmente humano, em pacientes com artrite psoriática moderada a grave: um ensaio de prova de conceito de fase II, randomizado, duplo-cego, controlado por placebo, de 24 semanas. Ana. Reum. Dis. 2014;73:349–356. doi: 10.1136/annrheumdis-2012-202646. [ DOI ] [ PubMed ] [ Google Scholar ]
- 117.Sieper J., Deodhar A., Marzo-Ortega H., Aelion J.A., Blanco R., Jui-Cheng T., Andersson M., Porter B., Richards H.B., MEASURE 2 Study Group Secukinumab efficacy in anti-TNF-naive and anti-TNF-experienced subjects with active ankylosing spondylitis: results from the MEASURE 2 Study. Ann. Rheum. Dis. 2017;76:571–592. doi: 10.1136/annrheumdis-2016-210023. [DOI] [PubMed] [Google Scholar]
- 118.Subramanian V., Knight J.S., Parelkar S., Anguish L., Coonrod S.A., Kaplan M.J., Thompson P.R. Design, synthesis, and biological evaluation of tetrazole analogs of Cl-amidine as protein arginine deiminase inhibitors. J. Med. Chem. 2015;58:1337–1344. doi: 10.1021/jm501636x. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 119.Kawalkowska J., Quirke A.M., Ghari F., Davis S., Subramanian V., Thompson P.R., Williams R.O., Fischer R., La Thangue N.B., Venables P.J. Abrogation of collagen-induced arthritis by a peptidyl arginine deiminase inhibitor is associated with modulation of T cell-mediated immune responses. Sci. Rep. 2016;6:26430. doi: 10.1038/srep26430. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 120. Agrawal N., Brown MA Associações genéticas e caracterização funcional de aminopeptidases M1 e doenças imunomediadas. Genes imunes. 2014;15:521–527. doi: 10.1038/gene.2014.46. [ DOI ] [ PubMed ] [ Google Scholar ]
- 121. Grupos de Trabalho CNV e Esquizofrenia do Consórcio de Genômica Psiquiátrica. Psychosis Endophenotypes International Consortium Contribuição de variantes do número de cópias para a esquizofrenia a partir de um estudo genômico amplo de 41.321 indivíduos. Nat Genet. 2017;49:27–35. doi: 10.1038/ng.3725. [ DOI ] [ artigo gratuito do PMC ] [ PubMed ] [ Google Scholar ]
- 122. Girard SL, Gauthier J., Noreau A., Xiong L., Zhou S., Jouan L., Dionne-Laporte A., Spiegelman D., Henrion E., Diallo O. Aumento da taxa de mutação exônica de novo em indivíduos com esquizofrenia. Nat. Geneta. 2011;43:860–863. doi: 10.1038/ng.886. [ DOI ] [ PubMed ] [ Google Scholar ]
- 123. Xu B., Roos JL, Dexheimer P., Boone B., Plummer B., Levy S., Gogos JA, Karayiorgou M. O sequenciamento Exome suporta um paradigma mutacional de novo para a esquizofrenia. Nat. Geneta. 2011;43:864–868. doi: 10.1038/ng.902. [ DOI ] [ artigo gratuito do PMC ] [ PubMed ] [ Google Scholar ]
- 124. Fromer M., Pocklington AJ, Kavanagh DH, Williams HJ, Dwyer S., Gormley P., Georgieva L., Rees E., Palta P., Ruderfer DM Mutações de novo na esquizofrenia implicam redes sinápticas. Natureza. 2014;506:179–184. doi: 10.1038/nature12929. [ DOI ] [ artigo gratuito do PMC ] [ PubMed ] [ Google Scholar ]
- 125. De Rubeis S., He X., Goldberg AP, Poultney CS, Samocha K., Cicek AE, Kou Y., Liu L., Fromer M., Walker S. Genes sinápticos, transcricionais e de cromatina interrompidos no autismo. Natureza. 2014;515:209–215. doi: 10.1038/nature13772. [ DOI ] [ artigo gratuito do PMC ] [ PubMed ] [ Google Scholar ]
- 126. McCarthy SE, Gillis J., Kramer M., Lihm J., Yoon S., Berstein Y., Mistry M., Pavlidis P., Solomon R., Ghiban E. Mutações de novo na esquizofrenia implicam remodelação e suporte da cromatina uma sobreposição genética com autismo e deficiência intelectual. Mol. Psiquiatria. 2014;19:652–658. doi: 10.1038/mp.2014.29. [ DOI ] [ artigo gratuito do PMC ] [ PubMed ] [ Google Scholar ]
- 127. Jiang YH, Yuen RK, Jin X., Wang M., Chen N., Wu X., Ju J., Mei J., Shi Y., He M. Detecção de variantes genéticas clinicamente relevantes no transtorno do espectro do autismo por sequenciamento do genoma completo. Sou. J. Hum. Geneta. 2013;93:249–263. doi: 10.1016/j.ajhg.2013.06.012. [ DOI ] [ artigo gratuito do PMC ] [ PubMed ] [ Google Scholar ]
- 128. Iossifov I., O'Roak BJ, Sanders SJ, Ronemus M., Krumm N., Levy D., Stessman HA, Witherspoon KT, Vives L., Patterson KE A contribuição de mutações de codificação de novo para o transtorno do espectro do autismo. Natureza. 2014;515:216–221. doi: 10.1038/nature13908. [ DOI ] [ artigo gratuito do PMC ] [ PubMed ] [ Google Scholar ]
- 129. Georgieva L., Rees E., Moran JL, Chambert KD, Milanova V., Craddock N., Purcell S., Sklar P., McCarroll S., Holmans P. CNVs de novo no transtorno afetivo bipolar e esquizofrenia. Zumbir. Mol. Geneta. 2014;23:6677–6683. doi: 10.1093/hmg/ddu379. [ DOI ] [ artigo gratuito do PMC ] [ PubMed ] [ Google Scholar ]
- 130. Roussos P., Mitchell AC, Voloudakis G., Fullard JF, Pothula VM, Tsang J., Stahl EA, Georgakopoulos A., Ruderfer DM, Charney A. Um papel para variação não codificada na esquizofrenia. 2014;9:1417–1429. doi: 10.1016/j.celrep.2014.10.015. [ DOI ] [ artigo gratuito do PMC ] [ PubMed ] [ Google Scholar ]
- 131. Gusev A., Lee SH, Trynka G., Finucane H., Vilhjálmsson BJ, Xu H., Zang C., Ripke S., Bulik-Sullivan B., Stahl E. Particionando herdabilidade de regulação e tipo de célula- variantes específicas em 11 doenças comuns. Sou. J. Hum. Geneta. 2014;95:535–552. doi: 10.1016/j.ajhg.2014.10.004. [ DOI ] [ artigo gratuito do PMC ] [ PubMed ] [ Google Scholar ]
- 132. Subgrupo de análise de redes e vias do Consórcio de Genômica Psiquiátrica As análises de estudos de associação do genoma psiquiátrico implicam vias neuronais, imunológicas e histonas. Nat. Neurosci. 2015;18:199–209. doi: 10.1038/nn.3922. [ DOI ] [ artigo gratuito do PMC ] [ PubMed ] [ Google Scholar ]
- 133.de Jong S., Vidler L.R., Mokrab Y., Collier D.A., Breen G. Gene-set analysis based on the pharmacological profiles of drugs to identify repurposing opportunities in schizophrenia. J. Psychopharmacol. (Oxford) 2016;30:826–830. doi: 10.1177/0269881116653109. [DOI] [PubMed] [Google Scholar]
- 134.Bell J.T., Pai A.A., Pickrell J.K., Gaffney D.J., Pique-Regi R., Degner J.F., Gilad Y., Pritchard J.K. DNA methylation patterns associate with genetic and gene expression variation in HapMap cell lines. Genome Biol. 2011;12:R10. doi: 10.1186/gb-2011-12-1-r10. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 135.Kettunen J., Tukiainen T., Sarin A.P., Ortega-Alonso A., Tikkanen E., Lyytikäinen L.P., Kangas A.J., Soininen P., Würtz P., Silander K. Genome-wide association study identifies multiple loci influencing human serum metabolite levels. Nat. Genet. 2012;44:269–276. doi: 10.1038/ng.1073. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 136.Shah S., McRae A.F., Marioni R.E., Harris S.E., Gibson J., Henders A.K., Redmond P., Cox S.R., Pattie A., Corley J. Genetic and environmental exposures constrain epigenetic drift over the human life course. Genome Res. 2014;24:1725–1733. doi: 10.1101/gr.176933.114. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 137. Shah S., Bonder MJ, Marioni RE, Zhu Z., McRae AF, Zhernakova A., Harris SE, Liewald D., Henders AK, Mendelson MM Melhorando a previsão fenotípica combinando associações genéticas e epigenéticas. Sou. J. Hum. Geneta. 2015;97:75–85. doi: 10.1016/j.ajhg.2015.05.014. [ DOI ] [ artigo gratuito do PMC ] [ PubMed ] [ Google Scholar ]
- 138. Marouli E., Graff M., Medina-Gomez C., Lo KS, Wood AR, Kjaer TR, Fine RS, Lu Y., Schurmann C., Highland HM Variantes de codificação raras e de baixa frequência alteram a altura humana adulta. Natureza. 2017;542:186–190. doi: 10.1038/nature21039. [ DOI ] [ artigo gratuito do PMC ] [ PubMed ] [ Google Scholar ]
- 139. Farh KK, Marson A., Zhu J., Kleinewietfeld M., Housley WJ, Beik S., Shoresh N., Whitton H., Ryan RJ, Shishkin AA Mapeamento fino genético e epigenético de variantes causais de doenças autoimunes. Natureza. 2015;518:337–343. doi: 10.1038/nature13835. [ DOI ] [ artigo gratuito do PMC ] [ PubMed ] [ Google Scholar ]
- 140. Espanha SL, Barrett JC Estratégias para mapeamento preciso de características complexas. Zumbir. Mol. Geneta. 2015;24(R1):R111–R119. doi: 10.1093/hmg/ddv260. [ DOI ] [ artigo gratuito do PMC ] [ PubMed ] [ Google Scholar ]
- 141. He X., Fuller CK, Song Y., Meng Q., Zhang B., Yang X., Li H. Sherlock: detectando associações gene-doença combinando padrões de expressão QTL e GWAS. Sou. J. Hum. Geneta. 2013;92:667–680. doi: 10.1016/j.ajhg.2013.03.022. [ DOI ] [ artigo gratuito do PMC ] [ PubMed ] [ Google Scholar ]
- 142. Hormozdiari F., Kostem E., Kang EY, Pasaniuc B., Eskin E. Identificando variantes causais em loci com múltiplos sinais de associação. Genética. 2014;198:497–508. doi: 10.1534/genética.114.167908. [ DOI ] [ artigo gratuito do PMC ] [ PubMed ] [ Google Scholar ]
- 143. Morris AP Meta-análise transétnica de estudos de associação genômica ampla. Geneta. Epidemiol. 2011;35:809–822. doi: 10.1002/gepi.20630. [ DOI ] [ artigo gratuito do PMC ] [ PubMed ] [ Google Scholar ]
- 144. Hunter DJ, Kraft P. Beber da mangueira de incêndio - questões estatísticas em estudos de associação genômica. N. Engl. J. Med. 2007;357:436–439. doi: 10.1056/NEJMp078120. [ DOI ] [ PubMed ] [ Google Scholar ]
- 145. Risch N., Merikangas K. O futuro dos estudos genéticos de doenças humanas complexas. Ciência. 1996;273:1516–1517. doi: 10.1126/ciência.273.5281.1516. [ DOI ] [ PubMed ] [ Google Scholar ]
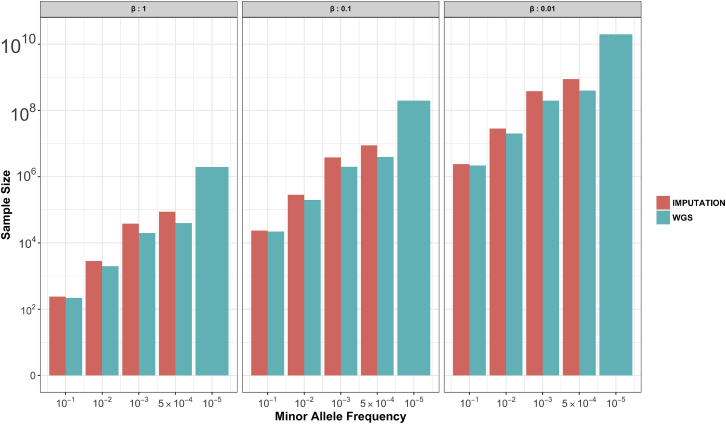
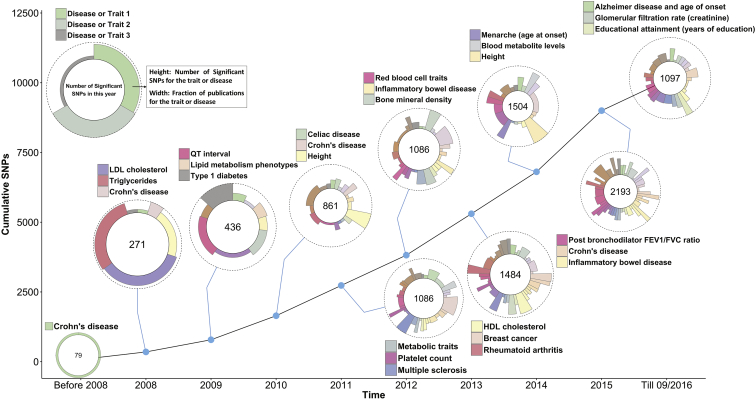
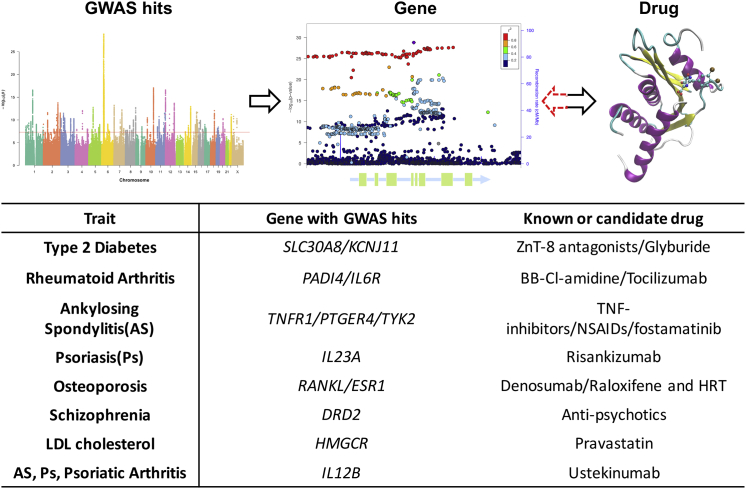
Nenhum comentário:
Postar um comentário
Observação: somente um membro deste blog pode postar um comentário.